7.1: Estructura del ADN
- Page ID
- 53034
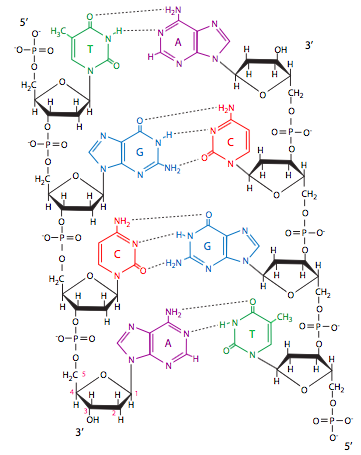
Como se puede apreciar en la Figura 1, los nucleótidos sólo varían ligeramente, y sólo en la base nitrogenada. En el caso del ADN, esas bases son adenina, guanina, citosina y timina. Obsérvese la similitud de las formas de adenina y guanina, y también la similitud entre citosina y timina. A y G se clasifican como purinas, mientras que C y T se clasifican como pirimidinas. Mientras estemos nombrando cosas, observe “desoxirribosa” y “ribosa”. Como su nombre lo indica, la desoxirribosa es solo una ribosa sin oxígeno. Más específicamente, donde hay un grupo hidroxilo unido al 2-carbono de la ribosa, solo hay un hidrógeno unido al 2-carbono de la desoxirribosa. Esa es la única diferencia entre los dos azúcares.
Al construir aleatoriamente una sola cadena de ácido nucleico in vitro, no existen reglas particulares en cuanto al orden de los nucleótidos con respecto a sus bases. Las identidades de sus bases nitrogenadas son irrelevantes porque los nucleótidos están unidos por enlaces fosfodiéster a través del grupo fosfato y la pentosa. Por lo tanto, a menudo se le conoce como la cadena principal de azúcar-fosfato. Si desglosamos la palabra “fosfodiéster”, vemos que describe con bastante facilidad la conexión: los azúcares están conectados por dos enlaces éster (—O—) con un fósforo en el medio. Una de las ideas que muchas veces confunde a los estudiantes es la direccionalidad de este vínculo, y por tanto, de los ácidos nucleicos en general. Por ejemplo, cuando hablamos de la ADN polimerasa, la enzima que cataliza la adición de nucleótidos en las células vivas, decimos que funciona en una dirección 5-prime (5') a 3-prime (3'). Esto puede parecer arcano molecular-biólogo-hablar, pero en realidad es muy sencillo. Eche otro vistazo a dos de los nucleótidos unidos por el enlace fosfodiéster (Figura\(\PageIndex{1}\), abajo a la izquierda). Un nucleótido de adenina se une a un nucleótido de citosina. El enlace fosfodiéster siempre enlazará el carbono 5 de una desoxirribosa (o ribosa en el ARN) al carbono 3 del siguiente azúcar. Esto también significa que en un extremo de una cadena de nucleótidos enlazados, habrá un grupo 5' fosfato libre (-PO 4), y en el otro extremo, un hidroxilo 3' libre (-OH). Estos definen la direccionalidad de una cadena de ADN o ARN.
El ADN se encuentra normalmente como una molécula bicatenaria en la célula mientras que el ARN es en su mayoría monocatenario. Sin embargo, es importante entender que bajo las condiciones apropiadas, el ADN podría hacerse monocatenario y el ARN puede ser bicatenario. De hecho, las moléculas son tan similares que incluso es posible crear moléculas híbridas bicatenarias con una cadena de ADN y otra de ARN. Curiosamente, las hélices dobles de ARN-ARN y las hélices dobles de ARN-ADN son en realidad ligeramente más estables que la doble hélice ADN-ADN más convencional.
La base de la naturaleza bicatenaria del ADN, y de hecho la base de los ácidos nucleicos como medio para el almacenamiento y transferencia de información genética, es el emparejamiento de bases. El emparejamiento de bases se refiere a la formación de enlaces de hidrógeno entre adeninas y timinas, y entre guaninas y citosinas. Estos pares son significativamente más estables que cualquier asociación formada con las otras bases posibles. Además, cuando estas asociaciones de pares de bases se forman en el contexto de dos cadenas de ácidos nucleicos, su espaciado también es uniforme y altamente estable. Tal vez recuerde que los enlaces de hidrógeno son enlaces relativamente débiles. Sin embargo, en el contexto del ADN, el enlace de hidrógeno es lo que hace que el ADN sea extremadamente estable y por lo tanto muy adecuado como medio de almacenamiento a largo plazo para la información genética. Ya que incluso en procariotas simples, las hélices dobles de ADN tienen al menos miles de nucleótidos de longitud, esto significa que hay varios miles de enlaces de hidrógeno que mantienen unidas las dos cadenas. Aunque cualquier interacción individual de enlace de hidrógeno de nucleótido a nucleótido podría interrumpirse temporalmente fácilmente por un ligero aumento de la temperatura, o un cambio minúsculo en la fuerza iónica de la solución, una doble hélice completa de ADN requiere temperaturas muy altas (generalmente superiores a 90 o C) para desnaturalizar completamente la doble hélice en hebras individuales.
Debido a que hay un emparejamiento exacto de nucleótidos uno a uno, resulta que las dos cadenas son esencialmente copias de respaldo entre sí, una red de seguridad en caso de que se pierdan nucleótidos de una cadena. De hecho, aunque partes de ambas hebras estén dañadas, siempre y cuando la otra hebra esté intacta en la zona de daño, entonces la información esencial sigue ahí en la secuencia complementaria de la hebra opuesta y puede escribirse en su lugar. Sin embargo, tenga en cuenta que si bien una hebra de ADN puede así actuar como un “respaldo” de la otra, las dos cadenas no son idénticas, son complementarias. Una consecuencia interesante de este sistema de hebras complementarias y antiparalelas es que las dos hebras pueden llevar cada una información única.
Los pares de genes bidireccionales son dos genes en cadenas opuestas de ADN, pero que comparten un promotor, que se encuentra entre ellas. Dado que el ADN solo se puede hacer en una dirección, 5' a 3', este promotor bidireccional, a menudo una isla CpG (ver siguiente capítulo), envía así la ARN polimerasa para cada gen en direcciones físicas opuestas. Esto se ha demostrado para una serie de genes involucrados en cánceres (mama, ovario), y es un mecanismo para coordinar la expresión de redes de productos génicos.
Las hebras de una doble hélice de ADN son antiparalelas. Esto significa que si observamos una doble hélice de ADN de izquierda a derecha, se construiría una hebra en la dirección 5' a 3', mientras que la cadena complementaria se construye en la dirección 3' a 5'. Esto es importante para la función de las enzimas que crean y reparan el ADN, ya que pronto estaremos discutiendo. En la Figura\(\PageIndex{1}\), la hebra izquierda es de 5' a 3' de arriba a abajo, y la otra es de 5' a 3' de abajo a arriba.
Desde un punto de vista físico, las moléculas de ADN están cargadas negativamente (todos esos fosfatos), y normalmente una doble hélice con un giro diestro. En este estado normal (también llamado conformación “B”), una torsión completa de la molécula abarca 11 pares de bases, con 0.34 nm entre cada base nucleotídica. Cada una de las bases nitrogenadas son planas, y cuando se emparejan con la base complementaria, forma un “peldaño” plano en la “escalera” del ADN. Éstas son perpendiculares al eje longitudinal del ADN. La mayor parte del ADN flotante en una célula, y la mayor parte del ADN en cualquier solución acuosa de osmolaridad y pH casi fisiológicos, se encuentra en esta conformación B. Sin embargo, se han encontrado otras conformaciones, generalmente bajo circunstancias ambientales muy específicas. Se observó una conformación comprimida, A-DNA, como artefacto de cristalización in vitro, con un poco más de bases por giro, longitud de giro más corta y pares de bases que no son perpendiculares al eje longitudinal. Otro, el Z-DNA, parece formarse transitoriamente en tramos de ADN ricos en GC en los que, curiosamente, el ADN se tuerce en dirección opuesta.
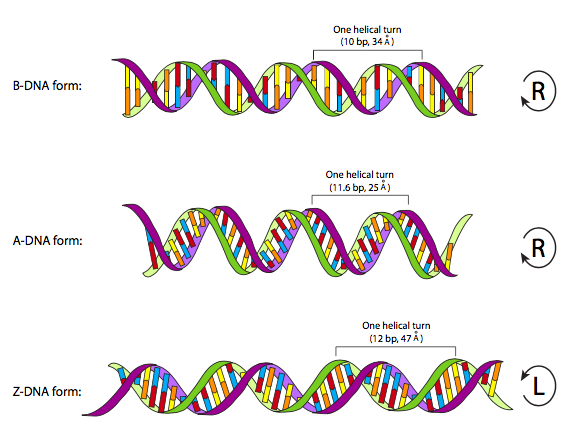
Se ha sugerido que tanto las formas A como Z del ADN son, de hecho, fisiológicamente relevantes. Hay evidencia que sugiere que la forma A puede ocurrir en hélices dobles híbridas ARN-ADN así como cuando el ADN está complejado con algunas enzimas. La conformación Z puede ocurrir en respuesta a la metilación del ADN. Además, la conformación “normal” del B-ADN es algo así como una estructura idealizada basada en estar completamente hidratada, ya que ciertamente es muy probable dentro de una célula. Sin embargo, ese estado de hidratación cambia constantemente, aunque minuciosamente, por lo que la conformación del ADN a menudo variará ligeramente de los parámetros de conformación B en la Figura\(\PageIndex{2}\).
En los procariotas, el ADN se encuentra en el citoplasma (bastante obvio ya que no hay otra opción en esos organismos simples), mientras que en los eucariotas, el ADN se encuentra dentro del núcleo. A pesar de las diferencias en sus ubicaciones, el nivel de protección frente a las fuerzas externas, y sobre todo, sus tamaños, tanto el ADN procariótico como el eucariota está empaquetado con proteínas que ayudan a organizar y estabilizar la estructura cromosómica general. Se entiende relativamente poco con respecto al empaquetamiento cromosómico procariótico aunque existen similitudes estructurales entre algunas de las proteínas que se encuentran en los cromosomas procariotas y eucariotas. Por lo tanto, la mayoría de los cursos introductorios de biología celular se adhieren al empaquetamiento cromosómico
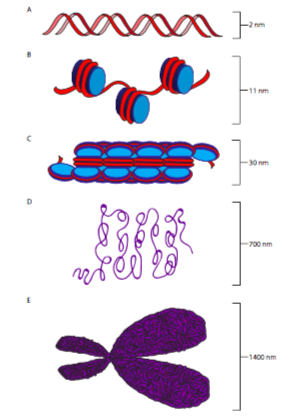
El ADN desnudo, ya sea procariota o eucariota, es una hebra extremadamente delgada de material, de aproximadamente 11 nm de diámetro. Sin embargo, dado el tamaño de los genomas eucariotas, si el ADN se almacenara de esa manera dentro del núcleo, se enredaría inmanejablemente. Imagínate un cubo en el que hayas arrojado cien metros de hilo sin ningún intento de organizarlo enrollándolo o agrupándolo. Ahora considere si sería capaz de alcanzar en ese cubo tirar de una hebra, y esperar tirar hacia arriba solo una hebra, o si en su lugar es probable que tire hacia arriba al menos una pequeña maraña de hilo. La celda hace esencialmente lo que harías con el hilo para mantenerlo organizado: se empaqueta ordenadamente en madejas más pequeñas y manejables. En el caso del ADN, cada cromosoma se enrolla alrededor de un complejo de histonas para formar el primer orden de organización cromosómica: el nucleosoma.
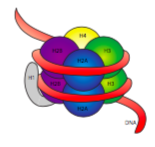
La fibra de 30 nm se mantiene unida por dos conjuntos de interacciones. Primero, el enlazador histona, H1, reúne los nucleosomas en una estructura aproximada de 30 nm. Esta estructura es estabilizada por enlaces disulfuro que se forman entre la histona H2A de un nucleosoma y la histona H4 de su vecino.
Las histonas son una familia de proteínas básicas (cargadas positivamente). Todos funcionan principalmente en la organización del ADN, y el nucleosoma se forma cuando el ADN se envuelve (un poco más de 2 veces) alrededor de un núcleo de ocho histonas, dos cada una de H2A, H2B, H3 y H4. El número y la posición de las cargas positivas (principalmente de lisinas y argininas) son cruciales para su capacidad de unirse fuertemente al ADN, que como se señaló anteriormente, tiene una carga muy negativa. Esa idea de “los opuestos se atraen” no es solo un consejo de citas de las columnas de consejos.

Al examinar la estructura 3D del complejo central de histonas, vemos que mientras que los dominios de interacción de proteínas relativamente no cargados mantienen las histonas juntas en el centro, los residuos cargados positivamente se encuentran alrededor del exterior del complejo, disponibles para interactuar con los fosfatos cargados negativamente de ADN.
En un capítulo posterior, discutiremos cómo las enzimas leen el ADN para transcribir su información en trozos de ARN más pequeños y manejables. Por ahora, sólo tenemos que ser conscientes de que en un momento dado, gran parte del ADN está empaquetado herméticamente, mientras que algunas partes del ADN no lo están. Debido a que las partes que están disponibles para su uso pueden variar dependiendo de lo que esté sucediendo a/en la célula en un momento dado, el empaque del ADN debe ser dinámico. Debe haber un mecanismo para aflojar rápidamente la unión del ADN a las histonas cuando ese ADN es necesario para la expresión génica, y para apretar la unión cuando no lo es. Resulta que este proceso implica acetilación y desacetilación de las histonas.
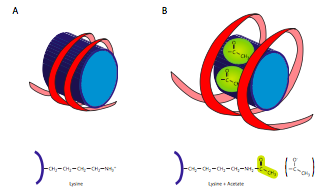
Las histonas acetiltransferasas (HAT) son enzimas que colocan un grupo acetilo en una lisina de una proteína histona. Los grupos acetilo están cargados negativamente, y la acetilación no solo agrega un grupo cargado negativamente, también elimina la carga positiva de la lisina. Esto tiene el efecto de no sólo neutralizar un punto de atracción entre la proteína y el ADN, sino incluso repelerlo ligeramente (con cargas similares). Por otro lado del mecanismo, las Histonas Deactilasas (HDAC) son enzimas que eliminan la acetilación, y con ello restauran la interacción entre la proteína histona y el ADN. Al tratarse de enzimas tan importantes, es lógico pensar que no se les permite operar de manera voluntaria sobre ninguna histona disponible, y de hecho, a menudo se encuentran en un complejo con otras proteínas que controlan y coordinan su activación con otros procesos como la activación de la transcripción.


