5.6: Mecanismos de Evolución Genómica
- Page ID
- 54122
Una vez que tenemos alineamientos de grandes regiones genómicas (o genomas completos) a través de múltiples especies relacionadas, podemos comenzar a hacer comparaciones para inferir las historias evolutivas de esas regiones.
Las tasas de evolución varían entre especies y regiones genómicas. En S. cerevisiae, por ejemplo, el 80% de las ambigüedades se encuentran en el 5% del genoma. Los telómeros son secuencias repetitivas de ADN al final de los cromosomas que protegen los extremos de los cromosomas del deterioro. Las regiones teloméricas son inherentemente inestables, tienden a sufrir una rápida evolución estructural, y el 80% de variación corresponde a 31 de las 32 regiones teloméricas. Las familias de genes contenidas dentro de estas regiones como HXT, FLO, COS, PAU e YRF muestran una evolución significativa en número, orden y orientación. Varias secuencias novedosas y codificantes de proteínas se pueden encontrar en estas regiones. Dado que se encuentran muy pocos reordenamientos genómicos en S. cerevisiae aparte de las regiones teloméricas, las regiones de cambio rápido pueden identificarse por expansiones de la familia de proteínas en los extremos cromosómicos.
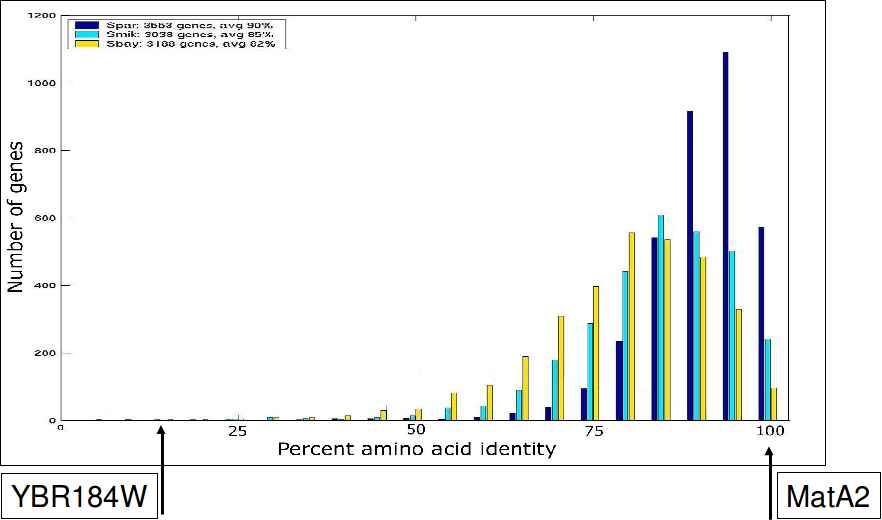
Figura 5.22: Vista dinámica de un gen cambiante.
Geness evoluciona a diferentes ritmos. Por ejemplo, como se ilustra en la Figura 5.22, en un extremo, hay YBR184W en la levadura que muestra una conservación de secuencia inusualmente baja y exhibe numerosas inserciones y deleciones entre especies. En el otro extremo se encuentra MatA2, que muestra una conservación perfecta de aminoácidos y nu- cleotide. Las tasas de mutación a menudo también varían según la clasificación funcional. Por ejemplo, las proteínas ribosómicas mitocondriales están menos conservadas que las proteínas ribosómicas.
El hecho de que algunos genes evolucionen más lentamente en una especie frente a otra puede deberse a factores como ciclos de vida más largos. La falta de cambio evolutivo en genes específicos, sin embargo, sugiere que existen funciones biológicas adicionales que son responsables de la presión para conservar la secuencia de nucleótidos. La levadura puede cambiar los tipos de apareamiento cambiando todos sus genes A y α y matA2 es uno de los cuatro genes de tipo apareamiento de levadura (matA2, Matα2, MatA1, Matα1). Su papel podría ser revelado potencialmente por análisis de conservación de nucleótidos.
Los genes de rápida evolución también pueden ser biológicamente significativos. Los mecanismos de cambio rápido de proteínas incluyen:
- Creación de dominios proteicos a través de tramos de Glutamina (Q) y Asparagina (N) e interacciones proteína-proteína,
- Desplazamientos compensatorios de fotogramas que permiten la exploración de nuevos marcos de lectura y la lectura/creación de señales de edición de ARN,
- Variaciones de codones de parada y lectura regulada donde las ganancias permiten cambios rápidos y las pérdidas pueden dar como resultado una nueva diversidad
- Las inteínas, que son segmentos de proteínas que pueden retirarse de una proteína y luego volver a unirse a la proteína restante, obtienen ganancias de transferencias horizontales de inteínas autoempalmantes postraduccionales.
Ahora analizamos las diferencias en el contenido de genes entre diferentes especies (S.cerevisiae, S.paradoxus, S.mikatae y S.bayanus.) Se puede revelar mucho sobre la pérdida y conversión de genes observando las posiciones de los parálogos a través de especies relacionadas y observando las tasas de cambio de los parálogos. Hay 8-10 genes únicos para cada genoma que están involucrados principalmente con el metabolismo, la regulación y el silenciamiento, y la respuesta al estrés. Además, hay cambios en la dosificación génica con duplicaciones tanto en tándem como en segmentos. Las expansiones de la familia de proteínas también están presentes con 211 genes con correspondencia ambigua. Sin embargo, en general, hay pocos genes novedosos en las diferentes especies.
Reordenamientos cromosómicos
Estos suelen estar mediados por mecanismos específicos como se ilustra para Saccharomyces en la Figura 5.23. [MattFox] Fig11cromovolimageissuperborrosasfarasicanse.dondequiera que se encontrara estohabía sido encontrado, debería ser colocadocon un alto
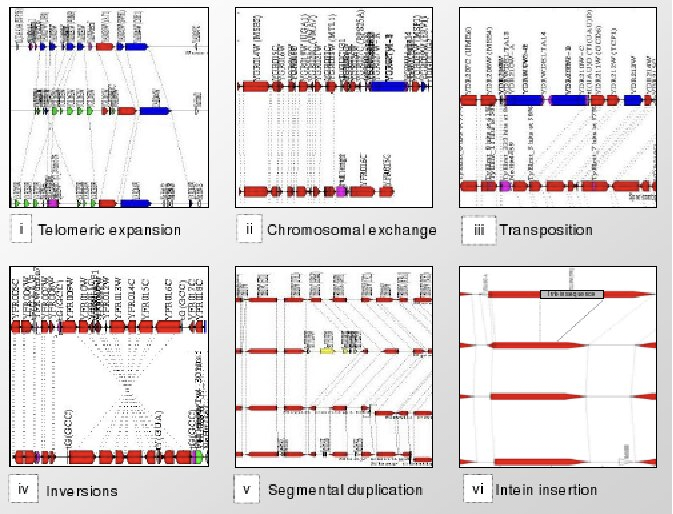
Figura 5.23: Mecanismos de evolución cromosómica.
Las translocaciones a través de genes diferentes a menudo ocurren a través de elementos genéticos transponibles (elementos Ty en levadura, por ejemplo). Las ubicaciones del transposón se conservan con inserciones recientes que aparecen en ubicaciones antiguas y restos de repeticiones terminales largas encontrados en otros genomas. Sin embargo, son evolutivamente activos (por ejemplo, siendo recientes los elementos Ty en la levadura), y normalmente aparecen en un solo genoma. La ventaja evolutiva de tales transposones conservados locacionalmente puede estar en la posibilidad de mediar arreglos reversibles. Las inversiones a menudo están flanqueadas por genes de ARNt en orientación transcripcional opuesta. Esto puede sugerir que se originan a partir de la recombinación entre genes de ARNt.


