4.3: Proteínas de transporte de membrana
- Page ID
- 53439
Las proteínas de membrana vienen en dos tipos básicos: proteínas integrales de membrana (a veces llamadas intrínsecas), que se insertan directamente dentro de la bicapa fosfolipídica, y proteínas de membrana periférica (a veces llamadas extrínsecas), que se localizan muy cerca o incluso en contacto con una cara de la membrana, pero no se extienden en el núcleo hidrófobo de la bicapa. Las proteínas integrales de membrana pueden extenderse completamente a través de la membrana en contacto tanto con el entorno extracelular como con el citoplasma, o pueden insertarse solo parcialmente en la membrana (a cada lado) y contactar solo con el citoplasma o el entorno extracelular. No se conocen proteínas que estén completamente enterradas dentro del núcleo de la membrana.
Las proteínas integrales de membrana (Figura\(\PageIndex{9}\)) se mantienen firmemente en su lugar por fuerzas hidrófobas, y la purificación de las mismas a partir de los lípidos requiere agentes disruptores de membrana tales como disolventes orgánicos (por ejemplo, metanol) o detergentes (por ejemplo, SDS, Triton X-100). Debido a la naturaleza de la bicapa, la porción de proteínas integrales de membrana que se encuentran dentro del núcleo hidrófobo de la membrana suelen ser de carácter muy hidrófobo, o tienen residuos hidrófobos orientados hacia afuera para interactuar con el núcleo de la membrana. Estos dominios transmembrana suelen tomar una de las dos formas representadas en las Figuras 8 y 14: hélices alfa -ya sea individualmente o en un conjunto con otras hélices alfa, o inserciones en forma de barril en las que las paredes del cañón están construidas de láminas plegadas beta. Las inserciones hidrófobas están unidas por una serie corta de residuos polares o cargados que interactúan con el ambiente acuoso y los grupos de cabeza polar para evitar que la porción hidrófoba de la proteína se deslice fuera de lugar. Además, las proteínas pueden tener múltiples dominios que abarcan la membrana.
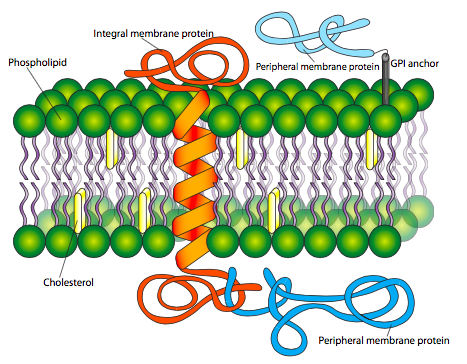
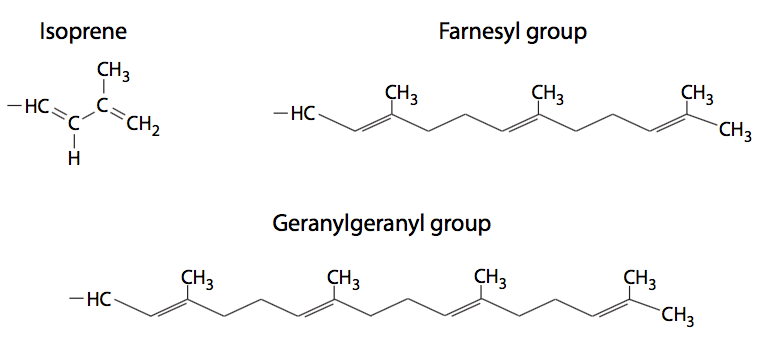
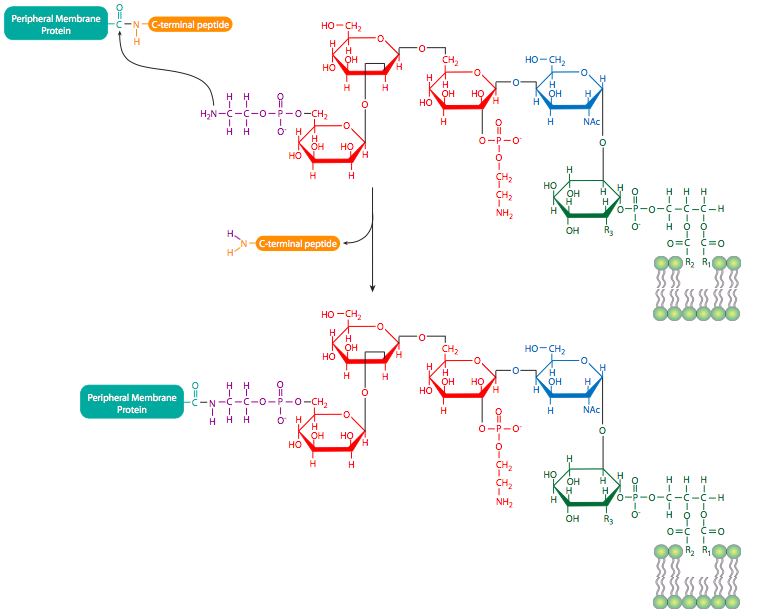
Las proteínas de membrana periférica (también mostradas en la Figura\(\PageIndex{9}\)) son menos predecibles en su estructura, pero pueden unirse a la membrana ya sea por interacción con proteínas integrales de membrana o por lípidos unidos covalentemente. Las modificaciones más comunes a las proteínas de membrana periférica son la acilación grasa, la prenilación y la unión a anclajes de glicosilfosfatidilinositol (GPI). La acilación grasa suele ser una miristoilación (una cadena de acilo 14:0) y palmitoilación (una cadena 16:0) de la proteína. Una proteína puede estar acilada con más de una cadena, aunque uno o dos grupos acilo es el más común. Estas cadenas de acilo graso se insertan establemente en el núcleo de la bicapa fosfolipídica. Si bien las proteínas miristoiladas se encuentran en una variedad de compartimentos, casi todas las proteínas palmitoiladas se encuentran en la cara citoplasmática de la membrana plasmática. Las proteínas preniladas, por otro lado, se encuentran principalmente unidas a membranas intracelulares. La prenilación es la unión covalente de los isoprenoides a la proteína, más comúnmente los grupos isopreno (un hidrocarburo C5), farnesilo (C15) o geranilgeranilo (C20) (Figura\(\PageIndex{10}\)). Los anclajes GPI (Figura\(\PageIndex{11}\)) se encuentran exclusivamente en proteínas en la superficie externa de la célula, pero no parece haber ninguna otra similitud en sus estructuras o funciones.
Por supuesto, no todas las proteínas de membrana, o incluso todas las proteínas transmembrana, son transportadoras, y las muchas otras funciones de las proteínas de membrana -como receptores, moléculas de adhesión, moléculas de señalización y moléculas estructurales- serán discutidas en captadores posteriores. El enfoque aquí está en el papel de las proteínas de membrana para facilitar el transporte de moléculas a través de la membrana celular.
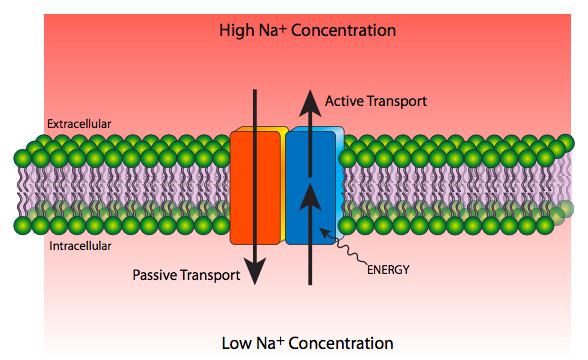
El transporte a través de la membrana puede ser pasivo, no requiriendo ninguna fuente externa de energía ya que el soluto viaja de alta a baja concentración, o activo, requiriendo gasto de energía a medida que el soluto viaja de baja a alta concentración (Figura\(\PageIndex{12}\)).
El transporte pasivo también se puede dividir en transporte no mediado, en el que el movimiento de los solutos se determina únicamente por difusión, y el soluto no requiere una proteína de transporte, y transporte pasivo mediado (también conocido como difusión facilitada) en el que se requiere una proteína de transporte para ayudar a un soluto a pasar de concentración alta a baja. Aunque esto a veces puede implicar un cambio en la conformación, no se requiere energía externa para este proceso. El transporte pasivo no mediado se aplica solo a pequeñas moléculas no polares solubles en membrana, y la cinética del movimiento está regida por la difusión, el grosor de la membrana y el potencial electroquímico de la membrana. El transporte activo es siempre un proceso de transporte mediado.
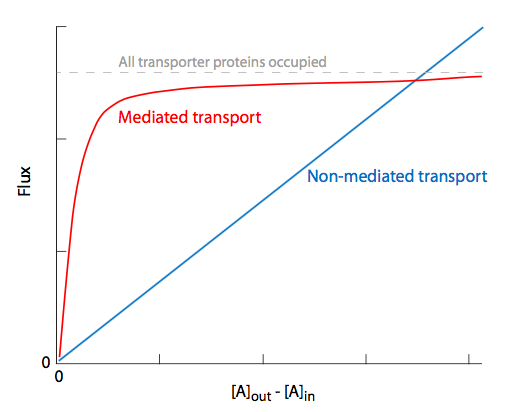
Comparando el flujo de soluto vs concentración inicial en la Figura\(\PageIndex{13}\), vemos que existe una relación lineal para el transporte no mediado, mientras que el transporte pasivo mediado (y para el caso, el transporte activo) muestra un efecto de saturación debido al factor limitante del número de proteínas disponibles para permitir el soluto a través. Una vez que haya suficiente soluto para ocupar constantemente todos los transportadores o canales, se alcanzará el ux máximo, y los aumentos de concentración no podrán superar este límite. Esto es cierto independientemente del tipo de proteína transportadora involucrada, aunque algunas están más íntimamente involucradas en el transporte que otras.
Además de los transportadores de proteínas, existen otras formas de facilitar el movimiento de iones a través de las membranas. Los ionóforos son pequeñas moléculas orgánicas, a menudo (pero no exclusivamente) hechas por bacterias, que ayudan a que los iones se muevan a través de las membranas. Muchos ionóforos son antibióticos que actúan haciendo que las membranas se vuelvan permeables a iones particulares, alterando el potencial electroquímico de la membrana y la composición química dentro de la célula. Los ionóforos son exclusivamente mecanismos de transporte pasivo, y se agrupan en dos tipos.
El primer tipo de ionóforo es un pequeño portador principalmente hidrofóbico casi completamente incrustado en la membrana, que se une y envuelve un ion especi c, protegiéndolo del lípido, y luego lo mueve a través de la membrana celular. El ionóforo de tipo portador más estudiado es la valinomicina, que se une a K+. La valinomicina es un depsipéptido cíclico de 12 residuos (contiene enlaces amida y éster) con aminoácidos alternantes d y l-. Todos los grupos carbonilo miran hacia adentro para interactuar con el ion, mientras que las cadenas laterales hidrófobas miran hacia afuera hacia el lípido de la membrana. Los ionóforos portadores no son necesariamente péptidos: el químico industrial 2,4-dinitrofenol es un portador H + e importante preocupación por los desechos ambientales, y la nistatina, un antifúngico utilizado para tratar infecciones por Candida albicans en humanos, es un portador K +.
El segundo tipo de portador forma canales en la membrana diana, pero nuevamente, no es una proteína. La gramicidina es un ejemplo prototípico, antibacteriano antigrampositivo (excepto por la fuente de gramicidinas, el Bacillus brevis grampositivo) y canal ionóforo para cationes monovalentes como Na +, K + y H +. Es permeable a los aniones, y puede ser bloqueada por el catión divalente Ca 2+. Al igual que la valinomicina, la gramicidina A también está compuesta por d- y l- aminoácidos alternantes, todos los cuales son hidrófobos (L-Val/ Ile-Gly-L-Ala-D-Leu-L-Ala-D-Val-L-Val-D-Val-L-Trp-D-Leu-L-Trp-D-Leu-L-Trp). La gramicidina A se dimeriza en la membrana para formar una estructura de lámina b comprimida conocida como hélice b. La dimerización se forma de N-terminal a N-terminal, colocando los residuos de Trp hacia los bordes externos de la membrana, con los grupos NH polares hacia las superficies extracelular y citoplásmica, anclando el poro en su lugar.
Los canales son esencialmente sistemas de transporte de manos libres que, como su nombre lo indica, proporcionan un paso de un lado de la celda a otro. Aunque los canales pueden estar cerrados, capaces de abrirse y cerrarse en respuesta a cambios en el potencial de membrana o la unión al ligando, por ejemplo, permiten que los solutos pasen a una velocidad alta sin unirlos estrechamente y sin cambios en la conformación. El soluto solo puede moverse a través de canales de alta a baja concentración. El canal de potasio representado a continuación (Figura\(\PageIndex{14}\) A) es un ejemplo: existe una selectividad lter (14B) de oxígenos carbonílicos alineados que posiciona transitoriamente los iones K+ para un paso rápido a través del canal, pero no se une al K + por ningún periodo significativo, ni el canal sufre cualquier cambio conformacional como resultado de la interacción. Los iones Na + más pequeños podrían (y en raras ocasiones lo hacen) pasar por el canal K+, pero debido a que son demasiado pequeños para ser posicionados correctamente por el filtro K +, generalmente vuelven a aparecer. Cabe señalar que este canal es un tetrámero (14C) y el diagrama de corte en (14A) solo muestra la mitad del canal para mayor claridad.
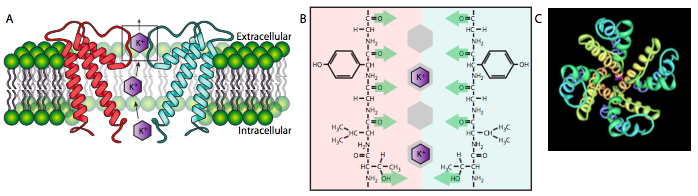
Si bien la mayoría de las proteínas llamadas “canales” están formadas por múltiples hélices alfa, las porinas están formadas por una lámina beta cilíndrica. En ambos casos, los solutos solo pueden bajar el gradiente de concentración de alto a bajo, y en ambos casos, los solutos no hacen contacto de signi cant con el poro o canal. El interior del poro suele ser hidrófilo debido a la alternancia de residuos hidrófilos/hidrófobos a lo largo de la cinta beta, lo que coloca las cadenas laterales hidrófobas en el exterior, interactuando con el núcleo de la membrana.
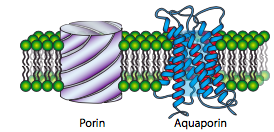
Las porinas se encuentran principalmente en bacterias gramnegativas, algunas bacterias grampositivas y en las mitocondrias y cloroplastos de eucariotas. Generalmente no se encuentran en la membrana plasmática de eucariotas. También, a pesar de la similitud en el nombre, están estructuralmente no relacionados con las acuaporinas, que son canales que facilitan la difusión del agua dentro y fuera de las células.
Las proteínas de transporte funcionan de manera muy diferente a los canales o poros. En lugar de permitir un flujo relativamente rápido de solutos a través de la membrana, las proteínas de transporte mueven los solutos a través de la membrana en cuantos discretos uniéndose al soluto en un lado de la membrana, cambiando la conformación para llevar el soluto al otro lado de la membrana, y luego liberando el soluto. Estas proteínas transportadoras pueden trabajar con moléculas individuales de soluto como los transportadores de glucosa, o pueden mover múltiples solutos. Los transportadores de glucosa son proteínas de transporte pasivo, por lo que solo mueven la glucosa de concentraciones mayores a menores, y no requieren una fuente de energía externa. Las cuatro isoformas son muy similares estructuralmente pero difieren en su distribución tisular dentro del animal: por ejemplo, GLUT2 se encuentra principalmente en las células b pancreáticas, mientras que GLUT4 se encuentra principalmente en las células musculares y grasas.
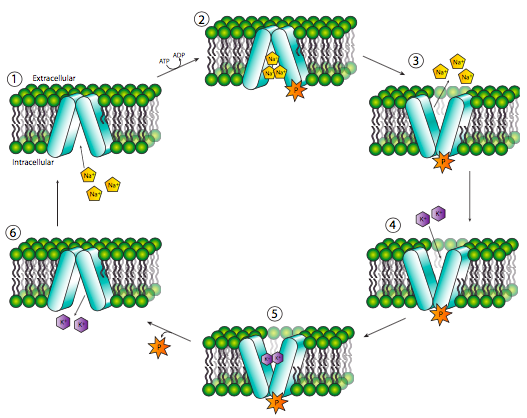
Por otro lado, el ejemplo clásico de una proteína de transporte activa, la Na + /K + ATPasa, también conocida como antipuerto Na + /K +, utiliza la energía de la hidrólisis de ATP para potenciar los cambios conformacionales necesarios para mover tanto Na + como K + iones contra el gradiente. Haciendo referencia a la Figura\(\PageIndex{16}\), en su estado de reposo, la ATPasa Na + /K + está abierta al citoplasma y puede unirse a tres iones Na + (1). Una vez que los tres Na + se han unido, el transportador puede catalizar la hidrólisis de una molécula de ATP, eliminando un grupo fosfato y transfiriéndolo a la propia ATPasa (2). Esto desencadena un cambio conformacional que abre la proteína al espacio extracelular y también cambia el sitio de unión de iones para que Na + ya no se une con alta afinidad y cae (3). Sin embargo, la especificidad del sitio de unión a iones también se ve alterada en este cambio conformacional, y estos nuevos sitios tienen una alta afinidad por los iones K + (4). Una vez que los dos K + se unen, se libera el grupo fosfato unido (5) y otro cambio conformacional vuelve a colocar la proteína transportadora en su conformación original, alterando los sitios de unión de K + para permitir la liberación de la K + en el citoplasma (6), y revelando Na + afinidad una vez más.
La ATPasa Na + /K + es un miembro de la familia de ATPasas tipo P. Se les llama por la autofosforilación que se produce cuando se hidroliza ATP para impulsar el transporte. Otros miembros destacados de esta familia de ATPasas son la Ca 2+ -ATPasa que bombea Ca 2+ fuera del citoplasma hacia orgánulos o fuera de la célula, y la H+ /K + ATPasa, aunque también hay bombas de tipo P H + en membranas plasmáticas fúngicas y vegetales, y en bacterias.
Los glucósidos cardíacos (también esteroides cardíacos) inhiben la ATPasa Na + /K + al unirse al lado extracelular de la enzima. Estos medicamentos, incluyendo la digitalis (extraída de la planta dedalera morada) y la ouabaína (extraída del árbol de ouabio) son comúnmente recetados medicamentos cardíacos que aumentan la intensidad de las contracciones cardíacas. La inhibición de Na + /K + ATPasa provoca un aumento en [Na +] en el que luego activa los antipuertos cardíacos de Na + /Ca 2+, bombeando el exceso de sodio y Ca 2+ adentro. El aumento del citoplasma [Ca 2+] es absorbido por el retículo sarcoplásmico, lo que lleva a Ca 2+ extra cuando se libera para desencadenar la contracción muscular, causando contracciones más fuertes.
A diferencia de Na + o K +, el gradiente de Ca 2+ no es muy importante con respecto al potencial electroquímico de membrana o el uso de su energía. Sin embargo, la regulación estricta del Ca 2+ es importante de una manera diferente: se usa como señal intracelular. Para optimizar la efectividad del Ca 2+ como señal, sus niveles citoplásmicos se mantienen extremadamente bajos, con bombas de Ca 2+ empujando el ion hacia el ER (SR en músculos), Golgi y fuera de la célula. Estas bombas están reguladas por niveles de Ca 2+ a través de la proteína calmodulina. A niveles bajos de Ca 2+, la bomba está inactiva y un dominio inhibitorio de la bomba en sí impide su actividad. Sin embargo, a medida que aumentan los niveles de Ca 2+, los iones se unen a la calmodulina, y el complejo Ca 2+ -calmodulina puede unirse a la región inhibitoria de la bomba de Ca 2+, aliviando la inhibición y permitiendo que el exceso de Ca 2+ sea bombeado fuera del citoplasma.
Hay otras tres familias de ATPasas: las ATPasas tipo F son bombas de protones en bacterias y mitocondrias y cloroplastos que también pueden funcionar para formar ATP corriendo “hacia atrás” con protones debidos a través de ellos por el gradiente de concentración. Serán discutidos en el próximo capítulo (Metabolismo). También hay ATPasas tipo V que regulan el pH en vesículas ácidas y vacuolas vegetales, y finalmente, hay ATPasas transportadoras de aniones.
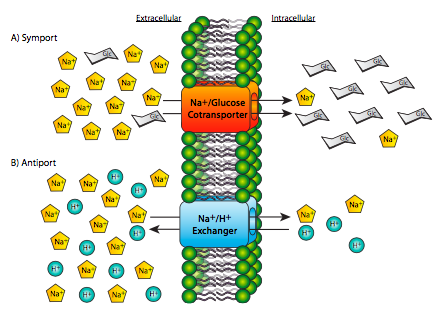
La hidrólisis del ATP, si bien es una fuente común de energía para muchos procesos biológicos, no es la única fuente de energía para el transporte. El transporte activo de un soluto contra su gradiente puede acoplarse con la energía del transporte pasivo de otro soluto hacia abajo en su gradiente. Dos ejemplos se muestran en la Figura\(\PageIndex{17}\): aunque uno es un simport (ambos solutos cruzan la membrana en la misma dirección física) y uno es un antipuerto (los dos solutos cruzan la membrana en direcciones físicas opuestas), ambos tienen un soluto que se desplaza hacia abajo por su gradiente, y un soluto viajando contra su gradiente de concentración. Como sucede, hemos utilizado el movimiento Na + como la fuerza impulsora detrás de ambos ejemplos. De hecho, el gradiente de Na + a través de la membrana es una fuente de energía extremadamente importante para la mayoría de las células animales. Sin embargo esto no es universal para todas las células, ni siquiera para todas las células eucariotas. En la mayoría de las células vegetales y organismos unicelulares, el gradiente H + (protón) juega el papel que Na + hace en los animales.
Los receptores de acetilcolina (AChR), que se encuentran en algunas neuronas y en las células musculares en las uniones neuromusculares, son canales iónicos regulados por ligandos. Cuando el neurotransmisor (acetilcolina) o un agonista como la nicotina (para receptores de tipo nicotínico) o muscarina (para receptores de tipo muscarínico) se une al receptor, abre un canal que permite el ow de cationes pequeños, principalmente Na + y K +, en direcciones opuestas, por supuesto. La precipitación de Na + es mucho más fuerte y conduce a la despolarización inicial de la membrana que o bien inicia un potencial de acción en una neurona, o en el músculo, inicia la contracción.


