5.4: Fosforilación oxidativa
- Page ID
- 52934
La fosforilación oxidativa denota la fosforilación de ADP en ATP, utilizando la energía de sucesivos transportes de electrones (de ahí el “oxidativo”). El concepto básico es que la oxidación del NADH, al ser altamente exergónico, puede generar la energía necesaria para fosforilar el ADP. Dado que la oxidación de NADH por oxígeno puede liberar potencialmente 52 Kcal/mol (218 kJ/mol), y la energía necesaria para fosforilar ATP es aproximadamente 7.5 Kcal/mol (30.5 kJ/mol), deberíamos ser capaces de esperar la formación de varios ATP por NADH oxidado. En efecto, esto es lo que sucede, aunque no directamente. Como se señaló con la descomposición de la glucosa, una oxidación en un solo paso generaría demasiada energía para que los procesos celulares la manejaran, y la mayoría se desperdiciaría. Entonces, en lugar de oxidar el NADH directamente con O 2, los electrones se transfieren a una serie de portadores de menor energía gradualmente hasta llegar finalmente al oxígeno. Esta secuencia es la cadena de transporte de electrones.
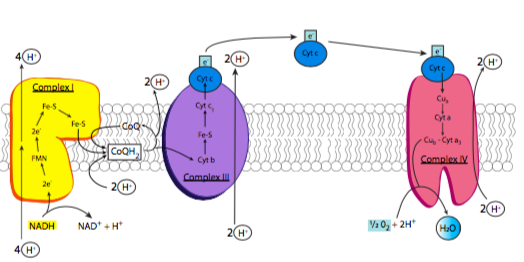
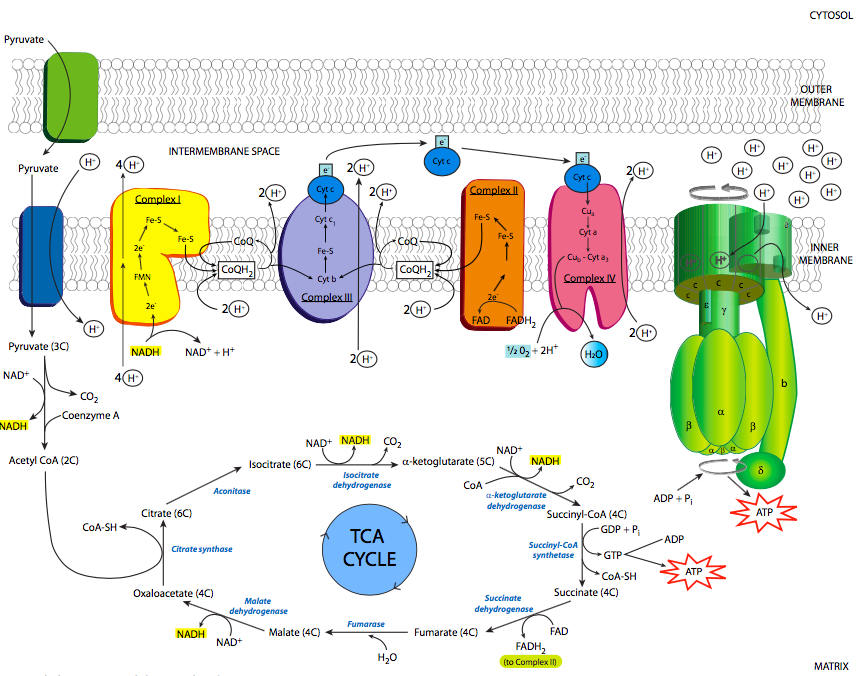
La cadena de transporte de electrones se basa en la actividad de cuatro complejos enzimáticos principales (convenientemente llamados complejos I-IV) incrustados en la membrana mitocondrial interna, junto con algunos pequeños portadores de electrones fácilmente difusibles para mover los electrones de un complejo al siguiente. Estos complejos están presentes en números extremadamente altos como corresponde a su necesidad de generar energía, que comprenden casi el 75% de la masa de la membrana interna (en comparación, la membrana plasmática de una célula eucariota promedio tiene una concentración de proteína más cercana al 50%). En la Figura se muestra una visión general del proceso\(\PageIndex{6}\): como se señaló anteriormente, los electrones son despojados del NADH, y eventualmente terminan en oxígeno. A medida que los electrones se mueven a portadores de menor energía, la energía se libera y se usa para bombear protones desde la matriz mitocondrial al espacio intermembrana.
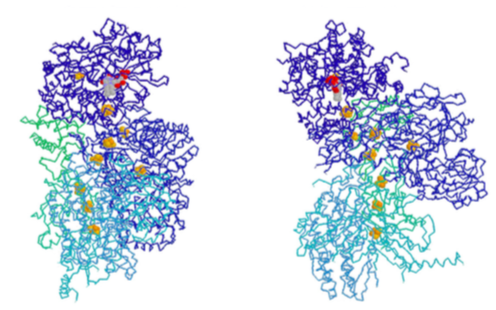
El complejo I es una NADH deshidrogenasa. Mostrado en amarillo en la Figura\(\PageIndex{6}\), su propósito es eliminar un par de electrones del NADH y transferirlos a ubiquinona (Coenzima Q o CoQ), un pequeño portador de electrones hidrófobo que luego puede llevar los electrones al complejo III. Este es un proceso de múltiples etapas que implica primero transferir los electrones a una molécula asociada de mononucleótido de flavina (FMN), que luego transfiere los electrones a un conjunto de restos hierro-azufre conectados al propio complejo enzimático (estructura en la Figura\(\PageIndex{7}\)). Finalmente, los electrones se mueven hacia la ubiquinona. A medida que ocurren estas transferencias, la energía que se libera durante estas transferencias alimenta el bombeo de 4 iones H+ a través de la membrana mitocondrial interna. El complejo I es inhibido por la rotenona, un pesticida que se usa principalmente contra insectos y ella.
Tomaremos un pase mental en el complejo II por ahora y lo golpearemos al final de esta lista. Entonces serán evidentes las razones.
El complejo III también se conoce como el complejo citocromo bc 1 (Figura\(\PageIndex{6}\), púrpura). El propósito de este complejo es pasar los electrones de la ubiquinona al citocromo c. El uso de ubiquinona es importante aquí, porque es estable con dos, o solo uno, electrones extra. El citocromo c, por otro lado, sólo puede portar un electrón. Entonces, este complejo atraca la ubiquinona, y la retiene hasta que ha pasado su primer electrón al citosoma c, que luego pasa al complejo IV, y luego su segundo electrón a otro citocromo c. Con cada transferencia, se bombean dos protones a través de la membrana.
Finalmente, el citocromo c deja caer el electrón al complejo IV, citocromo c oxidasa (Figura\(\PageIndex{6}\), rojo). La citocromo c oxidasa logra el paso final: transferir electrones a átomos de oxígeno para hacer agua. Lo realmente interesante de este proceso es que la enzima debe aferrarse a los electrones ya que son transferidos uno a la vez desde el citocromo c, hasta que contiene cuatro electrones. Entonces, puede transferir un par a cada uno de los átomos de oxígeno en el oxígeno molecular (O 2). Es muy importante hacer esto porque transferir menos de los cuatro electrones conduciría a la creación de especies reactivas de oxígeno (ROS) que podrían causar daños a las enzimas y membranas de las mitocondrias.
De hecho, algunos venenos bien conocidos actúan exactamente en este punto. Tanto el cianuro como el monóxido de carbono pueden unirse con mayor afinidad que el oxígeno en el hemo en el complejo IV. Como ninguno puede aceptar electrones, el efecto es como si no hubiera oxígeno disponible.
Aunque la citocromo c oxidasa a veces se abrevia COX, no es el objetivo de los inhibidores de la COX-2 que se usan de manera farmacéutico en el manejo del dolor, por ejemplo, Bextra, Celebrex o Vioxx. Eso se refiere a una familia de enzimas conocidas como las ciclooxigenasas.
El oxígeno es absolutamente necesario. Si no hay oxígeno disponible, no hay lugar para transferir los electrones, y muy rápidamente, la cadena de transporte de electrones se detiene y portadores como el citocromo c y CoQ no pueden liberar sus electrones y eventualmente no hay más portadores disponibles. De igual manera, cuando eso sucede, el NAD + no se regenera, por lo que el ciclo de TCA también está atascado. Esto deja solo el ciclo anaeróbico de glucólisis- fermentación que no requiere oxígeno para generar ATP.
Ahora volvemos al complejo II (ver Figura\(\PageIndex{10}\)). Se mencionó el complejo II como succinato deshidrogenasa al discutir el ciclo de TCA. También participa en la cadena de transporte de electrones pasando electrones a ubiquinona. Sin embargo, en lugar de transferir electrones que se originaron del NADH como los otros tres complejos de la cadena de transporte de electrones, los electrones se originan del portador de electrones unido covalentemente FADH2 (dinucleótido de flavina adenina), que recibió los electrones del succinato, como se describe en el ciclo de TCA sección. Una vez que los electrones han pasado a la ubiquinona, luego pasa al complejo III para dejar esos electrones al citocromo c, y el resto de la cadena de transporte de electrones continúa. El FAD, la forma oxidada de FADH2, está entonces lista para participar en el siguiente ciclo redox.
El propósito de esta cadena de transporte de electrones, con respecto a la generación de ATP, es el bombeo de H+ desde la matriz mitocondrial hacia el espacio intermembranoso. Dado que la concentración de protones es mayor en el espacio intermembrana, se necesitará energía para moverlos contra el gradiente de concentración, que es donde entran en escena nuestros electrones de alta energía. A medida que pasan de un portador a otro, van pasando de un estado energético superior a uno más bajo. Esto implica que se pierde algo de energía del electrón, y parte de esa energía es aprovechada por las enzimas de la cadena de transporte de electrones para mover protones de la matriz al espacio intermembrana.
Existen dos métodos mediante los cuales se mueven los protones: el bucle redox y la bomba de protones. La bomba de protones, que es el método por el cual el complejo IV mueve protones, es la más fácil de entender: H + se une en el lado de la matriz de la enzima en su estado reducido (después de haber recibido un electrón), y se produce un cambio conformacional tras la reoxidación para abrir la enzima hasta la intermembrana lado, y se libera el H +. El bucle redox, que ocurre en el complejo I, y en el complejo III en una variación llamada ciclo Q, esencialmente postula que un centro redox inicial requiere la unión tanto del electrón de alta energía como de un protón desde el lado de la matriz de la membrana. Cuando el electrón se transfiere al siguiente centro redox en la cadena, se libera un protón al espacio intermembrana.
Sea cual sea el mecanismo, ¿cuál es el sentido de todo este bombeo de protones? Como podrías sospechar, usar energía para bombear un ion contra su gradiente de concentración no se hace por diversión. Más bien, esto genera energía potencial significativa a través de la membrana mitocondrial interna. Y, da la casualidad de que existe una enzima que puede convertir esa energía en la forma química fisiológicamente útil del ATP. Esta enzima es, no sorprendentemente, denominada ATP sintasa (Figura\(\PageIndex{8}\)). También se le conoce en algunos textos como la F1F0-ATPasa, basada en su actividad inversa (a expensas del ATP, puede bombear protones), y el hecho de que se puede descomponer en dos unidades funcionales principales: F1 que puede hidrolizar pero no sintetizar ATP y es una proteína soluble, y F0 que es una insoluble proteína transmembrana.
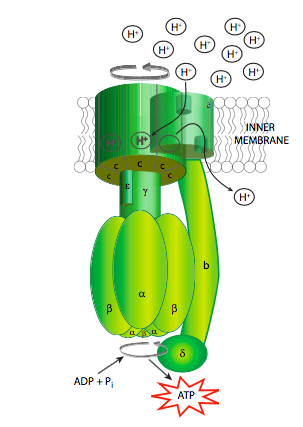
La ATP sintasa es un ejemplo extraordinario de una enzima que transforma la energía inherente a un gradiente de concentración a través de una membrana en energía mecánica y finalmente en energía de enlace químico. Se le llama descriptivamente un “motor rotativo” porque la secuencia muy generalizada de eventos es la siguiente: los protones bajan su gradiente a través de una subunidad de canal de protones de la ATP sintasa, en debido al gradiente, se libera energía, esta energía provoca la rotación de una subunidad similar a una “rueda” multisubunidad unido a un husillo/eje (subunidad g) que también gira. El giro de esta unidad de husillo de forma asimétrica provoca cambios conformacionales en la subunidad catalítica (compuesta por las subunidades a y b) a la que está unida, cambiando un sitio de unión de ADP+Pi a un sitio catalítico que puede “exprimir” las moléculas juntas en un ATP, y luego finalmente abrirse para liberar el ATP ( Figura\(\PageIndex{9}\)).
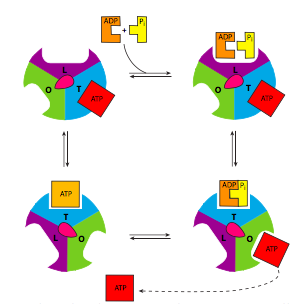
Por supuesto, no es tan sencillo (Figura\(\PageIndex{8}\)). Comenzando con el movimiento inicial de las pro- toneladas, a medida que se mueven desde el espacio intermembrana hacia la ATP sintasa, ingresan a un pequeño canal hidrófilo (a) y luego se unen a una de las subunidades c del anillo en C de la “rueda hidráulica”. La unión de la H + a la subunidad c provoca que pierda afinidad por la subunidad a-, lo que le permite girar, y simultáneamente provoca un cambio conformacional que esencialmente empuja contra la subunidad a, iniciando el movimiento. Una vez que ha girado casi un giro completo, el H + se posiciona por otro canal (b), que lo canaliza de la subunidad c a la matriz. La estructura de la subunidad c está conectada a un husillo asimétrico que a su vez está conectado a las subunidades catalíticas.


