8.3: Transcripción eucariota
- Page ID
- 53298
La transcripción en eucariotas es más complicada, pero sigue las mismas ideas generales. Las secuencias promotoras son mucho más variadas tanto en ubicación (con respecto al sitio de inicio) como en tamaño. Como veremos en el próximo capítulo, los genes eucariotas tienen muchos más elementos de control que regulan su expresión que los genes procariotas. No sólo hay más elementos de control, también hay más ARN polimerasas, que sirven diferentes funciones celulares específicas. Obviamente, la amplia función y localización de todas las ARN polimerasas es la misma: leer un molde de ADN y transcribir una copia de ARN del mismo; y dado que el ADN se encuentra sólo en el núcleo, también lo son las polimerasas. Sin embargo, las polimerasas difieren exactamente en qué tipos de ARN producen. La poli- merasa I de ARN está especializada en la producción de pre-ARNr (ARNr = ARN ribosómico). El pre-ARNr se escinde postranscripcionalmente y se incorpora a los ribosomas. Dado que los ribosomas se ensamblan en el nucleolo, esa es la parte del núcleo en la que se concentra la mayor parte de la ARN Polimerasa I. La ARN Polimerasa III también produce un ARN (5S) que se incorpora al ribosoma. También produce otros ARN no traducidos como los ARNt y una variedad de ARN nucleares pequeños. La única ARN polimerasa que hace que el ARN traducible (ARNm, o ARN mensajero) que la mayoría de la gente piensa cuando se refiere genéricamente al ARN, es la ARN polimerasa II. Esta es la ARN polimerasa que produce pre-ARNm, que después de algún procesamiento, se convierte en ARNm, se transporta fuera del núcleo, y finalmente se traduce en proteínas. Todas las ARN polimerasas eucariotas están compuestas por dos subunidades grandes, aproximadamente análogas a las subunidades β y β' de la RNAP procariota, pero en lugar de solo tres o cuatro subunidades, hay más de una docena de subunidades más pequeñas a las holoenzimas de la ARN polimerasa eucariota.
El inicio de la transcripción también es mucho más complicado. No sólo existe gran variedad en promotores reconocidos por RNAP II, tanto RNAP I como RNAP III reconocen promotores con características estructurales particulares.
Las ARN polimerasas eucariotas se denominaron I, II y III según su orden de elución a partir de la purificación por cromatografía de intercambio iónico. También son parcialmente distinguibles por su sensibilidad a la α-amanitina y a los venenos relacionados con hongos de la familia amatoxina. La RNAP I (y la RNAP procariota) es insensible a estas toxinas, la RNAP III es algo sensible (K d ~10 -6 M) y la RNAP II es altamente sensible (K d ~10 -8 M). Estas toxinas actúan uniéndose a un sitio en la hendidura ARN-ADN e interfiriendo con la translocación del ARN. Es decir, no hay problema con importar un nucleótido o con unirlo al nuevo ARN, pero la cadena de ARN no puede moverse a través del sitio activo y permitir que se agregue el siguiente nucleótido.
Uno de los promotores eucariotas RNAP II más comunes es la caja TATA, llamada así por el motivo altamente conservado que la define. Aunque parece similar a la caja Pribnow en procariotas, generalmente se localiza más aguas arriba del sitio de inicio, y su posición es mucho más variable. Mientras que la caja Pribnow se encuentra en -10, la caja TATA puede estar ubicada más cerca de -30 +/- 4. Además, en lugar de solo un factor sigma para reconocer el promotor junto con la enzima del núcleo de la polimerasa, el promotor eucariota es reconocido por un complejo de múltiples subunidades llamado factor de transcripción IID (TFIID). El TFIID está compuesto por proteína de unión a TATA (TBP) y varios factores asociados a TBP (TAF).
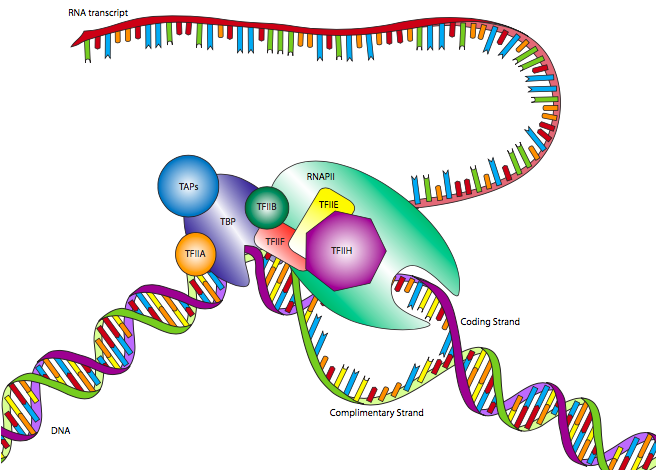
Esta unión del promotor por TFIID ocurre independientemente de la ARN Polimerasa II, y de hecho, RNAP II no se unirá a TFIID en este momento. Después de que TFIID se haya unido a la caja TATA, dos factores de transcripción más, TFIIA y TFIIB, se unen al TFIID así como al ADN cercano, estabilizando el complejo. TFIIF se une a TFIID y TFIIB para permitir el acoplamiento de la ARN Polimerasa II. El complejo aún no está listo para comenzar la transcripción: se requieren dos factores más. TFIIE se une a TFIIF y RNAP II, y finalmente, TFIIH se une a RNAP II, proporcionando una actividad helicasa necesaria para separar las dos cadenas del ADN y permitir que la polimerasa lea una de ellas. TFIIH también tiene otra actividad enzimática importante: también es una serina quinasa que fosforila el dominio carboxilo-terminal (CTD) de la ARN polimerasa II. Hay varias serinas en el CTD, y como se fosforilan secuencialmente, el CTD se extiende como una cola (cargada negativamente) y ayuda a promover la separación entre el RNAP II y el TFIID/promotor.
La elongación de la cadena de ARN en eucariotas es muy similar a la de los procariotas con la obvia diferencia de que la transcripción ocurre en el núcleo más que en el citoplasma. Así, en procariotas, el ARN puede ser utilizado para la traducción de proteínas ¡incluso cuando todavía se está transcribiendo a partir del ADN! En los eucariotas, la situación es significativamente más compleja: hay una serie de eventos postranscripcionales (5' terminalmente, poliadenilación 3', y a menudo empalme de ARN) que deben ocurrir antes de que el ARN esté listo para ser transportado fuera del núcleo y puesto a disposición para su traducción en el citoplasma.
La terminación de la transcripción eucariota no está bien descrita en este escrito. RNAP I parece requerir un factor de terminación de unión a ADN, que no es análogo al factor Rho procariota, que es una proteína de unión a ARN. RNAP III termina la transcripción sin ningún factor externo, y esta terminación generalmente ocurre después de agregar una serie de residuos de uridina. Sin embargo, no parece utilizar la estructura de bucle de horquilla que se encuentra en la transcripción bacteriana independiente de rho-independent. La terminación de los transcritos de RNAP II que codifican proteínas está ligada a un complejo enzimático que también escinde parte del extremo 3' del ARN y agrega una cola poli-A. Sin embargo, no está claro cómo el complejo de poliadenilación está involucrado en la determinación del punto de terminación de la transcripción, que puede estar más de 1000 nucleótidos más allá del sitio poli-A (por ejemplo, el gen de β-globina en Mus musculus). Tras la terminación y liberación del RNAP II y ADN molde, el ARN se conoce como el transcrito primario, pero debe someterse a procesamiento postranscripcional antes de que sea un ARN mensajero maduro (ARNm) listo para ser exportado al citoplasma y utilizado para la traducción directa.


