8.4: Procesamiento postranscripcional de ARN
- Page ID
- 53299
El primero de los eventos postranscripcionales es la caperuza en el extremo 5'. Una vez que el extremo 5' de un ARN naciente se extiende libre del RNAP II aproximadamente 20-30 nt, está listo para ser tapado por una estructura de 7-metilguanosina. Esta “tapa” 5' sirve como sitio de reconocimiento para el transporte del ARNm completo fuera del núcleo y hacia el citoplasma.
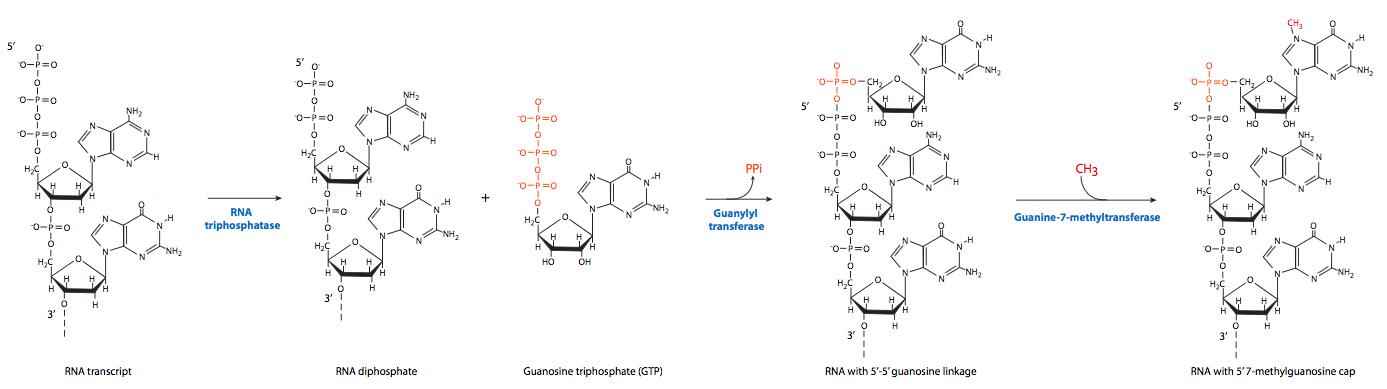
El proceso en realidad implica tres pasos. Primero, la ARN trifosfatasa elimina el grupo trifosfato 5'-terminal. La guanilación por GTP es catalizada por la enzima de remate, formando un inusual enlace 5'-5' “hacia atrás” entre la nueva guanina y el primer nucleótido del transcrito de ARN. Finalmente, la guanina-7-metiltransferasa metila la guanina recién adherida.
En el extremo opuesto del ARN, en el 3'-OH libre, se produce la poliadenilación. Como se señaló anteriormente, un complejo enzimático que se acopla a un sitio en la cola CTD de RNAP II escinde una porción del extremo 3' cerca de una secuencia de reconocimiento de AAUAAA y luego agrega en serie un gran número de residuos de adenina. La cola poli (A) no es necesaria para la traducción, pero tiene un efecto sobre la estabilidad de los transcritos en el citoplasma. A medida que las moléculas de ARNm permanecen en el citoplasma más tiempo, la cola de poli (A) se elimina gradualmente. Una vez que la cola poli (A) se haya ido, el ARNm pronto será destruido. Las moléculas de ARNm con colas poli (A) más largas generalmente tienen una vida más larga en el citoplasma que aquellas con colas más cortas, pero actualmente no hay evidencia de un efecto directamente proporcional.
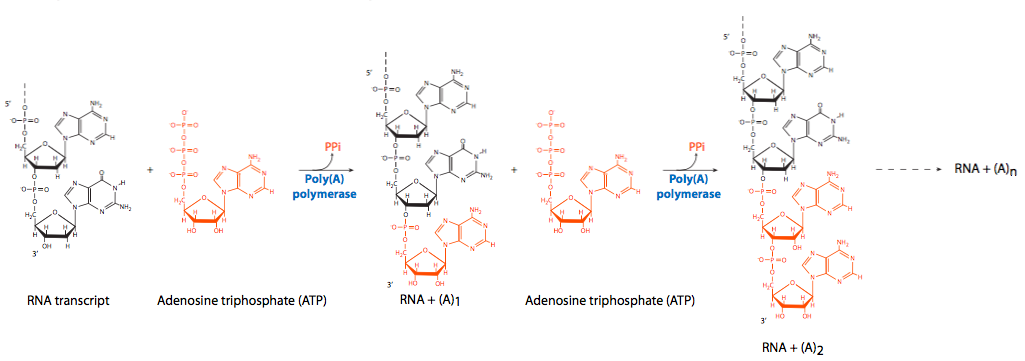
Aunque no se ha identificado la enzima que escinde el transcrito primario en preparación para la poliadenilación, se han implicado dos factores no enzimáticos, el factor de escisión I (CFI) y el factor de escisión II (CFII) con el nombre excitante. La adenilación en serie proviene de la actividad de la poli (A) polimerasa (PAP) en conjunto con CPSF (factor de ciudad especi de escisión y poliadenilación), que se une al ARN. La PAP en sí tiene una afinidad relativamente pobre por el ARN. Al igual que con otras polimerasas de ácido nucleico, agrega nuevos nucleótidos al 3'-OH libre de la cadena preexistente. Para fomentar la procesividad (polimerización continua), la proteína de unión a poli (A) II (PABII) se une al complejo de poliadenilación y participa en el control de la longitud final de la cola de poli (A). Cabe señalar que PABII es una proteína nuclear y no debe confundirse con PABP (proteína de unión a poli (A)) que se une a moléculas de ARNm en el citoplasma y juega un papel en protegerlas del ataque de nucleasas.
La tercera y más complicada modificación del ARN eucariota recién transcrito es el splicing. A diferencia del ARN procariota, que es una región codificante continuamente traducible inmediatamente cuando sale de la ARN polimerasa, la mayoría de los ARN eucariotas tienen regiones codificantes interrumpidas. El empalme es el proceso mediante el cual se eliminan las regiones no codificantes, conocidas como intrones, y las regiones codificantes, conocidas como exones, se conectan entre sí. En algunos ARN, esto puede suceder de manera autónoma, con parte del ARN actuando como catalizador enzimático para el proceso. Esto requiere que el ARN tenga una estructura secundaria y terciaria específica, acercando los dos exones mientras se enrolla el intrón. Fue el estudio de este fenómeno lo que llevó al descubrimiento de ribozimas, que son enzimas hechas de ARN.
Hasta el descubrimiento de las ribozimas, se había asumido que solo las enzimas podían generarse con la diversidad de estructuras posibles con los aminoácidos en las proteínas.
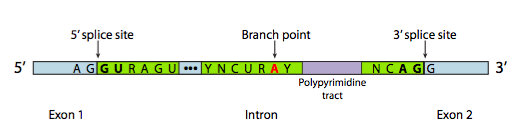
En la mayoría de los casos, sin embargo, el corte y empalme se lleva a cabo mediante un complejo proteico de múltiples subunidades conocido como spliceosoma. Ya sea autoempalmado o por spliceosoma, hay tres componentes principales de la secuencia necesarios para definir un intrón que va a ser empalmado (Figura\(\PageIndex{8}\)). Existe un sitio de empalme 5' con la secuencia consenso AG|GUAAGU. Existe un sitio de corte y empalme en 3' que inicia con un tramo de polipirimidina de 11 nucleótidos seguido de NCAG|G. Y en algún lugar entre los dos, hay una adenina de ramificación, típicamente dentro de una secuencia YNCURAY (Y es una pirimidina, N es cualquier nucleótido, R es una purina). El empalme es en realidad un conjunto de dos reacciones secuenciales de transesterificación, y requiere la proximidad física de los sitios reactivos por flexión y bucle del ARN, ya sea de forma autónoma o alrededor de factores proteicos conocidos como SNRNP (pronunciados “snurps”). SNRNP es un acrónimo de ribonucleoproteínas nucleares pequeñas. Contienen tanto una proteína como un pequeño componente de ARN nuclear (ARNsn); este último ayuda con el reconocimiento de secuencias. El examen de la estructura de la parte de ARNsn de estos snRNP de spliceosomas muestra que son muy similares a las formas tomadas por el propio transcrito de ARN en casos de autocorte y empalme. Teniendo esto en cuenta, gran parte de la siguiente descripción del empalme mediado por spliceosoma también ocurre en el autoempalme.
Aunque los snRNP son los componentes principales del spliceosoma, también juegan un papel importante una variedad de otros factores de empalme. Los más destacados son U2AF (factor asociado a U2, que se une al tracto de polipirimidina, y SF1 (factor de empalme 1, también conocido como proteína de punto de ramificación BPP) que se une a la secuencia consenso cerca del punto de ramificación. Juntos ayudan a posicionar correctamente el snRNP U2. También hay una variedad de otros factores de corte y empalme menos estudiados de la familia de proteínas SR (motivo de unión a serina-arginina C-terminal) y las familias hnRNP (proteína ribonucleo nuclear heterogénea) que actúan para reclutar a los miembros primarios del spliceosoma a sus ubicaciones adecuadas.
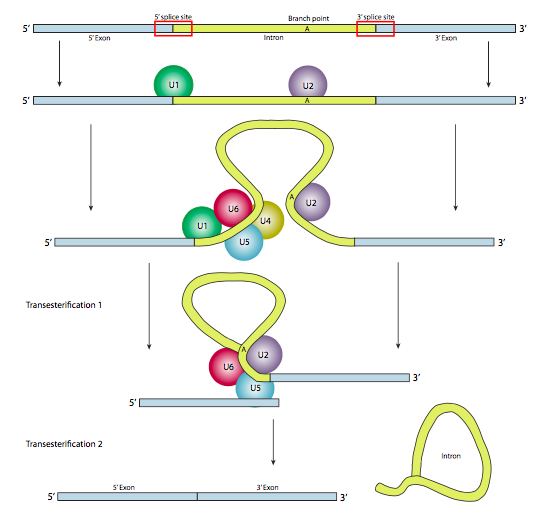
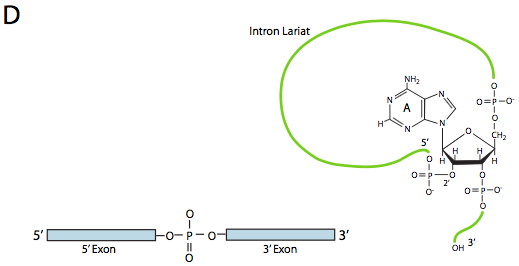
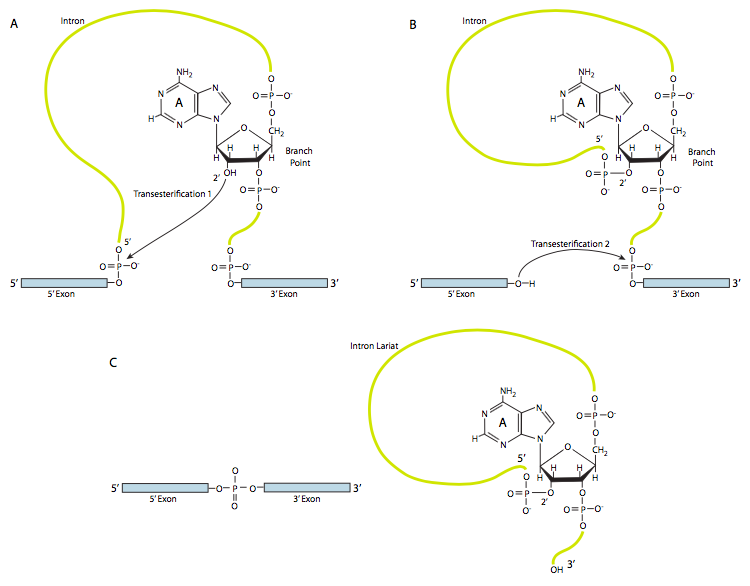
En la primera etapa, el snRNP U1 se une a la porción intrónica del sitio de empalme 5'. A continuación, el snRNP U2 se une al sitio de consenso alrededor del punto de ramificación, pero lo que es importante, no hay emparejamiento de bases con el propio punto de ramificación A. En cambio, debido al emparejamiento de bases de U2 con la secuencia circundante, el punto de ramificación A se ve obligado a sobresalir del resto del ARN en esa región. U4, U5 y U6 unen el spliceosoma juntos, pero mientras que U5 se une al exón 5', y U6 desplaza a U1 en el sitio de empalme 5', U4 solo se une transitoriamente y también se cae del spliceosoma antes de la primera reacción de transesterificación. Como muestra la Figura, en esta reacción, el 2'-OH del punto de ramificación A ataca nucleofílicamente al 5'-fosfato del nucleótido del primer intrón para formar una estructura de lariat en la que el extremo 5' del intrón está conectado al punto de ramificación a través de un enlace 2',5'-fosfodiéster. Esto libera el exón 5' (y la mitad 5' completa del ARN para el caso), pero se mantiene muy cerca del exón 3' (y el resto del ARN) por U5, que se une a ambos exones. Esto permite que tenga lugar la segunda transesterificación, en la que el 3'- OH del primer exón ataca al fosfato 5' al inicio del segundo exón, rompiendo así simultáneamente el enlace entre el intrón y el segundo exón, y conectando también los dos exones a través de un enlace 3',5'-fosfodiéster convencional . El intrón, en forma de lariat, se libera así y se degradará rápidamente.
El empalme es una forma eficiente (con respecto al tamaño del genoma) para generar diversidad proteica. En el corte y empalme alternativo, algunos intrones potenciales pueden ser empalmados bajo ciertas circunstancias pero permanecen como secuencia codificante en otras circunstancias. Recordemos que los sitios de corte y empalme son reconocidos por el emparejamiento de bases y por lo tanto, puede haber sitios de corte y empalme más fuertes y débiles dependiendo de lo cerca que estén del consenso y la secuencia complementaria en los snRNP. Por lo tanto, un gen con varios intrones potenciales puede tener todos los intrones empalmados el 80% del tiempo, pero el otro 20% del tiempo, tal vez solo uno o dos intrones son empalmados. Agregando variabilidad, hay factores de empalme que pueden unirse cerca de los sitios de empalme y pueden hacerlos más fácilmente reconocibles o casi ocultos.
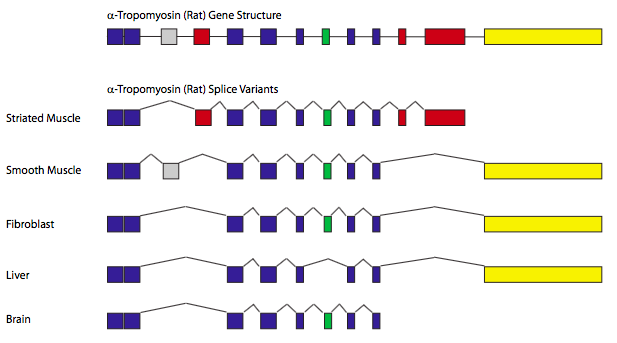
El ejemplo clásico de corte y empalme alternativo es el gen que codifica α-tropomiosina (Figura\(\PageIndex{11}\)). Mediante el corte y empalme de diferentes combinaciones de exones, un solo gen puede generar siete proteínas diferentes, dependiendo del tipo de tejido. En estos casos, tipos particulares de células o tejidos contienen combinaciones específicas de factores de corte y empalme, y por lo tanto controlan el reconocimiento de sitios de corte y empalme específicos, dando lugar a los diferentes patrones de corte y empalme.
Si bien esto concluye la discusión de los mecanismos básicos de transcripción, el siguiente capítulo es realmente una continuación de éste: el control de la expresión génica en su forma más simple es regular el reconocimiento de una secuencia promotora por una ARN polimerasa.