9.1: Regulación transcripcional procariota
- Page ID
- 52931
A diferencia de los organismos multicelulares, en los que la mayoría de las células se encuentran en un ambiente interno estrechamente regulado, la mayoría de las células procariotas responden constantemente a condiciones cambiantes en su entorno inmediato, como cambios en la concentración de sal, temperatura, acidez o disponibilidad de nutrientes. Debido a que estos organismos deben responder rápidamente, la vida útil de un ARN se mantiene corta, del orden de varios minutos, por lo que los productos génicos que no son útiles en las nuevas condiciones no desperdician recursos. Por la misma razón, el inicio de la nueva transcripción también debe ocurrir muy rápidamente, de manera que los productos génicos que se necesitan para estabilizar la célula en las nuevas condiciones estén rápidamente disponibles. Se necesita un sistema de control rápido y eficiente, y en los procariotas, esto significa que los controles sobre la transcripción son simples activadores y represores. Para algunos genes, ambos pueden ser utilizados para la regulación, mientras que para otros, solo se necesita uno para cambiar de un estado predeterminado de expresión o no expresión.
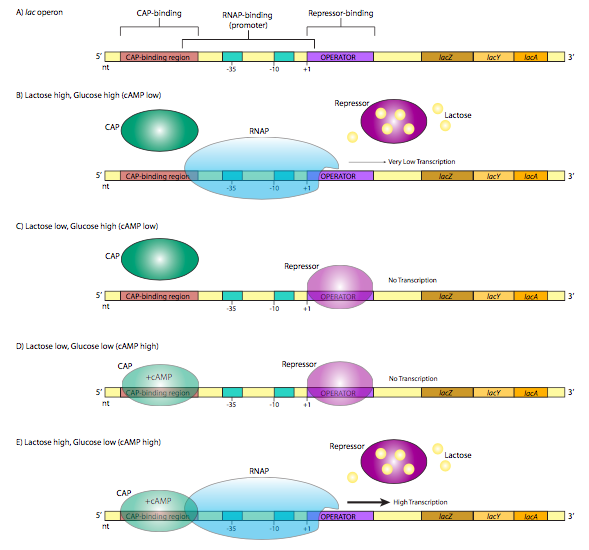
Un ejemplo clásico de control represor de la expresión génica, el operón lac, también ilustra otro método por el cual las bacterias pueden controlar la expresión de genes. Un operón es un grupo de genes cuyos productos participan en la misma vía metabólica, y se transcriben bajo el control de un solo promotor. El operón lac consta de tres genes (lacZ, lacY, lacA) que participan en el catabolismo del disacárido, la lactosa. LacZ es β-galactosidasa, una enzima que escinde la lactosa en galactosa y glucosa. LaCy es β-galactósido permeasa, que transporta lactosa desde el ambiente extracelular a la célula. Ambos son necesarios para el catabolismo de la lactosa. Curiosamente, LacA no es absolutamente necesario para el metabolismo de la lactosa, pero su función está relacionada con las otras dos: es una β-galactósido transacetilasa que transfiere grupos acetilo de acetil-CoA a lactosa. Los tres son traducidos (conservan sus codones individuales de inicio y parada para la traducción, para no confundirse con el inicio y la parada de la transcripción) a partir de una sola transcripción. De particular interés con respecto a la regulación de esta transcripción es la estructura de la región promotora. Obsérvese que además del promotor σ 70 esperado aguas arriba del sitio de inicio, hay otra secuencia de control en cada lado del sitio de inicio (Figura\(\PageIndex{1}\) A).
Tenga en cuenta que el motivo hélice-giro-hélice (HTH), que es común en las proteínas bacterianas de unión al ADN, no es lo mismo que las proteínas de unión a ADN hélice-bucle-hélice que se utilizan en muchos sistemas eucariotas. Una elaboración del motivo HTH básico, conocido como el motivo hélice alada, también se encuentra en una variedad de proteínas procariotas de unión a ADN.
El operador es una secuencia de ADN que se encuentra entre el promotor y el sitio de inicio. Es reconocido por el represor lac, una proteína de unión al ADN con un motivo hélice-giro-hélice. En ausencia de lactosa (Figura\(\PageIndex{1}\) C), el represor lac tiene una alta afinidad por la secuencia operadora y se une fuertemente, obstruyendo el sitio de inicio y formando un “obstáculo” físico para la transcripción al evitar que la ARN polimerasa avance desde el promotor. Esto tiene sentido fisiológicamente porque la célula es más eficiente metabolizando la glucosa, y si no hay lactosa alrededor, entonces es un desperdicio de recursos para elaborar enzimas que la metabolizan. No obstante, ¿y si de repente hay abundancia de lactosa en el ambiente? A medida que la lactosa se introduce en la célula, los niveles intracelulares aumentan, y ahora se necesitan enzimas para utilizar esta nueva fuente de alimento. ¡La lactosa en realidad enciende la expresión de enzimas que la metabolizarán! Específicamente, la lactosa se une a la proteína represora lac (4 sitios de unión a lactosa), lo que provoca un cambio conformacional que la libera de la secuencia operadora (Figura\(\PageIndex{1}\) B). Ahora una ARN polimerasa que se une al promotor del operón lac puede proceder a transcribir el mensaje sin obstáculos, produciendo ARN y posteriormente proteínas que se utilizan para descomponer la lactosa. Esto continúa siempre y cuando haya abundante lactosa en la célula. A medida que bajan los niveles de lactosa, las proteínas represoras ya no están unidas por la lactosa, y pueden unirse nuevamente al operador e inhibir la expresión del operón una vez más. Por ahora, ignore la proteína CAP en la Figura\(\PageIndex{1}\), y las partes D y E. Volveremos a eso. El operón lac es un ejemplo de un operón inducible, en el que el estado nativo está “apagado” y la introducción de un inductor (en este caso lactosa) unirá el represor y encenderá el operón.
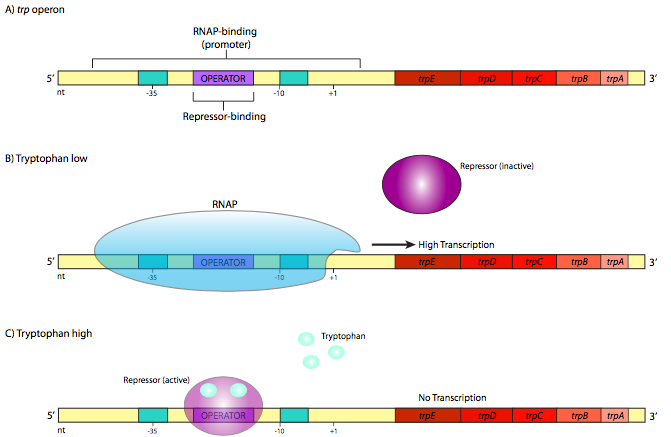
En contraste, también hay operones con el mecanismo inverso. Un ejemplo de tal operón reprimible es el operón trp (Figura\(\PageIndex{2}\)). Este operón contiene genes ve que intervienen en la síntesis del aminoácido triptófano: trpE y trpD, que en conjunto codifican las subunidades de antranilato sintetasa, trpC, que codifica N- (5'- fosforribosil) -antranilato isomerasa, y trpB y trpA, cada uno de los cuales codifica subunidades de triptófano sintetasa. El represor trp es más grande y más complejo que el represor lac, pero también utiliza un motivo de unión a ADN de hélice-vuelta-hélice.
Sin embargo, difiere en un aspecto crucial. En su forma nativa, no se une a la secuencia operadora. Solo se une al operador después de que se haya unido primero al triptófano (dos moléculas de trp se unen a un represor). Esto es lo opuesto al represor lac, pero al considerar la función fisiológica de estos genes, esto debería tener perfecto sentido. Mientras no haya triptófano, el operador está desunido, permitiendo que la ARN polimerasa transcriba los genes necesarios para producir triptófano (Figura\(\PageIndex{2}\) B). Cuando se ha acumulado suficiente triptófano en la célula, parte del triptófano “extra” se une al represor trp, lo que lo activa y permite que se una al operador (Figura\(\PageIndex{2}\) C). Cuando esto sucede, el RNAP no puede llegar al sitio de inicio, y no se desperdician recursos transcribiendo genes para enzimas que hacen algo que la célula ya tiene mucho.
Volvamos ahora al operón lac en Figura\(\PageIndex{1}\). Resulta que incluso cuando el operón es inducido por la presencia de lactosa, la tasa de transcripción es baja. La limitación no es del represor -que se ha eliminado como se ha descrito anteriormente (Figura\(\PageIndex{1}\) B). En cambio, la baja expresión se debe a un promotor de baja afinidad. Esto es cierto no solo del operón lac, sino también de otros genes del catabolismo del azúcar que no son de la vía de la glucosa. Hay una explicación simple: aunque haya abundantes azúcares alternos disponibles (por ejemplo, lactosa), si hay glucosa disponible, es la vía más eficiente y preferida de la célula para la producción de energía, y la producción de enzimas para otras vías sería un uso ineficiente de los recursos. Entonces, ¿cuándo y cómo está realmente encendido el operón lac?
La respuesta está en una CAP, proteína activadora del gen catabolito, también conocida como CRP, o proteína receptora de AMPc. Es una pequeña proteína de unión a ADN homodimérica que se une a una secuencia que se solapa con el lado 5' del promotor. En presencia de AMPc, que se une a la proteína, CAP tiene una alta afinidad por la secuencia de reconocimiento de ADN, y se une a ella (Figura\(\PageIndex{1}\) E). La proteína entonces ayuda a reclutar la RNAP al sitio promotor, uniéndose directamente al dominio C-terminal de la subunidad a de RNAP para aumentar la afinidad de la polimerasa por la secuencia promotora para superar un promotor débil.
¿Qué tiene que ver el AMPc con esto? Cuando hay abundante glucosa extracelular, hay poco AMPc. La enzima que sintetiza AMPc, la adenilato ciclasa, está regulada negativamente por el transporte de glucosa. Sin embargo, cuando hay poca glucosa ambiental, la adenilato ciclasa es más activa, produce AMPc, que se une a CAP, y conduce a una producción robusta de enzimas de catabolismo de lactosa. CAP es un ejemplo de un activador que puede controlar la expresión génica en una dirección positiva.
En E. coli, los niveles de AMPc no están directamente ligados a los niveles intracelulares de glucosa ni al metabolismo Más bien, los niveles de AMPc se ven alterados por el transporte de glucosa a través de un sistema de fosfotransferasa dependiente de fosfoenolpiruvato (PTS), parte del cual se desfosforila (el producto génico crr, también conocido como EIIA) cuando la glucosa se mueve hacia adentro. La EIIA~P fosforilada es un activador de la adenilato ciclasa. Entonces, a medida que la glucosa entra en la célula, los niveles de AMPc bajan debido a la adenilato ciclasa
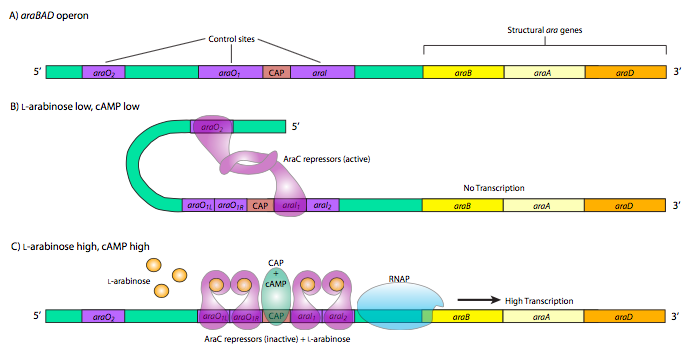
El último y más complicado ejemplo de control génico metabólico procariota es el operón araBAD. Este operón produce enzimas utilizadas para el catabolismo del azúcar de 5 carbonos, la L-arabinosa. Lo interesante de este operón es la presencia de elementos de control tanto positivos como negativos que son utilizados por la misma proteína de control, AraC. Cuando hay poca o ninguna arabinosa, la araC se une a las secuencias operadoras AraO2 y AraI1. Luego, las dos proteínas AraC interactúan, lo que hace que el ADN se mueva alrededor evitando que la ARNAP se una al promotor y transcriba araBAD. Además, este operón también está bajo el control de CAP, y la estructura de bucle AraC doble también evita que CAP se una. Sin embargo, cuando hay abundante arabinosa, los repres- sores de AraC se unen a la arabinosa y luego interactúan de manera diferente, formando dímeros, pero ahora en una conformación diferente que conduce a la unión de AraO1L y AraO1R juntos así como AraI1 y AraI2. El AraC unido a arabinosa en los sitios AraI interactúa con RNAP y junto con CAP promueven una fuerte activación de la expresión de AraBAD.
El sistema de reparación SOS
No todos los operones se ocupan de coordinar las actividades metabólicas. Un operón no metabólico importante en E. coli es el operón de respuesta Lexa/ recA SOS, que contiene genes que están involucrados en la reparación del ADN. Se invoca el sistema de reparación SOS para permitir que la replicación del ADN continúe a través de áreas de ADN dañado, pero con la penalización de baja fidelidad. Uno de los productos génicos de este operón, RecA, es importante para reconocer y reparar los daños causados por la luz UV. También funciona como regulador de la proteína represora LexA. LexA es en realidad un represor para múltiples operones SOS, que se une a una secuencia de operador común aguas arriba de cada gen/operón. Se activa cuando RecA, al detectar daños en el ADN, experimenta un cambio conformacional y activa la actividad proteasa, que luego escinde LexA, permitiendo la transcripción de los genes/operones SOS.
La reparación de SOS es propensa a errores porque cuando el replisoma encuentra daños voluminosos, sufre un “colapso de horquilla de replicación” en el que se liberan las unidades de ADN polimerasa III. Las polimerasas de reemplazo, o bypass, Pol IV (DinB) y Pol V (umuDC), no tienen actividad exonucleasa correctora 3'—5'. La mala incorporación de dímeros de timina G opuestos ocurre a aproximadamente la mitad de la velocidad de incorporación apropiada de A, y generalmente, las polimerasas de derivación son aproximadamente 1000 veces más propensas a errores que Pol II o Pol I.


