10.8: Reglamento de Traducción
- Page ID
- 53367
La expresión génica está regulada principalmente a nivel pre-transcripcional, pero también hay una serie de mecanismos para la regulación de la traducción. Un sistema animal bien estudiado es la proteína de unión a ARN sensible al hierro, que regula la expresión de genes involucrados en la regulación de los niveles intracelulares de iones de hierro. Dos de estos genes, la ferritina, que secuestra de manera segura iones de hierro dentro de las células, y la transferrina, que transporta hierro de la sangre a la célula, utilizan este sistema de regulación traslacional en un bucle de retroalimentación para responder a la concentración intracelular de hierro, pero reaccionan en caminos opuestos. La interacción clave es entre los elementos de respuesta de hierro (IRE), que son secuencias de ARNm que forman estructuras cortas tallo-bucle, e IRE-BP, la proteína que reconoce y se une a los IRE. En el caso del gen de ferritina, las secuencias IRE están situadas aguas arriba del codón de inicio. Cuando hay alto contenido de hierro, el IRE-BP es inactivo, y las estructuras tallo-bucle son fundidas e invadidas por el ribosoma, permitiendo la traducción de ferritina, que es una proteína de unión al hierro. A medida que disminuye la concentración de hierro, el IRE-BP se activa y se une alrededor de las estructuras tallo-bucle IRE, estabilizándolas e impidiendo que el ribosoma continúe. Esto evita la producción de ferritina cuando hay poco hierro que unir.
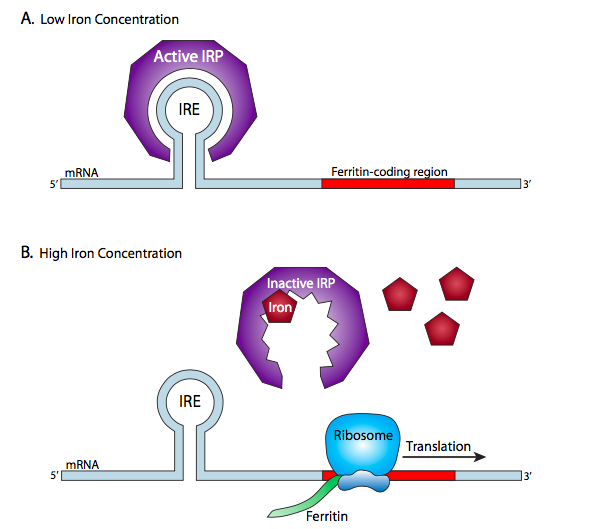
La transferrina también utiliza elementos de respuesta de hierro y proteínas de unión a IRE, pero en un mecanismo muy diferente. Las secuencias IRE del gen de la transferrina se localizan aguas abajo del codón de terminación, y no desempeñan un papel directo en permitir o prevenir la traducción.
Sin embargo, cuando hay poco hierro intracelular y se necesita más transferrina para llevar hierro a la célula, el IRE-BP se activa como en el caso anterior, y se une a los IRE para estabilizar las estructuras tallo-bucle. En este caso; sin embargo, evita la degradación de la cola poli-A 3' que normalmente ocurriría con el tiempo. Una vez que la cola poli-A se degrada, el resto del ARNm se destruye poco después. Como se menciona en el capítulo de transcripción, las colas poli-A más largas se asocian con mayor persistencia en el citoplasma, permitiendo una mayor traducción antes de que sean destruidas. El sistema IRE-BP en este caso prolonga externamente la vida útil del ARNm cuando ese producto génico es necesario en mayores cantidades.
Dado que el ARNm es un ácido nucleico monocatenario y por lo tanto capaz de unirse a una secuencia complementaria, no es demasiado sorprendente encontrar que una de las formas en que una célula puede regular la traducción es usar otra pieza de ARN. Los micro ARN (miRNAs) fueron descubiertos como genes no codificantes de proteínas muy cortos (~20 nucleótidos) en el nematodo C. elegans. Desde su descubrimiento inicial (Lee et al, Cell 75: 843-54, 1993), cientos se han encontrado en diversos eucariotas, incluyendo humanos. El patrón de expresión de los genes miARN es altamente específico para el tejido y la etapa de desarrollo. Se predice que muchos forman estructuras tallo-bucle, y parece que hibridan con secuencias 3' no traducidas de ARNm bloqueando así el inicio de la traducción en esas moléculas de ARNm. También pueden funcionar a través de un mecanismo similar al ARNip discutido a continuación, pero hay evidencia clara de que los niveles de ARNm no están necesariamente alterados por el control traduccional dirigido por miARN.
Los microARN están actualmente bajo investigación por sus papeles como oncogenes o supresores de tumores (revisado en Garzon et al, Ann. Rev. Med. 60: 167-79, 2009). Aproximadamente la mitad de los miARN humanos conocidos se encuentran en sitios frágiles, puntos de ruptura y otras regiones asociadas con cánceres (Calin et al, Proc. Nat. Acad. Sci. (USA) 101: 2999-3004, 2004). Por ejemplo, miR-21 no solo está sobrerregulada en varios tumores, su sobreexpresión bloquea la apoptosis, un paso necesario para permitir que las células anormales continúen viviendo y dividiéndose en lugar de morir. Por el contrario, miR-15a está significativamente deprimido en algunas células tumorales, y la sobreexpresión puede ralentizar o detener el ciclo celular, incluso induciendo apoptosis.
Otro mecanismo para el control traduccional que utiliza pequeñas moléculas de ARN es la interferencia de ARN (ARN i). Esto se descubrió por primera vez como una represión inducida experimentalmente de la traducción cuando se introdujeron en las células moléculas cortas de ARN bicatenario, de unos pocos cientos de nucleótidos de longitud y que contenían la misma secuencia que un ARNm diana. El efecto fue dramático: la mayor parte del ARNm con la secuencia diana se destruyó rápidamente. El modelo mecanicista actual de la represión del ARN i es que primero, las moléculas bicatenarias son escindidas por una endonucleasa llamada Dicer, que escinde con extremos 3' monocatenarios salientes. Esto permite que los fragmentos cortos (ARNip, ~20nt de largo) formen un complejo con varias proteínas (RISC, complejo silenciador inducido por ARN). El RISC divide los fragmentos bicatenarios en cadenas simples, una de las cuales es un complemento exacto del ARNm. Debido a la complementariedad, esta es una interacción estable, y la región bicatenaria parece señalar una endonucleasa para destruir el híbrido de ARNm/ ARNip.
El método final para controlar los niveles de expresión génica es el control después del hecho, es decir, mediante la destrucción dirigida de la proteína producto génico. Si bien algunas proteínas siguen funcionando hasta que se desmoronan, otras solo están pensadas para su uso a corto plazo (por ejemplo, para señalar una fase corta en el ciclo celular) y necesitan ser eliminadas para que la célula funcione correctamente. La remoción, en este sentido, sería un eufemismo para picadas y recicladas. El sistema ubiquitina-proteasoma es un mecanismo de etiquetar y destruir en el que las proteínas que han sobrevivido a su utilidad son poliubiquitinadas. La ubiquitina es una proteína eucariota pequeña (76 aminoácidos, ~5.6 kDa), altamente conservada (96% entre secuencias humanas y levaduras) (Figura\(\PageIndex{9}\)) que puede unirse a otras proteínas a través de la acción de tres etapas enzimáticas secuenciales, cada una catalizada por una enzima diferente.
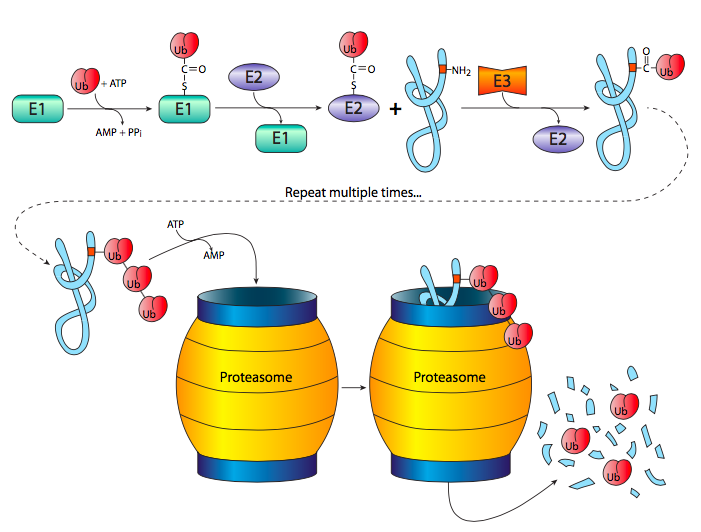
E1 activa la ubiquitina combinándola con ATP para hacer ubiquitina-adenilato, y luego transfiere la ubiquitina a sí misma a través de un enlace tioéster de cisteína. A través de una reacción de esterificación trans (tio), la ubiquitina se transfiere luego a una cisteína en la enzima E2, también conocida como enzima conjugadora de ubiquitina. Finalmente, E3, o ubiquitina ligasa, interactúa tanto con E2-ubiquitina como con la proteína designada para la destrucción, transfiriendo la ubiquitina a la proteína diana. Después de varias rondas, la proteína poliubiquitinada es enviada al proteasoma para su destrucción.
Las mutaciones en los genes E3 pueden causar una variedad de trastornos médicos humanos como los trastornos del neurodesarrollo síndrome de Angelman, síndrome de Hippel-Lindau o el trastorno general del crecimiento conocido como síndrome 3-M. Actualmente no se conocen los mecanismos que vinculan el mal funcionamiento en las vías de ubiquitinación y los síntomas de estos trastornos
Los proteasomas son complejos proteicos muy grandes dispuestos como un barril de cuatro capas (la subunidad 20S) tapado por una subunidad reguladora (19S) en cada extremo. Los dos anillos exteriores están compuestos cada uno por 7 subunidades α que funcionan como puertas de entrada a los anillos centrales, cada uno de los cuales está compuesto por 7 subunidades β, y que contienen a lo largo de la superficie interior, 6 sitios proteolíticos. Las unidades reguladoras 19S controlan la apertura y cierre de las compuertas en el barril catalítico 20S. El proteasoma completo a veces se conoce como una partícula 26S.
Una proteína poliubiquitinada se une primero a la unidad reguladora 19S en una reacción dependiente de ATP (la 19S contiene actividad ATPasa). La unidad 19S abre las puertas de la unidad 20S, posiblemente involucrando hidrólisis de ATP, y guía la proteína hacia la cámara proteolítica central. La actividad proteasa de los proteasomas es única ya que es una treonina proteasa, y corta la mayoría de las proteínas en polipéptidos regulares de 8-9 residuos, aunque esto puede variar.
Como veremos en el capítulo del ciclo celular, los proteasomas son un componente crucial para la regulación precisa de las funciones proteicas.

