13.14: Uniones Brecha
- Page ID
- 53424
A diferencia de los otros tipos de adhesión célula-célula, la unión gap (a veces llamada nexo) conecta no solo el exterior de dos células, también conecta su citoplasma. Cada célula tiene un conexón (también conocido como hemicanal) compuesto por seis proteínas conexinas. Las conexinas pueden ser todas del mismo tipo, o combinaciones de diferentes, de las cuales hay 20 conocidas en humanos y ratones. El conexón interactúa con un conexón en una célula adyacente para conectar el citoplasma de ambas células en una unión gap.
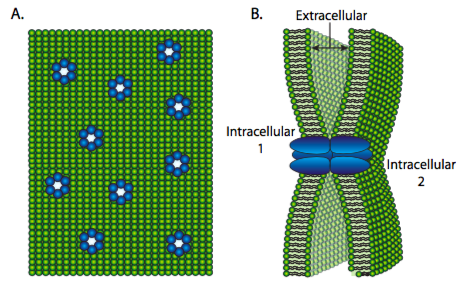
El tamaño de poro de la unión gap varía dependiendo del tipo de conexinas, pero generalmente, las moléculas menores de 1 kDa son capaces de atravesar mientras que las más grandes no pueden. Por lo tanto, las células conectadas por uniones gap están conectadas eléctricamente (los iones pueden pasar libremente), pueden compartir energía celular (ATP) y moléculas de señalización de segundos mensajeros como Ca 2+ o IP 3, pero no la mayoría de las proteínas o ácidos nucleicos. Los poros no siempre están abiertos, sino que están controlados por la fosforilación de varias serinas en los dominios intracelulares de cada conexina.
Aunque ahora se han encontrado en la mayoría de los tipos de tejido metazoico, son particularmente importantes en el músculo cardíaco. Aquí, las uniones gap aseguran una propagación eficiente de señales contráctiles para que el músculo cardíaco pueda contraerse en sincronía. También es importante en el desarrollo cardíaco: la inactivación génica de la conexina 43, la conexina cardíaca primaria, conduce a un bucle retardado de la extremidad ascendente del tubo cardíaco embrionario, lo que significa malformaciones especialmente en el ventrículo derecho, la válvula tricúspide y el tracto de salida subpulmonar.
Cuando la mayoría de las personas, incluyendo la mayoría de los biólogos, piensan en conexiones neuronales y sinapsis, piensan en sinapsis químicas en las que una célula señala a otra por liberación de neurotransmisores. Sin embargo, ahora está bien establecido que en el SNC, las sinapsis eléctricas a través de uniones gap son una parte significativa del repertorio de comunicación neuronal. La retina es un excelente ejemplo con numerosas uniones gap entre neuronas. De hecho, los neurotransmisores activados por luz pueden activar vías de proteína quinasa que fosforilan conexinas, alterando así la conductancia a través de las uniones gap. Un ejemplo llamativo es el acoplamiento eléctrico basado en uniones de hueco de neuronas fotorreceptoras de cono. Se acoplan cerca de la base de las células, de manera que la excitación de una impulsa la excitación de varias otras. Esto es importante para generar una señal visual clara porque la reacción primaria, la fototransducción, es un proceso sucio. Debido a la simple presencia de fotones aleatorios que rebotan, la relación señal/ruido de la excitación inducida por la luz es muy baja. Sin embargo, debido a que el acoplamiento eléctrico resume la señal de los vecinos cercanos pero no el ruido de fondo, ¡la salida de señal de estas neuronas tiene una mejora en la relación señal/ruido de ~ 77%! Este tema se revisa en Bloomfield y Volgyi, Nature Reviews (Neurociencia), 10:495-506, 2009.

