16.9: Reordenamiento del ADN
- Page ID
- 53227
Uno de los supuestos centrales a lo largo de nuestro estudio de la célula ha sido que aunque el ARN y las proteínas en cualquier célula pueden diferir, cualquier célula de un organismo dado que no sea los gametos debería tener el mismo ADN. Este no es el caso de las células B o las células T. En estas células, parte del proceso de maduración es crear una disposición única de diferentes dominios para formar un anticuerpo específico (o receptor de células T). El ADN de la línea germinal, o el ADN que se encuentra en todas las demás células somáticas del organismo, contiene muchos segmentos diferentes de este tipo, pero solo unos pocos se juntan para hacer el anticuerpo/TCR. Este es un proceso estocástico, y con este tipo de reordenamiento ocurriendo tanto en genes de cadena pesada como en dos genes de cadena ligera diferentes (designados κy λ), hay más de 10 billones (1013) combinaciones diferentes para generar inmunoglobulinas en humanos, ¡y aún más combinaciones para receptores de células T! ¿Cómo se logra esto?
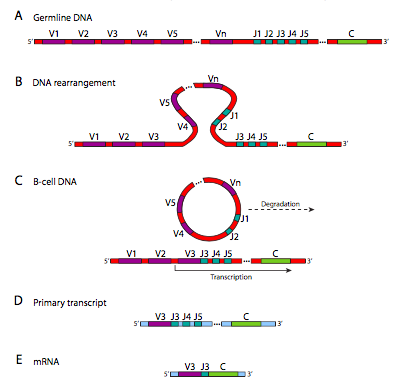
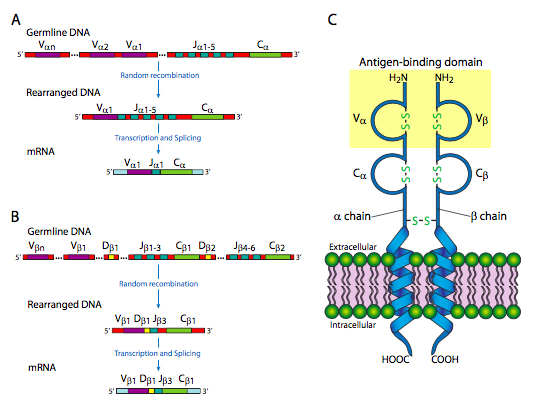
En la figura se\(\PageIndex{19}\) muestran los reordenamientos de ADN que tienen lugar en la generación de diversidad de la cadena El locus de la cadena λ tiene una disposición ligeramente diferente, y tiene solo 30 genes V, con 4 segmentos J y 4 genes C. La cadena pesada tiene un dominio extra: hay 40 genes V, los cuales están unidos a uno de 25 segmentos D, luego 6 segmentos J potenciales, y un gen C. Estos reordenamientos, aunque parecen algo así como el empalme de ARN que vimos anteriormente en este curso, están ocurriendo a nivel de ADN. Una vez que ha ocurrido, esa célula y su progenie ya no pueden hacer las otras combinaciones porque esas partes del genoma han sido cortadas y destruidas. Esto es claramente diferente del empalme alternativo del ARN, en el que la información genética aún está ahí, y bajo diferentes condiciones aún podría generar otras variaciones del producto génico.
La enzima que produce esta diversidad es un complejo llamado recombinasa V (D) J. La recombinación ocurre en dos partes: primero se realizan roturas bicatenarias en los sitios de la secuencia señal de re-combinación (RSS), luego las roturas se reparan mediante el mecanismo general de reparación de roturas bicatenarias. Dependiendo de qué segmento J se elija, puede quedar más de uno en el gen después del reordenamiento. Sin embargo, solo se usa el más cercano al segmento V, y los otros se empalman fuera del transcrito primario (por corte y empalme de ARN normal) en el proceso de conexión del segmento C con el V y J para el ARNm final. Si bien este proceso genera gran diversidad, existe otro mecanismo que puede generar mayor diversidad bajo ciertas circunstancias.
RAG1 y RAG2, genes activadores de recombinación específica de linfocitos, reconocen los sitios RSS y realizan los cortes bicatenarios. Una vez realizados los cortes, la porción extirpada une sus extremos para formar una junta circular de señal (SJ) que luego se degrada. Las porciones codificantes tienen extremos cortados asimétricos que se pliegan en formaciones de horquilla y evitan su fusión. Estas horquillas son rotas por Artemis, una nucleasa reclutada por la proteína quinasa dependiente de ADN (DNA-PK), que también reúne a XRCC4, XLF, ADN ligasa IV y una ADN polimerasa. El XRCC y XLF encienden los extremos del ADN, recluta una transferasa terminal que agrega aleatoriamente nucleótidos a los extremos, luego la ADN polimerasa λ o μ rellena los salientes y la ligasa completa la unión. Curiosamente, este proceso agrega aún más variabilidad a la inmunoglobulina, ya que Artemis corta la horquilla al azar, y la transferasa terminal también agrega nucleótidos al azar.
¡La hipermutación somática hace que los segmentos V reordenados muten a 105 veces la velocidad de otro ADN! Este mecanismo se lleva a cabo mediante la Citidina Desaminasa Inducida por Activación (AID), la cual convierte las citidinas en uracilos, generando un desapareamiento G:U que se “corrige” mediante polimerasas reparadoras sin una fuerte corrección de errores. Esta hipermutación se inicia por la activación de una célula B mediante el reconocimiento y la unión a un ligando. Como veremos en los siguientes párrafos, cuando eso suceda, la célula B inicia una proliferación rápida y es entonces cuando la hipermutación somática surte efecto, de manera que muchas de las células B portarán anticuerpos muy similares pero sutilmente diferentes a los de la célula B inicial que reconoció y fue activada por el antígeno . La idea es que algunas de estas mutaciones sutiles pueden conducir a anticuerpos con mayor afinidad por el antígeno y por lo tanto una respuesta más rápida la próxima vez que este patógeno en particular intente infectar al organismo.
La razón por la que este tipo de reordenamiento del ADN es necesario es que el “diseño” de anticuerpos no es un sistema reactivo, sino un sistema proactivo. Un error común es que el sistema inmune se encuentra con un patógeno y crea anticuerpos que lo t. Desafortunadamente, no existe un mecanismo conocido por el cual una célula pueda “sentir” la forma de algo y crear una proteína que coincida con ella. En cambio, el sistema inmune fabrica de manera preventiva tantos anticuerpos diferentes (y TCR) como sea posible, de modo que inicialmente, la mayoría de las células B y T en el cuerpo son en realidad genéticamente diferentes. Si ocurre una infección, la mayoría de las células B y T chocarán con el patógeno y rebotarán de inmediato, sin reconocerlo, pero algunas de ellas tendrán la combinación de anticuerpos adecuada para unirse a una parte del invasor patógeno. Aunque las células B pueden reconocer antígenos de superficie por sí mismas, en la mayoría de los casos, se necesita una célula T auxiliar para activar la célula B (Figura\(\PageIndex{21}\)).
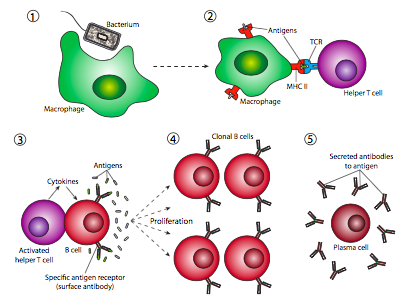
Este proceso es iniciado por un macrófago que ingiere de manera no específica un patógeno (1), lo rompe y presenta fragmentos de él en su superficie celular en asociación con el MHC (complejo mayor de histocompatibilidad). Una célula T auxiliar con un TCR que puede reconocer el antígeno presentado por el macrófago se une a él (2) y que conduce a la activación de la célula T. La célula T auxiliar activada se une y activa a una célula B (3) que también se ha unido al antígeno de interés, lo que lleva a una proliferación masiva de células B (4), proporcionando así al cuerpo muchas más copias de células que tienen el anticuerpo adecuado para localizar y eliminar la infección. Esto hace dos cosas: proporciona refuerzos de linfocitos para tratar específicamente con un patógeno en particular (pero no otras células B, Figura\(\PageIndex{22}\)), y una vez que el patógeno ha sido eliminado, existe un mayor charco circulante de estas células para responder más rápidamente a cualquier infección posterior por esta tipo particular de patógeno.
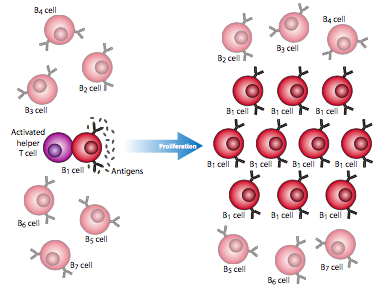
Finalmente, algunas de las células B se diferenciarán en células plasmáticas (Figura\(\PageIndex{21}\) -5), que secretan anticuerpos al torrente sanguíneo para proporcionar una respuesta humoral. Otras se convierten en células de memoria, que son como las células B en que el anticuerpo está en su superficie celular y no se secreta, pero se puede pensar en ellas como “preactivadas” y pueden responder más rápidamente que las células B ingenuas a la reinfección.
La gran pregunta que debería haber estado acechando en el fondo de tu mente a través de todo esto es, ¿cómo dicen los anticuerpos y los receptores de células T qué es lo extraño y qué forma parte del propio cuerpo? Llegaremos a eso en breve. En primer lugar, recordar la activación de la célula T colaboradora. Ocurre cuando el receptor de células T reconoce un antígeno de un patógeno ingerido que es presentado por la molécula MHC de clase II en una célula presentadora de antígeno (por ejemplo, un macrófago).
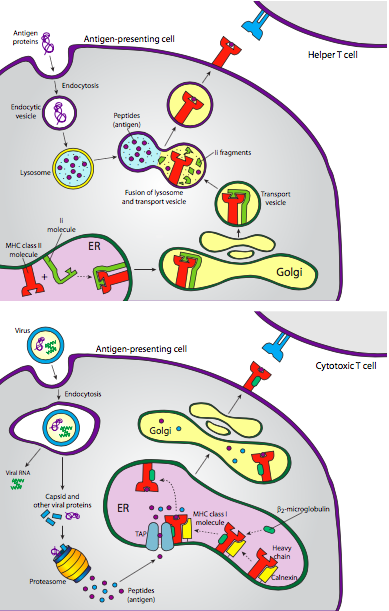
La figura\(\PageIndex{23}\) muestra la vía desde la ingestión del patógeno hasta la presentación de sus partes moleculares en una molécula MHC. Una vía similar también se aplica a la presentación de antígenos en moléculas MHC clase I junto con células T citotóxicas (asesinas). Las células T citotóxicas trabajan contra las células comprometidas, ya sean infectadas por un virus u otro patógeno (Figura\(\PageIndex{24}\)).
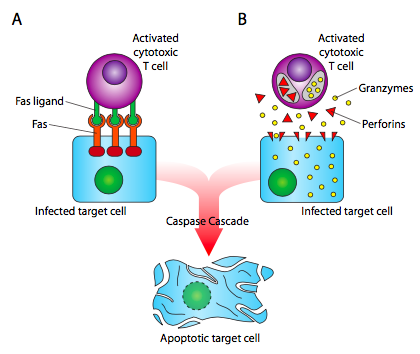
Hay dos vías principales para matar la célula infectada. Una es la activación de un “receptor de muerte”, Fas, que induce una cascada de transducción de señales para activar caspasas y apoptosis. La otra vía es la liberación de granzimas y perforinas. Las perforinas perforan la membrana de la célula diana y se convierten en poros que permiten, entre otras cosas, que las granzimas ingresen a la célula, donde activan las caspasas por proteólisis, y de nuevo inducen apoptosis. Una parte importante de esto es que el receptor de células T reconoce el antígeno en combinación con la molécula MHC que lo está presentando. Además, hay muchas variaciones de moléculas MHC debido a que hay 6 loci cada uno con muchos alelos conocidos.
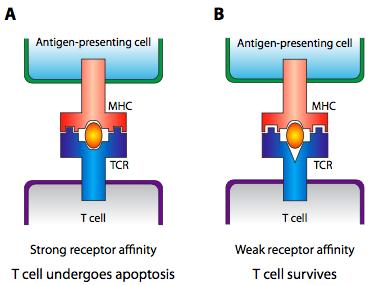
Entonces, ¿qué tiene que ver todo esto con el autoreconocimiento vs no auto reconocimiento? Al principio del desarrollo del sistema inmune, el MHC no presenta fragmentos de patógenos digeridos, presenta fragmentos de las propias células del organismo que han pasado por un proteasoma o lisosoma. En este momento temprano, las células T se comportan de manera algo diferente, y si el receptor de células T se une fuertemente, la célula T comete apoptosis. Esto elimina los genes TCR que reaccionan fuertemente a las propias células del organismo. Si las células T no reaccionan en absoluto, también se invoca la apoptosis, porque los genes TCR que no pueden reconocer el MHC no serán útiles en una respuesta inmune. Solo sobreviven aquellas células T que se unen muy débilmente al MHC autopresentante (Figura\(\PageIndex{25}\)).