20.1: Introducción
- Page ID
- 54812
La biología molecular y celular describen un sistema enormemente diverso de componentes que interactúan que es capaz de producir fenómenos intrincados y complejos. Las interacciones dentro del proteoma describen el metabolismo celular, las cascadas de señalización y la respuesta al ambiente. Las redes son una herramienta valiosa para ayudar a representar, comprender y analizar las complejas interacciones entre los componentes biológicos. Los sistemas vivos pueden verse como una composición de múltiples capas que codifican cada una información sobre el sistema. Algunas capas importantes son:
- Genoma: Incluye ADN codificante y no codificante. Los genes definidos por ADN codificante se utilizan para construir ARN, y los elementos reguladores de la CIS regulan la expresión de estos genes.
- Epigenoma: Definido por la configuración de la cromatina. La estructura de la cromatina se basa en la forma en que las histonas organizan el ADN. El ADN se divide en regiones libres de nucleosomas y nucleosomas, formando su forma final e influyendo en la expresión génica. 1
- Los ARN del transcriptoma (por ejemplo, ARNm, miARN, ncRNA, piRNA) se transcriben a partir del ADN. Tienen funciones reguladoras y fabrican proteínas.
- Proteoma Compuesto por proteínas. Esto incluye factores de transcripción, proteínas de señalización y enzimas metabólicas.
Cada capa consiste en una red de interacciones. Por ejemplo, los ARNm y los miARN interactúan para regular la producción de proteínas. Las capas también pueden interactuar entre sí, formando una red entre redes. Por ejemplo, un ARN largo no codificante llamado Xist produce cambios epigenómicos en el cromosoma X para lograr una compensación de dosis a través de la inactivación de X.
Presentamos Redes Biológicas
Cinco tipos ejemplares de redes biológicas: Red
Reguladora — conjunto de interacciones regulatorias en un organismo.
• Los nodos representan reguladores (por ejemplo, factores de transcripción) y dianas asociadas.
• Los bordes representan la interacción regulatoria, dirigida desde el factor regulatorio hasta su objetivo. Se firman según el eect positivo o negativo y se ponderan según la fuerza de la reacción.
Red Metabólica — conecta los procesos metabólicos. Hay cierta flexibilidad en la representación, pero un ejemplo es una gráfica que muestra productos metabólicos compartidos entre enzimas.
• Los nodos representan enzimas.
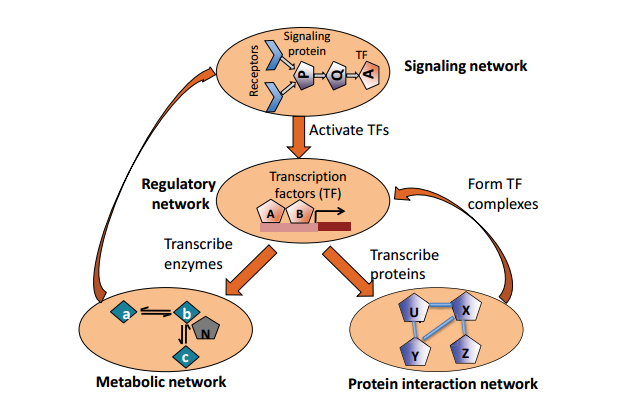
Figura 20.1: Interacciones entre redes biológicas.
• Los bordes representan reacciones reguladoras, y se ponderan de acuerdo con la fuerza de la reacción. Los bordes no están dirigidos.
Red de Señalización — representa rutas de señales biológicas.
- Los nodos representan proteínas llamadas receptores de señalización.
- Los bordes representan señales biológicas transmitidas y recibidas, dirigidas del transmisor al receptor. Los bordes están dirigidos y no ponderados.
Red de proteínas: muestra interacciones físicas entre proteínas.
- Los nodos representan proteínas individuales.
- Los bordes representan interacciones físicas entre pares de proteínas. Estos bordes son no dirigidos y no ponderados.
Red de coexpresión: describe las funciones de coexpresión entre genes. Muy general; representa redes de interacción funcionales más que físicas, a diferencia de los otros tipos de redes. Potente herramienta en el análisis computacional de datos biológicos.
• Los nodos representan genes individuales.
• Los bordes representan relaciones de coexpresión. Estos bordes son no dirigidos y no ponderados.
Hoy, nos centraremos exclusivamente en las redes regulatorias. Las redes reguladoras controlan la expresión génica específica del contexto y, por lo tanto, tienen un gran control sobre el desarrollo. Vale la pena estudiarlos porque son propensos al mal funcionamiento y están asociados con enfermedades.
Interacciones entre redes biológicas
Las redes biológicas individuales (es decir, capas) pueden considerarse nodos en una red más grande que representa todo el sistema biológico. Podemos, por ejemplo, tener una red de señalización detectando el entorno que gobierna la expresión de los factores de transcripción. En este ejemplo, la red mostraría que los TFs gobiernan la expresión de las proteínas, las proteínas pueden desempeñar papeles como enzimas en las vías metabólicas, y así sucesivamente.
Las rutas generales de intercambio de información entre estas redes se muestran en la figura 21.1a.
fuente desconocida. Todos los derechos reservados. Este contenido está excluido de nuestra licencia Creative Commons. Para obtener más información, consulte ocw.mit.edu/help/faq-fiar-use/.
Representación de la red
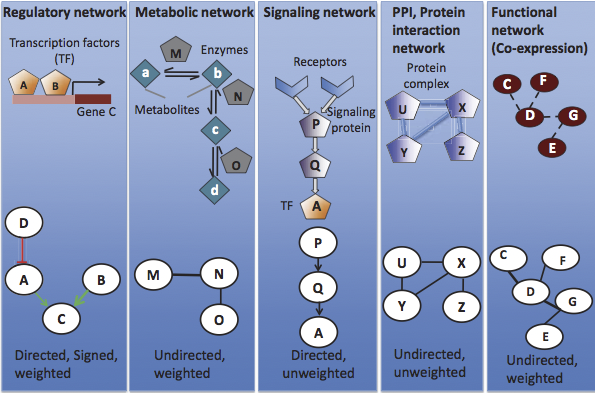
En la figura 20.2 se muestran varias de estas redes y sus visualizaciones como gráficas. Sin embargo, ¿cómo decidimos que estas redes particulares representaran los modelos biológicos subyacentes? Dado un gran conjunto de datos biológicos, ¿cómo podemos entender las dependencias entre objetos biológicos y cuál es la mejor manera de modelar estas dependencias? A continuación, presentamos varios enfoques para la representación de la red. En la práctica, ningún modelo es perfecto. La elección del modelo debe equilibrar el conocimiento biológico y la computabilidad para un análisis razonablemente eficiente.
Las redes se describen típicamente como gráficas. Las gráficas están compuestas por 1. nodos, que representan objetos; y 2. bordes, que representan conexiones o interacciones entre nodos. Hay tres formas principales de pensar sobre las redes biológicas como gráficas.
Redes probabilísticas — también conocidas como modelos gráficos. Modelan una distribución de probabilidad entre nodos.
- Modelado de la distribución conjunta de probabilidad de variables mediante gráficas.
- Algunos ejemplos son Bayesian Networks (dirigido), Markov Random Fields (Undirected). Más sobre las redes bayesianas en los capítulos posteriores.
Redes Físicas — En este esquema solemos pensar en los nodos como interactuando físicamente entre sí y los bordes capturan esa interacción.
• Los bordes representan la interacción física entre los nodos. • Ejemplo: redes regulatorias físicas.
Relevancia Red — Modele la correlación entre nodos. • Los pesos de borde representan similitudes de nodos.
• Ejemplo: redes regulatorias funcionales.
Redes como Gráficas
Los informáticos consideran subtipos de gráficos, cada uno con diferentes propiedades para sus bordes y nodos.
• Gráfica ponderada: Los bordes tienen un peso asociado. Los pesos son generalmente positivos. Cuando todos los pesos son 1, entonces lo llamamos una gráfica no ponderada.
• Gráficas dirigidas: Los bordes poseen direccionalidad. Por ejemplo A! B no es lo mismo que A B. Cuando los bordes no tienen dirección, lo llamamos una gráfica no dirigida.
• Multígrafos (pseudografías): Cuando permitimos que más de un borde vaya entre dos nodos (más de dos si está dirigido) entonces lo llamamos multígrafo. Esto puede ser útil para modelar múltiples interacciones entre dos nodos cada uno con pesos dierent por ejemplo.
• Gráfica simple: Todos los bordes no están dirigidos y no ponderados. Se prohíben múltiples aristas entre nodos y bordes propios.
Representación Matriz de Gráficas
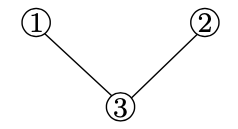
Matriz de adyacencia Una forma de representar una red es usar la llamada matriz de adyacencia. La matriz de adyacencia de una red con n nodos es una\(n \times n\) matriz A donde A ij es igual a uno si hay un borde entre los nodos i y j, y 0 en caso contrario. Por ejemplo, la matriz de adyacencia del gráfico representado en la figura 21.6b viene dada por:
\ [A=\ left [\ begin {array} {lll}
0 & 0 & 1\\
0 & 0 & 1\\
1 & 1 & 0 & 0
\ end {array}\ derecha]\]
Si la red está ponderada (es decir, si cada uno de los bordes de la red tiene un peso asociado), la definición de la matriz de adyacencia se modifica para que A ij mantenga el peso del borde entre i y j si el borde existe, y cero en caso contrario.
Otra conveniencia que viene con la representación de la matriz de adyacencia es que cuando tenemos una matriz binaria (gráfica no ponderada) entonces la suma de la fila i nos da el grado de nodo i. En una gráfica no dirigida, el grado de un nodo es el número de aristas que tiene. Ya que cada entrada en la fila nos dice si el nodo i está conectado a otro nodo, al sumar todos estos valores sabemos a cuántos nodos está conectado el nodo i, así obtenemos el grado.
1 Más en la conferencia de epigenética.


