29.4: Selección natural
- Page ID
- 53942
A mediados del siglo XIX el concepto de evolución no era una idea infrecuente, pero no fue antes de que Darwin y Wallace propusieran la selección natural como mecanismo que impulsa la evolución en la naturaleza que la teoría de la evolución obtuvo un reconocimiento generalizado. Pasaron 70 años (1948) hasta que J.B.S Haldanes Malaria Hypothesis encontró el primer ejemplo de selección natural en humanos. Mostró una correlación entre las mutaciones genéticas en los glóbulos rojos y la distribución de la prevalencia de malaria y descubrió que los individuos que tenían una mutación específica que los hacía sufrir anemia falciforme también los hacían resistentes a la malaria.
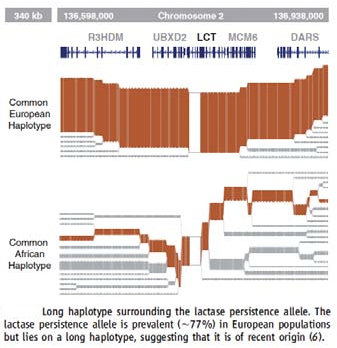
La tolerancia a la lactosa (que dura hasta la edad adulta) es otro ejemplo de selección natural. Tales ejemplos explícitos fueron difíciles de probar sin secuencias genómicas. Con la secuenciación del genoma completo fácilmente disponible, ahora podemos buscar en el genoma regiones con los mismos patrones que estos ejemplos conocidos para identificar otras regiones en proceso de selección natural.
Señales genómicas de selección natural
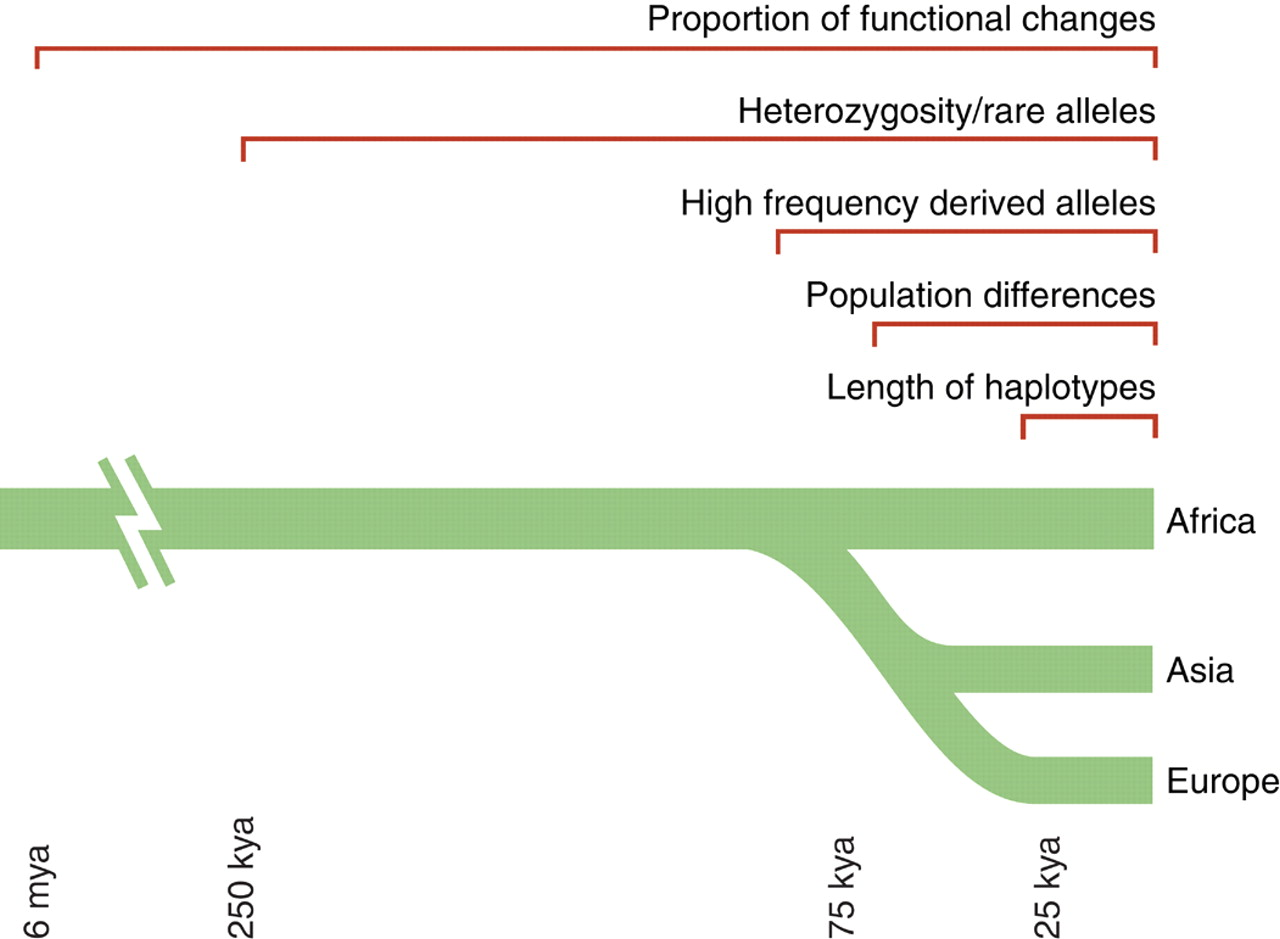
• Relación Ka/Ks de cambios no sinónimos a sinónimos por gen
• Baja diversidad y muchos alelos raros en una región (ex D de Tajima con respecto a la anemia falciforme)
• Alta frecuencia alélica derivada (o baja) sobre una región (ex Fay y H de Wu)
• Diferenciación entre poblaciones más rápida de lo esperado a partir de la deriva (Medido con Fst)
• Haplotipos largos: evidencia de barrido selectivo.
• Prevalencia exponencial de una característica en generaciones secuenciales
• Mutaciones que ayudan a que una especie prospere
Asociación Americana para el Avance de la Ciencia. Todos los derechos reservados. Este contenido está excluido
de nuestra licencia Creative Commons. Para obtener más información, consulte http://ocw.mit.edu/help/faq-fair-use/.
Fuente: Sabeti, P.C., et al. “Selección Natural Positiva en el Linaje Humano”. Ciencia 312, núm. 5780
(2006): 1614-20.
Ejemplos de Selección Negativa (Purificante)
• En todas las especies vemos selección negativa de nuevas mutaciones en elementos funcionales conservados (exones, etc.).
• Los alelos nuevos dentro de una especie tienden a tener frecuencias alélicas más bajas si el alelo no es sinónimo que sinónimo. Los alelos letales tienen frecuencias muy bajas.
Ejemplos de selección positiva (adaptativa)
- Similar a la selección negativa en esa selección positiva más probable en elementos funcionales o no-
alelos sinónimos.
- En todas las especies en un elemento conservado, una mutación seleccionada positivamente podría ser la misma sobre la mayoría de los mamíferos, pero cambio en una especie específica porque una mutación seleccionada positivamente apareció después de la especi- ción o causó especiación.
- Dentro de una especie, los alelos seleccionados de manera positiva probablemente difieren en la frecuencia alélica (F st) entre las poblaciones. Los ejemplos incluyen la resistencia a la malaria en poblaciones africanas (29.6) y la persistencia de lactosa en poblaciones europeas (29.7).
- La selección poligénica dentro de las especies puede surgir cuando se selecciona un rasgo que depende de muchos genes. Un ejemplo es la altura humana donde se sabe que 139 SNP están relacionados con la altura. La mayoría no son mutaciones específicas de la población, sino alelos en todos los humanos que se seleccionan en algunas poblaciones más que en otras. (29.8)
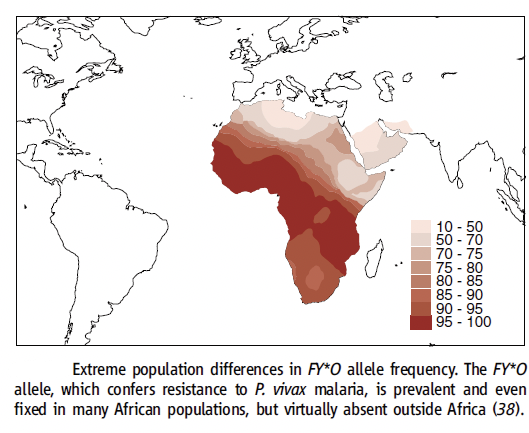
Figura 29.6: Selección positiva localizada para resistencia a la malaria dentro de las especies Sabeti et al. Ciencia 2006
Pruebas estadísticas
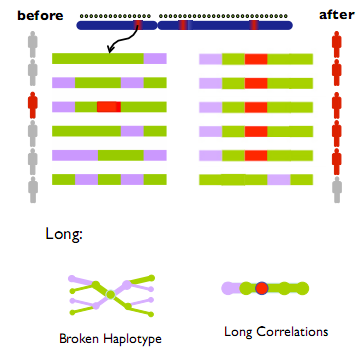
- Correlaciones de largo alcance (IHs, Xp, EHH): Si etiquetamos secuencias genéticas en un individuo en función de su ascendencia, terminamos con un haplotipo roto, donde el número de roturas (cambios de color) se correlaciona con el número de recombinaciones y puede decirnos cuánto tiempo hace que estuvo una ascendencia particular introducido.
- BARREO Un programa desarrollado por Pardis Sabeti, Ben Fry y Patrick Varilly. SWEEP detecta evidencia de selección natural mediante el análisis de estructuras de haplotipos en el genoma usando el rango largo
de nuestra licencia Creative Commons. Para obtener más información, consulte http://ocw.mit.edu/help/faq-fair-use/.
Fuente: Sabeti, P.C., et al. “Selección Natural Positiva en el Linaje Humano”. Ciencia 312, núm. 5780
(2006): 1614-20.
Figura 29.7: Selección positiva localizada para el alelo de persistencia de lactasa Sabeti et al. Ciencia 2006
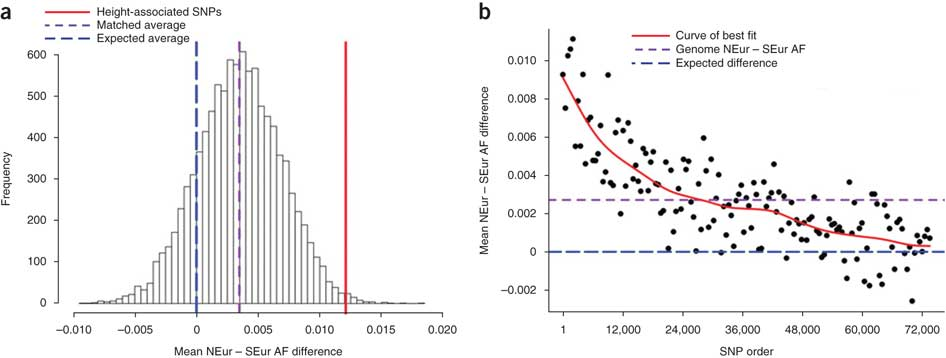
Fuente: Turchin, Michael C., et al. “Evidencia de selección generalizada sobre variación de pie
en Europa en SNPs asociados a la Altura”. Nature Genetics 44, núm. 9 (2012): 1015-9.
Figura 29.8: Diferencia de frecuencia alélica media de SNP de altura, SNPS emparejados y SNPS de todo el genoma entre poblaciones de Europa del Norte y del Sur
Turchin et al., Nature Genetics (2012)
prueba de haplotipo (LRH). Busca alelos de alta frecuencia con desequilibrio de ligamiento de largo alcance que insinúa una proliferación a gran escala de un haplotipo que ocurrió a una velocidad mayor que la recombinación podría romperlo de sus marcadores.
• Alelos Derivados de Alta Frecuencia Busque picos grandes en la frecuencia de alelos derivados en posiciones establecidas.
• Alta Diferenciación (F st) Grandes picos en diferenciación en ciertas posiciones.
Mediante estas pruebas, podemos encontrar regiones genómicas bajo presión selectiva. Un problema es que un solo SNP bajo selección positiva permitirá que los SNP cercanos viajen y viajen. Es difícil distinguir el SNP bajo selección de sus vecinos con una sola prueba. Bajo selección, todas las pruebas son fuertemente
correlacionados; sin embargo, en ausencia de selección son generalmente independientes. Por lo tanto, al emplear un estadístico compuesto construido a partir de todas estas pruebas, es posible aislar el SNP individual bajo selección.
Ejemplos en los que un solo SNP ha sido implicado en un rasgo:
• Pigmentación de la piel Chr15 en el norte de Europa
• Rasgos de cabello ChR2 en Asia
• Chr10 Rasgo desconocido en Asia
• Rasgo desconocido Chr12 en África

