9.2: Replicación de ADN
- Page ID
- 54020
Cuando una célula se divide, es importante que cada célula hija reciba una copia idéntica del ADN. Esto se logra mediante el proceso de replicación del ADN. La replicación del ADN ocurre durante la fase de síntesis, o fase S, del ciclo celular, antes de que la célula entre en mitosis o meiosis.
La elucidación de la estructura de la doble hélice proporcionó una pista sobre cómo se copia el ADN. Recordemos que los nucleótidos de adenina se emparejan con nucleótidos de timina, y citosina con guanina. Esto significa que las dos hebras son complementarias entre sí. Por ejemplo, una cadena de ADN con una secuencia de nucleótidos de AGTCATGA tendrá una cadena complementaria con la secuencia TCAGTACT (Figura\(\PageIndex{1}\)).
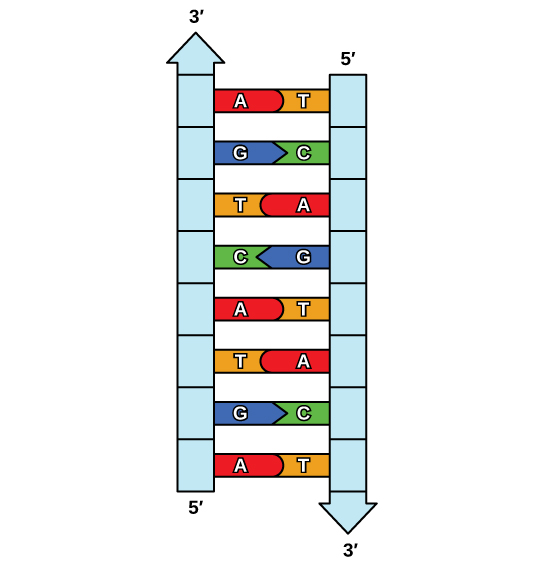
Debido a la complementariedad de las dos hebras, tener una hebra significa que es posible recrear la otra hebra. Este modelo de replicación sugiere que las dos cadenas de la doble hélice se separan durante la replicación, y cada hebra sirve como plantilla a partir de la cual se copia la nueva cadena complementaria (Figura\(\PageIndex{2}\)).
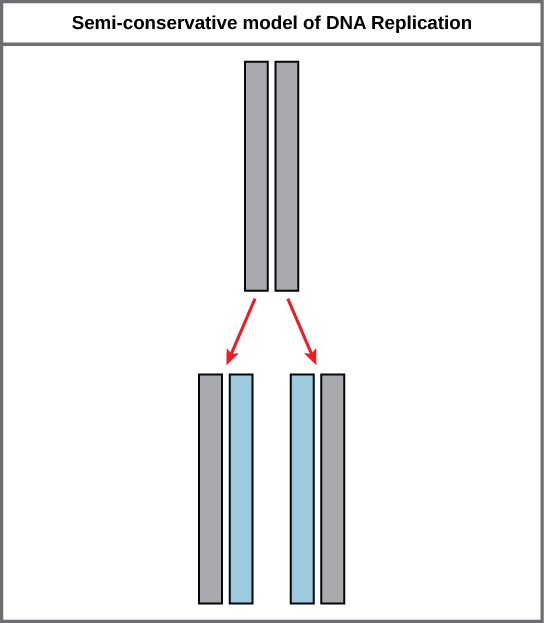
Durante la replicación del ADN, cada una de las dos hebras que componen la doble hélice sirve como plantilla a partir de la cual se copian nuevas hebras. La nueva hebra será complementaria a la hebra parental o “vieja”. Cada nueva hebra doble consiste en una hebra parental y una nueva hebra hija. Esto se conoce como replicación semiconservativa. Cuando se forman dos copias de ADN, tienen una secuencia idéntica de bases nucleotídicas y se dividen por igual en dos células hijas.
Replicación de ADN en eucariotas
Debido a que los genomas eucariotas son muy complejos, la replicación del ADN es un proceso muy complicado que involucra varias enzimas y otras proteínas. Ocurre en tres etapas principales: iniciación, elongación y terminación.
Recordemos que el ADN eucariota está unido a proteínas conocidas como histonas para formar estructuras llamadas nucleosomas. Durante la iniciación, el ADN se hace accesible a las proteínas y enzimas involucradas en el proceso de replicación. ¿Cómo sabe la maquinaria de replicación en qué parte de la doble hélice de ADN comenzar? Resulta que existen secuencias nucleotídicas específicas llamadas orígenes de replicación en las que comienza la replicación. Ciertas proteínas se unen al origen de la replicación mientras una enzima llamada helicasa se desenrolla y abre la hélice del ADN. A medida que el ADN se abre, se forman estructuras en forma de Y llamadas horquillas de replicación (Figura\(\PageIndex{3}\)). Dos bifurcación de replicación se forman en el origen de la replicación, y éstas se extienden en ambas direcciones a medida que avanza la replicación. Existen múltiples orígenes de replicación en el cromosoma eucariota, de tal manera que la replicación puede ocurrir simultáneamente desde varios lugares del genoma.
Durante la elongación, una enzima llamada ADN polimerasa agrega nucleótidos de ADN al extremo 3' del molde. Debido a que la ADN polimerasa solo puede agregar nuevos nucleótidos al final de una cadena principal, una secuencia de cebador, que proporciona este punto de partida, se agrega con nucleótidos de ARN complementarios. Este cebador se elimina posteriormente, y los nucleótidos se reemplazan con nucleótidos de ADN. Una hebra, que es complementaria a la cadena de ADN parental, se sintetiza continuamente hacia la horquilla de replicación para que la polimerasa pueda agregar nucleótidos en esta dirección. Esta cadena sintetizada continuamente se conoce como la cadena principal. Debido a que la ADN polimerasa solo puede sintetizar ADN en una dirección de 5' a 3', la otra nueva cadena se junta en trozos cortos llamados fragmentos de Okazaki. Los fragmentos de Okazaki requieren cada uno de un cebador hecho de ARN para iniciar la síntesis. La hebra con los fragmentos de Okazaki se conoce como la hebra rezagada. A medida que avanza la síntesis, una enzima elimina el cebador de ARN, que luego se reemplaza con nucleótidos de ADN, y los huecos entre los fragmentos son sellados por una enzima llamada ADN ligasa.
El proceso de replicación del ADN se puede resumir de la siguiente manera:
- El ADN se desenrolla en el origen de la replicación.
- Se añaden nuevas bases a las hebras parentales complementarias. Un nuevo hilo se hace de forma continua, mientras que el otro hilo se hace en piezas.
- Se eliminan los cebadores, se ponen nuevos nucleótidos de ADN en lugar de los cebadores y la cadena principal se sella mediante ADN ligasa.
CONEXIÓN ART
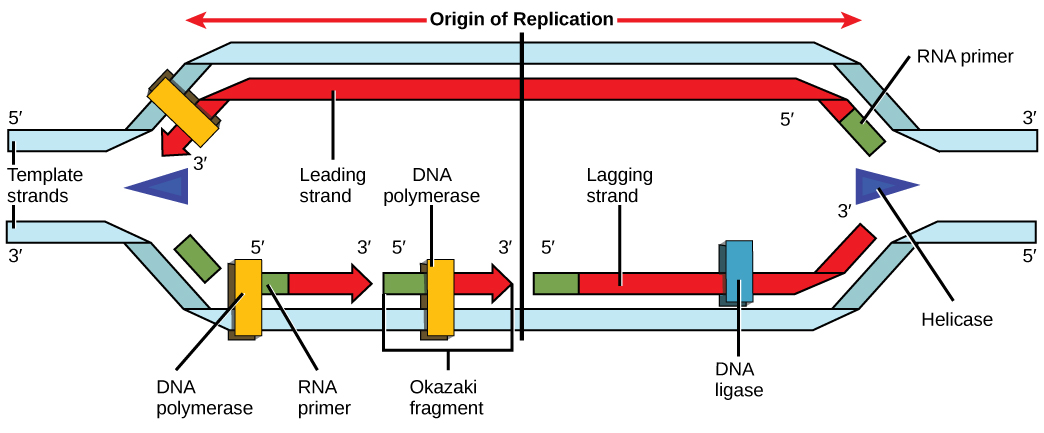
Se aísla una cepa celular en la que se altera la unión de los fragmentos de Okazaki y se sospecha que se ha producido una mutación en una enzima que se encuentra en la horquilla de replicación. ¿Qué enzima es más probable que mute?
Replicación de Telómeros
Debido a que los cromosomas eucariotas son lineales, la replicación del ADN llega al final de una línea en los cromosomas eucariotas. Como ya has aprendido, la enzima ADN polimerasa puede agregar nucleótidos en una sola dirección. En la cadena principal, la síntesis continúa hasta que se alcanza el final del cromosoma; sin embargo, en la hebra rezagada no hay lugar para que se haga un cebador para que el fragmento de ADN se copie al final del cromosoma. Esto presenta un problema para la celda porque los extremos permanecen desapareados, y con el tiempo estos extremos se acortan progresivamente a medida que las celdas continúan dividiéndose. Los extremos de los cromosomas lineales se conocen como telómeros, los cuales tienen secuencias repetitivas que no codifican para un gen en particular. Como consecuencia, son los telómeros los que se acortan con cada ronda de replicación del ADN en lugar de los genes. Por ejemplo, en humanos, una secuencia de seis pares de bases, TTAGGG, se repite de 100 a 1000 veces. El descubrimiento de la enzima telomerasa (Figura\(\PageIndex{4}\)) ayudó en la comprensión de cómo se mantienen los extremos cromosómicos. La telomerasa se une al extremo del cromosoma y se agregan bases complementarias al molde de ARN en el extremo de la cadena de ADN. Una vez que la plantilla de la hebra retrasada es suficientemente alargada, la ADN polimerasa ahora puede agregar nucleótidos que son complementarios a los extremos de los cromosomas. Así, los extremos de los cromosomas se replican.
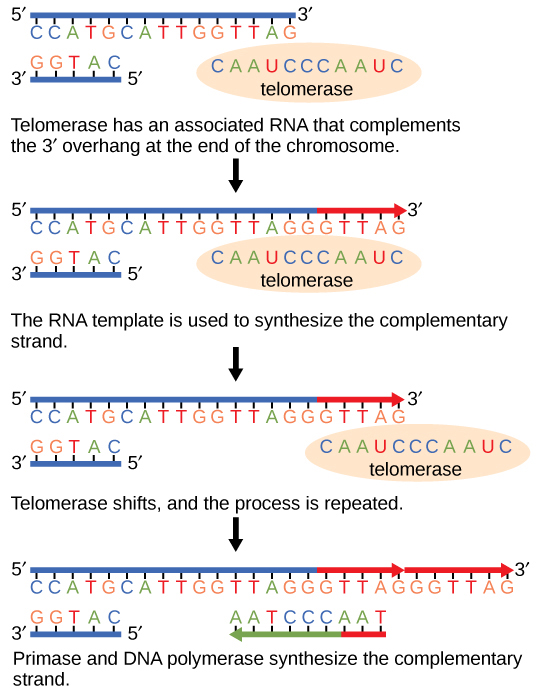
Normalmente se encuentra que la telomerasa es activa en las células germinales, las células madre adultas y algunas células cancerosas. Por su descubrimiento de la telomerasa y su acción, Elizabeth Blackburn (Figura\(\PageIndex{5}\)) recibió el Premio Nobel de Medicina y Fisiología en 2009.

La telomerasa no es activa en las células somáticas adultas. Las células somáticas adultas que se someten a división celular continúan teniendo sus telómeros acortados. Esto esencialmente significa que el acortamiento de los telómeros está asociado con el envejecimiento. En 2010, los científicos encontraron que la telomerasa puede revertir algunas afecciones relacionadas con la edad en ratones, y esto puede tener potencial en la medicina regenerativa. 1 En estos estudios se utilizaron ratones deficientes en telomerasa; estos ratones presentan atrofia tisular, agotamiento de células madres, falla del sistema de órganos y respuestas deterioradas a lesiones tisulares. La reactivación de la telomerasa en estos ratones causó extensión de telómeros, reducción del daño en el ADN, neurodegeneración inversa y mejora del funcionamiento de los testículos, bazo e intestinos. Por lo tanto, la reactivación de los telómeros puede tener potencial para tratar enfermedades relacionadas con la edad en humanos.
Replicación de ADN en Procariotas
Recordemos que el cromosoma procariota es una molécula circular con una estructura enrollada menos extensa que los cromosomas eucariotas. El cromosoma eucariota es lineal y altamente enrollado alrededor de las proteínas. Si bien hay muchas similitudes en el proceso de replicación del ADN, estas diferencias estructurales requieren algunas diferencias en el proceso de replicación del ADN en estas dos formas de vida.
La replicación del ADN ha sido muy bien estudiada en procariotas, principalmente por el pequeño tamaño del genoma y el gran número de variantes disponibles. Escherichia coli tiene 4.6 millones de pares de bases en un solo cromosoma circular, y todo se replica en aproximadamente 42 minutos, comenzando desde un único origen de replicación y avanzando alrededor del cromosoma en ambas direcciones. Esto significa que se agregan aproximadamente 1000 nucleótidos por segundo. El proceso es mucho más rápido que en eucariotas. La tabla\(\PageIndex{1}\) resume las diferencias entre replicaciones procariotas y eucariotas.
| Propiedad | Procariotas | Eucariotas |
|---|---|---|
| Origen de replicación | Sencillo | Múltiple |
| Tasa de replicación | 1000 nucleótido/s | 50 a 100 nucleótido/s |
| Estructura cromosómica | circular | lineal |
| Telomerasa | No presente | Presente |
CONCEPT EN ACCIÓN
Haga clic en un tutorial sobre la replicación del ADN.
Reparación de ADN
La ADN polimerasa puede cometer errores al agregar nucleótidos. Edita el ADN mediante la corrección de cada base recién agregada. Las bases incorrectas se eliminan y reemplazan por la base correcta, y luego continúa la polimerización (Figura\(\PageIndex{6}\) a). La mayoría de los errores se corrigen durante la replicación, aunque cuando esto no sucede, se emplea el mecanismo de reparación de desajustes. Las enzimas reparadoras de desapareamientos reconocen la base mal incorporada y la extirpan del ADN, reemplazándola por la base correcta (Figura\(\PageIndex{6}\) b). En otro tipo de reparación, la reparación por escisión de nucleótidos, la doble cadena de ADN se desenrolla y separa, se eliminan las bases incorrectas junto con algunas bases en el extremo 5' y 3', y estas se reemplazan copiando el molde con la ayuda de la ADN polimerasa (Figura\(\PageIndex{6}\) c). La reparación por escisión de nucleótidos es particularmente importante en la corrección de dímeros de timina, los cuales son causados principalmente por la luz ultravioleta. En un dímero de timina, dos nucleótidos de timina adyacentes entre sí en una cadena están unidos covalentemente entre sí en lugar de sus bases complementarias. Si el dímero no es removido y reparado conducirá a una mutación. Los individuos con defectos en sus genes de reparación de escisión de nucleótidos muestran extrema sensibilidad a la luz solar y desarrollan cánceres de piel temprano en la vida.
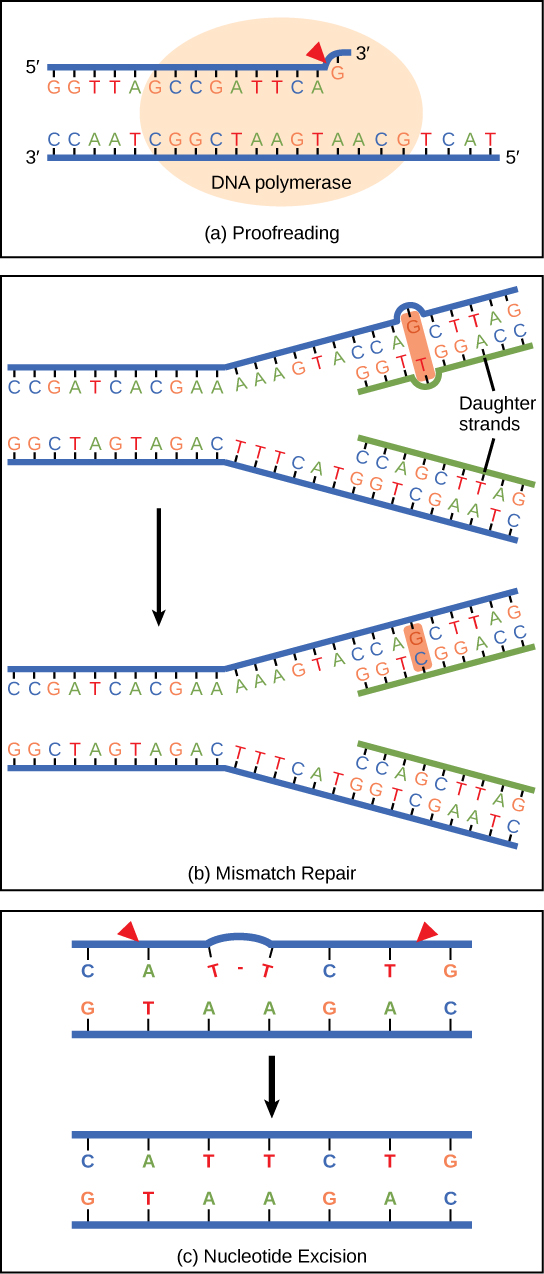
La mayoría de los errores se corrigen; si no lo son, pueden resultar en una mutación, definida como un cambio permanente en la secuencia de ADN. Las mutaciones en los genes reparadores pueden llevar a consecuencias graves como el cáncer.
Resumen
El ADN se replica por un método semi-conservador en el que cada una de las dos cadenas de ADN parentales actúan como molde para que se sintetice nuevo ADN. Después de la replicación, cada ADN tiene una hebra parental o “vieja”, y una hebra hija o “nueva”.
La replicación en eucariotas comienza en múltiples orígenes de replicación, mientras que la replicación en procariotas parte de un único origen de replicación. El ADN se abre con enzimas, dando como resultado la formación de la horquilla de replicación. La primasa sintetiza un cebador de ARN para iniciar la síntesis por la ADN polimerasa, que puede agregar nucleótidos en una sola dirección. Una hebra se sintetiza continuamente en la dirección de la bifurcación de replicación; esto se llama la hebra principal. La otra hebra se sintetiza en una dirección alejada de la horquilla de replicación, en tramos cortos de ADN conocidos como fragmentos de Okazaki. Esta hebra se conoce como la hebra rezagada. Una vez completada la replicación, los cebadores de ARN se reemplazan por nucleótidos de ADN y el ADN se sella con ADN ligasa.
Los extremos de los cromosomas eucariotas plantean un problema, ya que la polimerasa es incapaz de extenderlos sin un cebador. La telomerasa, una enzima con un molde de ARN incorporado, extiende los extremos copiando el molde de ARN y extendiendo un extremo del cromosoma. La ADN polimerasa puede entonces extender el ADN usando el cebador. De esta manera, se protegen los extremos de los cromosomas. Las células tienen mecanismos para reparar el ADN cuando se daña o se cometen errores en la replicación. Estos mecanismos incluyen la reparación de desapareamientos para reemplazar los nucleótidos que se emparejan con una base no complementaria y la reparación por escisión de nucleótidos, que elimina las bases que están dañadas como los dímeros de timina.
Conexiones de arte
Figura\(\PageIndex{3}\): Se aísla una cepa celular en la que se altera la unión de fragmentos de Okazaki y se sospecha que se ha producido una mutación en una enzima encontrada en la horquilla de replicación. ¿Qué enzima es más probable que mute?
- Contestar
-
Ligasa, ya que esta enzima une los fragmentos de Okazaki.
Notas al pie
- 1 Mariella Jaskelioff, et al., “La reactivación de la telomerasa revierte la degeneración tisular en ratones envejecidos deficientes en telomerasa”, Nature, 469 (2011) :102—7.
Glosario
- ADN ligasa
- la enzima que cataliza la unión de fragmentos de ADN
- ADN polimerasa
- una enzima que sintetiza una nueva cadena de ADN complementaria a una cadena molde
- helicasa
- una enzima que ayuda a abrir la hélice del ADN durante la replicación del ADN al romper los enlaces de hidrógeno
- hebra rezagada
- durante la replicación de la cadena 3' a 5', la cadena que se replica en fragmentos cortos y lejos de la horquilla de replicación
- hebra principal
- la cadena que se sintetiza continuamente en la dirección 5' a 3' que se sintetiza en la dirección de la bifurcación de replicación
- reparación de desajuste
- una forma de reparación del ADN en la que los nucleótidos no complementarios son reconocidos, escindidos y reemplazados con nucleótidos correctos
- mutación
- una variación permanente en la secuencia de nucleótidos de un genoma
- reparación por escisión de nucleótidos
- una forma de reparación del ADN en la que la molécula de ADN se desenrolla y se separa en la región del daño nucleotídico, los nucleótidos dañados se eliminan y se reemplazan por nuevos nucleótidos usando la cadena complementaria, y la cadena de ADN se vuelve a sellar y se permite que vuelva a unirse a su complemento
- Fragmentos de Okazaki
- los fragmentos de ADN que se sintetizan en tramos cortos en la hebra retrasada
- imprimación
- un tramo corto de nucleótidos de ARN que se requiere para iniciar la replicación y permitir que la ADN polimerasa se una y comience la replicación
- bifurcación de replicación
- la estructura en forma de Y formada durante el inicio de la replicación
- replicación semiconservativa
- el método utilizado para replicar ADN en el que se separa la molécula bicatenaria y cada hebra actúa como molde para una nueva cadena a sintetizar, por lo que las moléculas de ADN resultantes están compuestas por una nueva cadena de nucleótidos y una antigua cadena de nucleótidos
- telomerasa
- una enzima que contiene una parte catalítica y un molde de ARN incorporado; funciona para mantener los telómeros en los extremos cromosómicos
- telómero
- el ADN al final de los cromosomas lineales


