7.2: Genes y Genomas
- Page ID
- 53024
Fuente: BiochemFFA_7_1.pdf. Todo el libro de texto está disponible gratuitamente de los autores en http://biochem.science.oregonstate.edu/content/biochemistry-free-and-easy

Introducción
Durante muchos años, los científicos se preguntaron sobre la naturaleza de la información que dirigía las actividades de las células. ¿Qué tipo de moléculas transportaban la información y cómo se transmitía la información de una generación a otra? Experimentos clave, realizados entre los años 20 y 50, establecieron de manera convincente que esta información genética era transportada por el ADN. En 1953, con la elucidación de la estructura del ADN, se pudo comenzar a investigar cómo se transmite esta información, y cómo se usa.
Genomas
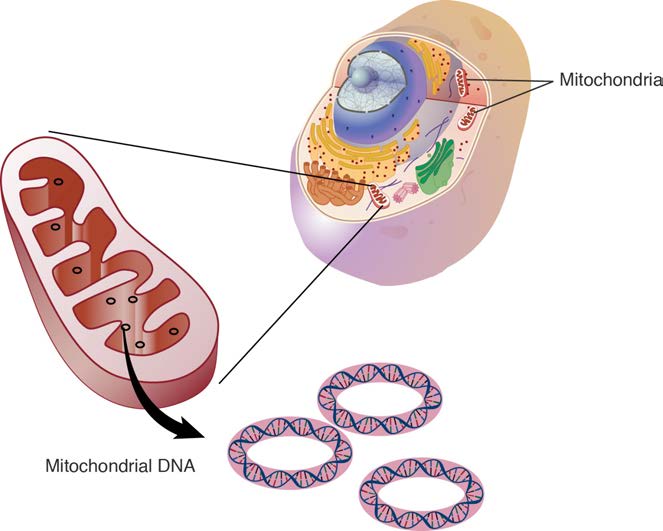
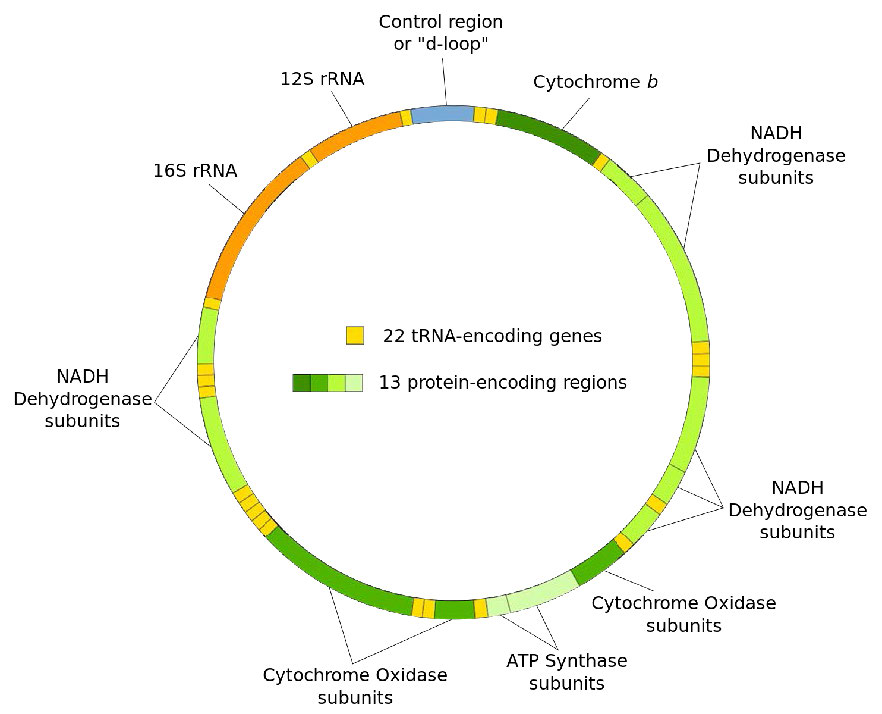
Usamos la palabra “genoma” para describir todo el material genético de la célula. Es decir, un genoma es la secuencia completa de nucleótidos en el ADN que está en todos los cromosomas de una célula. Cuando usamos el término genoma sin mayor calificación, generalmente nos referimos a los cromosomas en el núcleo de una célula eucariota. Como sabes, las células eucariotas tienen orgánulos como mitocondrias y cloroplastos que tienen su propio ADN (Figura 7.1 y 7.2). Estos son referidos como los genomas mitocondriales o cloroplastos para distinguirlos del genoma nuclear.
A partir de la década de 1980, los científicos comenzaron a determinar la secuencia completa de los genomas de muchos organismos, con la esperanza de comprender mejor cómo la secuencia de ADN especifica las funciones celulares. Hoy en día, las secuencias completas del genoma se han determinado para miles de especies de todos los dominios de la vida, y muchas más están en proceso de ser elaboradas por grupos de científicos de todo el mundo.
Iniciativa del genoma global
La Global Genome Initiative, un esfuerzo colaborativo para secuenciar al menos una especie de cada una de las 9.500 familias de invertebrados, vertebrados y plantas descritas, es una de las muchas empresas de este tipo. La información de estos diversos esfuerzos se recoge en enormes repositorios en línea, de manera que está disponible gratuitamente para los científicos. A medida que las bases de datos de secuencias compilan cada vez más información, han surgido los campos de la biología computacional y la bioinformática, para analizar y organizar los datos de una manera que ayude a los biólogos a comprender lo que significa la información en el ADN en el contexto celular.
Genes
Se sabe desde hace muchos años que los rasgos fenotípicos están controlados por regiones específicas del ADN que se denominaron “genes”. Así, el ADN se imaginó como una larga cadena de nucleótidos, en la que ciertas regiones, los genes, estaban separados por regiones no codificantes que simplemente se referían como secuencias intergénicas (inter=entre; genic=de genes). Los primeros experimentos en biología molecular sugirieron una relación simple entre la secuencia de ADN de un gen y su producto, y llevaron a los científicos a creer que cada gen portaba la información de una sola proteína. Los cambios, o mutaciones en la secuencia de bases de un gen se reflejarían en cambios en el producto génico, que a su vez, se manifestarían en el fenotipo o rasgo observable. Este cuadro sencillo, aunque sigue siendo útil, ha sido modificado por descubrimientos posteriores que demostraron que el uso de información genética por parte de las células es algo más complicado. Nuestra definición de gen también está evolucionando para tomar en consideración nuevos conocimientos.
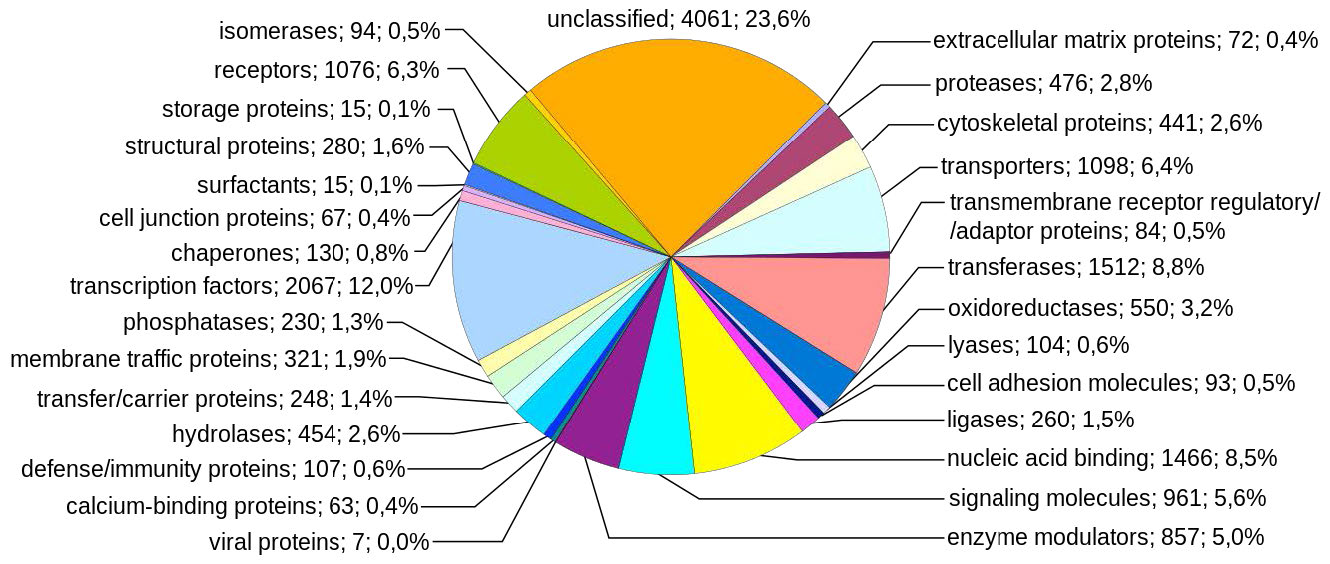
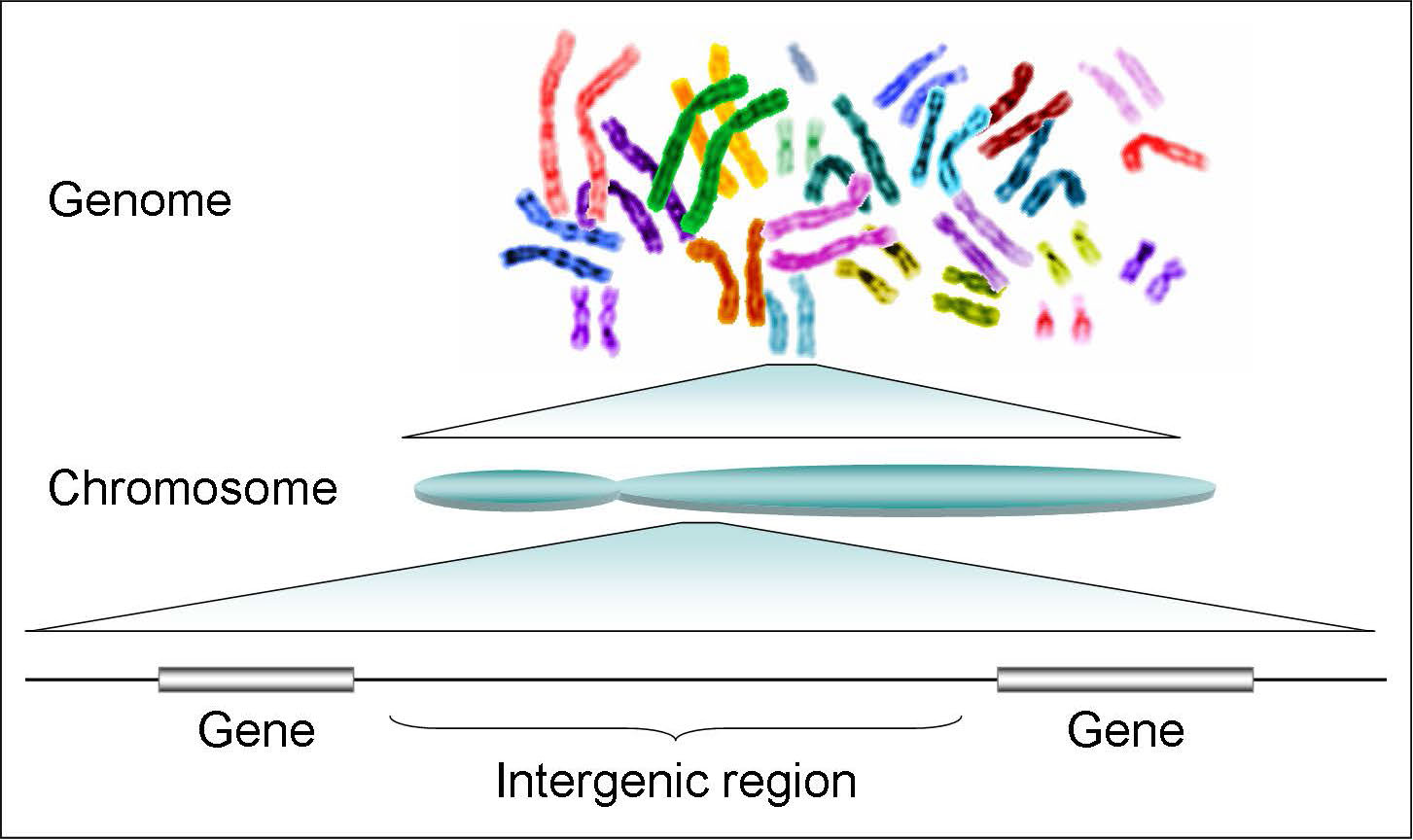
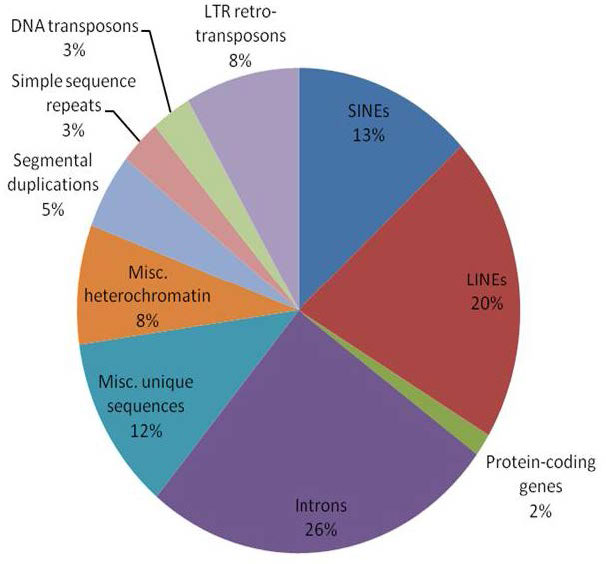
Figura 7.4 - Genes humanos ordenados por clase
Asuntos de tamaño
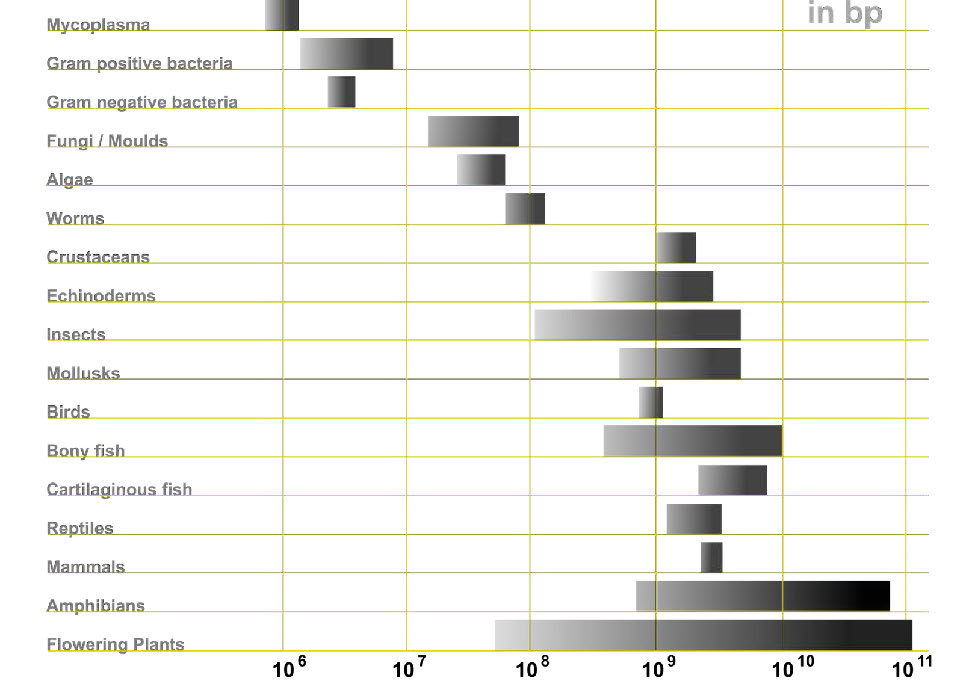
Una suposición de sentido común sobre los genomas sería que si los genes especifican proteínas, entonces cuantas más proteínas fabricara un organismo, más genes necesitaría tener, y así, mayor sería su genoma. La comparación de varios genomas muestra, sorprendentemente, que no existe necesariamente una relación directa entre la complejidad de un organismo y el tamaño de su genoma (Figura 7.5). Para entender cómo esto podría ser cierto, es necesario reconocer que si bien los genes están compuestos por ADN, todo el ADN no consiste en genes (para fines de nuestra discusión, definimos un gen como una sección del ADN que codifica un ARN o producto proteico). En el genoma humano, menos del 2% del ADN total parece ser el tipo de secuencia codificante que dirige la síntesis de proteínas. Durante muchos años, se creía que el ADN no codificante en los genomas era inútil, y fue descrito como “ADN basura” aunque era desconcertante que pareciera haber tanta secuencia “inútil”. Descubrimientos recientes, sin embargo, han demostrado que gran parte de este llamado ADN basura puede desempeñar papeles importantes en la evolución, así como en la regulación de la expresión génica.
Intrones
Entonces, ¿qué hace ahí todo el ADN no codificante? Sabemos que incluso las regiones codificantes de nuestro ADN están interrumpidas por secuencias no codificantes llamadas intrones. Esto es cierto para la mayoría de los genomas eucariotas. Un examen de genes en eucariotas muestra que las secuencias de intrones no codificantes pueden ser mucho más largas que las secciones codificantes del gen, o exones. La mayoría de los exones son relativamente pequeños y codifican menos de cien aminoácidos, mientras que los intrones pueden variar en tamaño desde varios cientos de pares de bases hasta muchos pares de kilobases (miles de pares de bases) de longitud. Para muchos genes en humanos, hay mucha más secuencia intrónica que secuencia codificante (también conocida como exón). Las secuencias de intrones representan aproximadamente una cuarta parte del genoma en humanos.
Otras secuencias no codificantes
¿Qué otro tipo de secuencias no codificantes hay? Una función para algunas secuencias de ADN que no codifican ARN o proteínas es especificar cuándo y en qué medida se usa o expresa un gen. Dichas regiones de ADN se denominan regiones reguladoras y cada gen tiene una o más secuencias reguladoras que controlan su expresión. Sin embargo, las secuencias reguladoras tampoco representan todo el resto del ADN en nuestros genomas.
Secuencias transponibles
Sorprendentemente, casi la mitad del genoma humano parece consistir en varios tipos de secuencias repetitivas. Se sabe que muchas de las secuencias repetitivas son elementos transponibles (transposones), secciones de ADN que pueden moverse dentro del genoma. A veces denominados “genes saltadores”, estos elementos transponibles pueden moverse de una ubicación cromosómica a otra, ya sea a través de un simple mecanismo de “cortar y pegar” que corta la secuencia de una región del ADN y la inserta en otra ubicación, o a través de un proceso llamado retrotransposición involucrando un intermedio de ARN.
LÍNEAS Y SINES
Hay millones de copias de cada una de las dos clases principales de tales elementos transponibles, los LINES (Elementos Intercalados Largos) y los SINEs (Elementos Intercalados Cortos) en nuestros genomas.
Las LINES y los SINE son una especie de elemento transponible llamado retrotransposones, secuencias que se copian en ARN, luego se transcriben de nuevo en ADN antes de insertarse en nuevas ubicaciones. Este movimiento no suele ser específico de secuencia, lo que significa que los transposones pueden insertarse aleatoriamente en el genoma, en muchos casos dentro de regiones codificantes. Como cabría esperar, esto puede alterar la función del gen. Los transposones también pueden insertarse dentro de las regiones reguladoras y cambiar la expresión de los genes que controlan. Como causa principal de mutación en los genomas, los transposones juegan un papel importante en la evolución.
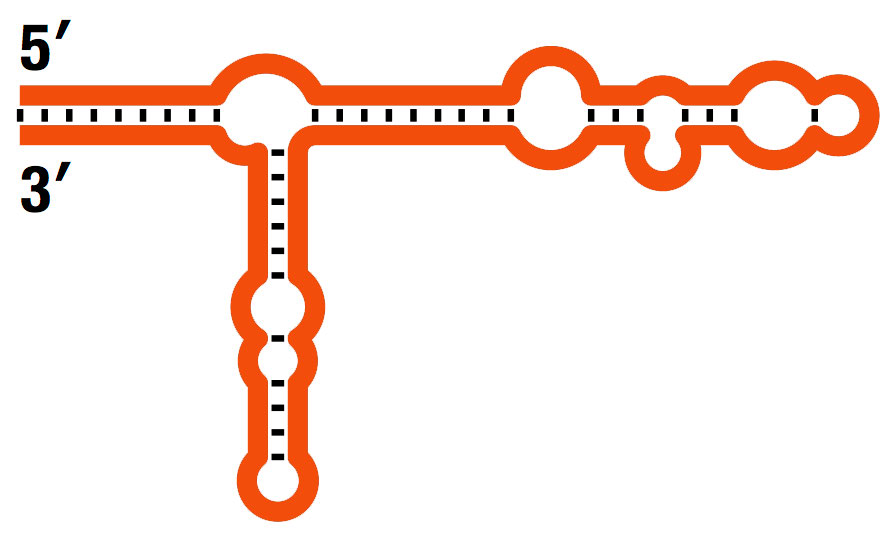
Finalmente, hallazgos recientes han demostrado que gran parte del genoma se transcribe en ARN, aunque solo alrededor del 2% codifica proteínas. ¿Cuáles son los ARN que no codifican proteínas? Los ARN ribosómicos (Figura 7.7) y los ARN de transferencia, junto con los ARN nucleares pequeños que funcionan en el corte y empalme, dan cuenta de algunos de estos transcritos no traducidos, pero no todos. Los ARN restantes son ARN reguladores, moléculas pequeñas que juegan un papel importante en la regulación de la expresión génica. A medida que entendemos más sobre los genomas, es cada vez más evidente que el llamado ADN “basura” es cualquier cosa menos.