8.7: Reacción en Cadena de Polimerasa (PCR)
- Page ID
- 53270
La clonación molecular fue el primer método disponible para aislar un gen de interés y hacer muchas copias del mismo para obtener cantidades suficientes del ADN para estudiar. Hoy en día, existe una manera más rápida y fácil de obtener grandes cantidades de una secuencia de ADN de interés -la reacción en cadena de la polimerasa (PCR). La PCR permite usar el poder de replicación del ADN para amplificar enormemente el ADN en un corto período de tiempo. Como saben, las células replican su ADN antes de dividirse, y al hacerlo, duplican la cantidad de ADN de la célula. La PCR imita esencialmente la replicación del ADN celular en el tubo de ensayo, copiando repetidamente el ADN diana una y otra vez, para producir grandes cantidades del ADN deseado.
Replicación selectiva
A diferencia de la replicación del ADN celular, que amplifica todo el ADN de una célula durante un ciclo de replicación, la PCR realiza una amplificación dirigida para replicar solo un segmento de ADN limitado por los dos cebadores que determinan dónde comienza la replicación de la ADN polimerasa. La Figura 8.34 ilustra el proceso. Cada ciclo de PCR implica tres etapas, desnaturalización, reasociación y extensión, cada una de las cuales ocurre a una temperatura diferente.
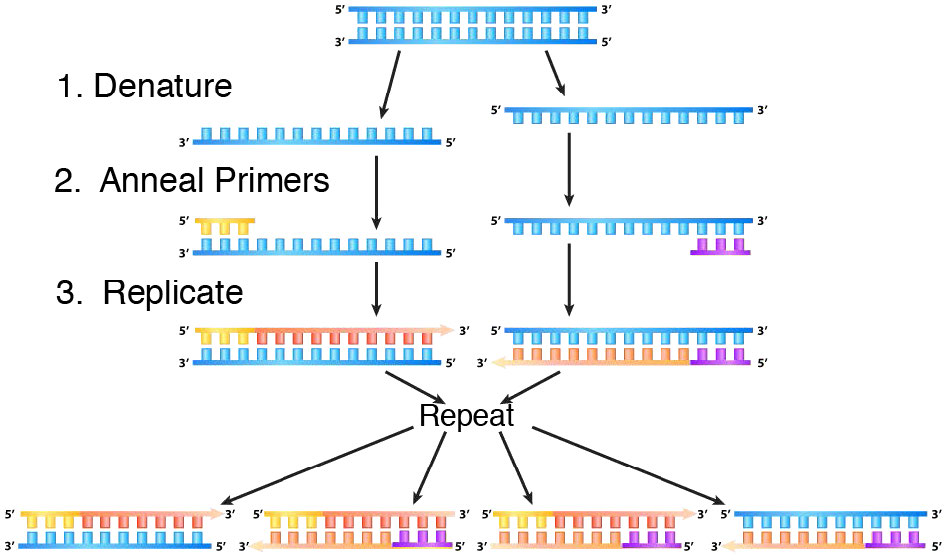
Los materiales de partida
Dado que la PCR es, básicamente, la replicación del ADN en un tubo de ensayo, se requieren todos los ingredientes habituales necesarios para la replicación del ADN:
- Una plantilla (el ADN que contiene la secuencia diana que se está copiando)
- Cebadores (para iniciar la síntesis de las nuevas cadenas de ADN)
- ADN polimerasa termoestable (para llevar a cabo la síntesis). La polimerasa necesita ser termoestable, ya que el calor se usa para separar las hebras de ADN molde en cada ciclo.
- DNTP (nucleótidos de ADN para construir las nuevas cadenas de ADN).
- El molde es el ADN que contiene la diana que quieres amplificar (la “diana” es la región específica del ADN que quieres amplificar).
Los cebadores son moléculas cortas de ADN monocatenario sintéticas cuya secuencia coincide con una región que flanquea la secuencia diana. Es posible sintetizar químicamente moléculas de ADN de cualquier secuencia de bases dada, para usar como cebadores. Para hacer cebadores de la secuencia correcta que se unirán al ADN molde, es necesario conocer un poco de la secuencia molde a cada lado de la región de ADN a amplificar. Las ADN polimerasas y los dNTP están disponibles comercialmente en empresas proveedoras de biotecnología.
Primero, todos los reactivos se mezclan entre sí. Los cebadores están presentes en millones de veces en exceso sobre el molde. Esto es importante porque cada hebra de ADN recién hecha parte de un cebador. El primer paso del proceso consiste en separar las hebras del ADN diana calentando hasta casi hervir.
A continuación, la solución se enfría a una temperatura que favorece que las secuencias de ADN complementarias se encuentren entre sí y hagan pares de bases, un proceso llamado recocido. Dado que los cebadores están presentes en gran exceso, las secuencias complementarias a las que se dirigen se encuentran fácilmente y se emparejan las bases con los cebadores. Estos cebadores dirigen la síntesis del ADN. Solo donde un cebador se hibrida con una cadena de ADN se producirá la replicación, ya que las ADN polimerasas requieren de un cebador para comenzar la síntesis de una nueva cadena.
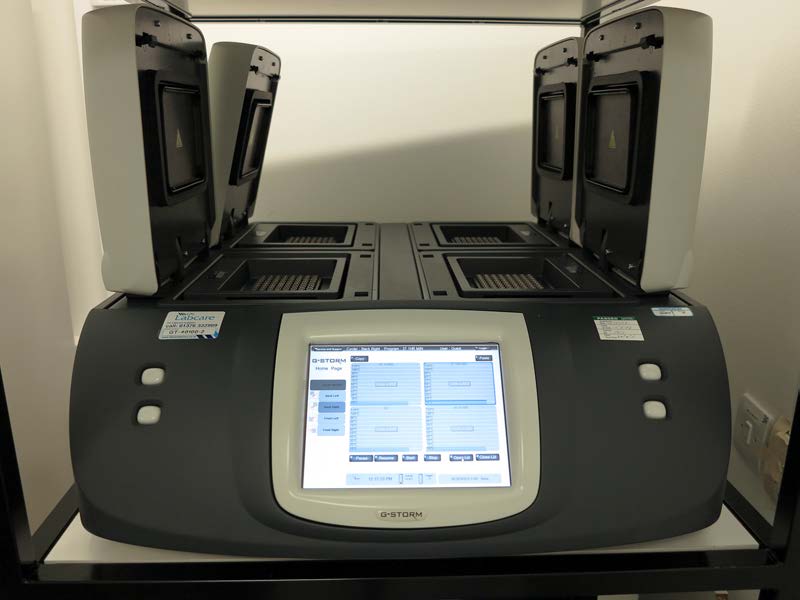
Figura 8.36 - Un sistema termociclador de PCR. Wikipedia
Extensión
En el tercer paso del proceso, la ADN polimerasa replica ADN por extensión desde el extremo 3' del cebador, formando una nueva cadena de ADN. Al final del primer ciclo, hay el doble de moléculas de ADN, al igual que en la replicación celular. Pero en PCR, el proceso se repite, generalmente por entre 25 y 30 ciclos. Al final del proceso, hay un rendimiento teórico de 230 (más de mil millones de veces) más ADN del que había para comenzar. (Este enorme poder de amplificación es la razón por la que la PCR es tan útil para las investigaciones forenses, donde muy pequeñas cantidades de ADN pueden estar disponibles en la escena del crimen).
Los ciclos de temperatura se controlan en un termociclador, el cual eleva y baja repetidamente las temperaturas de acuerdo con el programa establecido. Dado que cada ciclo se puede completar en un par de minutos, toda la amplificación se puede completar muy rápidamente. El ADN resultante se analiza en un gel para asegurar que es del tamaño esperado, y dependiendo de para qué se vaya a utilizar, también se puede secuenciar, para tener la certeza de que es el fragmento deseado.
Muagénesis
La PCR se usa frecuentemente para obtener secuencias génicas que se van a clonar en vectores para la expresión de proteínas, por ejemplo. Además de simplicidad y velocidad, la PCR también tiene otras ventajas. Debido a que se pueden sintetizar cebadores que difieren de la secuencia molde en cualquier posición dada, es posible usar PCR para mutagénesis dirigida al sitio. Es decir, la PCR puede usarse para mutar un gen en una posición deseada en la secuencia. Esto permite que las proteínas codificadas por los genes normales y mutantes sean expresadas, purificadas y comparadas.
Análisis de la expresión génica
También se puede usar PCR para medir la expresión génica. Donde en PCR no se determina la cantidad de producto amplificado hasta el final de todos los ciclos, se utiliza una variación llamada PCR cuantitativa en tiempo real, en la que se mide la acumulación de producto en cada ciclo. Esto es posible porque las máquinas de PCR en tiempo real cuentan con un módulo detector que puede medir los niveles de un marcador fluorescente en la reacción, con la cantidad de fluorescencia proporcional a la cantidad de producto amplificado. Al seguir la acumulación de producto a lo largo de los ciclos es posible calcular la cantidad de plantilla inicial. Para medir la expresión génica, el molde utilizado es ARNm transcrito inversamente en ADNc (ver más abajo). Este acoplamiento de transcripción inversa con PCR cuantitativa en tiempo real se denomina qRT-PCR.


