3.3: Proteínas
- Page ID
- 53634
Mientras que todos los nucleótidos son solubles en agua y tienen la misma composición básica (azúcar, base, fosfato) y los azúcares también son solubles en agua y en su mayoría contienen 5 o 6 carbonos (algunas excepciones), los aminoácidos (estructura general a continuación) son estructural y químicamente diversos.
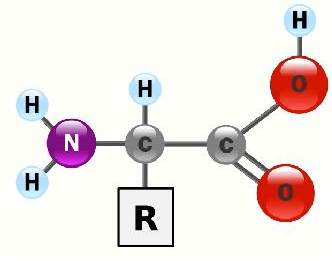
Aunque todos los aminoácidos son, de hecho, solubles en agua, las interacciones de sus cadenas laterales con el agua difieren significativamente. Esto es importante, porque es solo en las cadenas laterales (grupos R) donde los aminoácidos difieren entre sí. Con base en cadenas laterales, podemos agrupar los 20 aminoácidos que se encuentran en las proteínas de la siguiente manera:
- Aromático (fenilalanina, tirosina, triptófano)
- Alifático (leucina, isoleucina, alanina, metionina, valina)
- Hidroxilo/sulfhidrilo (treonina, serina, tirosina, cisteína)
- Carboxiamida (glutamina, asparagina)
- R-Acidos (ácido glutámico, ácido aspártico)
- R-aminas (lisina, histidina, arginina)
- impar (glicina, prolina)
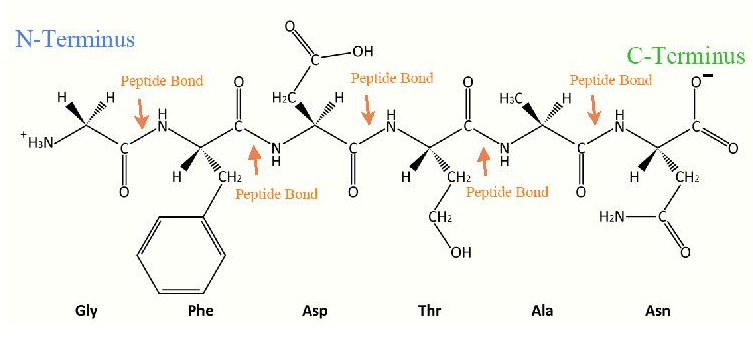
Tenga en cuenta que la tirosina tiene un grupo hidroxilo y se ajusta en dos categorías. Tenga en cuenta también que los libros de bioquímica varían en la forma en que organizan los aminoácidos en categorías. Los aminoácidos se unen entre sí mediante enlaces peptídicos. Esto introduce un aspecto ligeramente simplificador a la estructura de las proteínas: solo se necesita considerar el posicionamiento de los grupos R alrededor de cada enlace peptídico cuando se determina la estructura de la proteína de manera esquemática. Las proteínas que se encuentran en ambientes acuosos, como el citoplasma de la célula, tienen sus aminoácidos dispuestos de manera que aquellas con cadenas laterales hidrófilas (como la treonina o la lisina) predominan en el exterior de la proteína para interactuar con el agua. Los aminoácidos hidrófobos en estas proteínas se encuentran predominantemente en el interior. Cuando se examina la estructura de las proteínas en ambientes no acuosos, como el interior de una bicapa lipídica, la disposición se voltea; predominan los hidrófobos en el exterior donde pueden interactuar con las cadenas laterales hidrófobas de los ácidos grasos de membrana y se disponen los aminoácidos hidrófilos cualquier lugar donde puedan entrar en contacto con el agua. Para una proteína como la porina, que proporciona un canal interior a través del cual puede pasar el agua, aquí es donde se encuentran los hidrofílicos. Para las proteínas transmembrana, que se proyectan a través de ambos lados de la membrana, los hidrófilos se encuentran en cada punto donde la cadena polipeptídica emerge de la membrana.
Estructura primaria
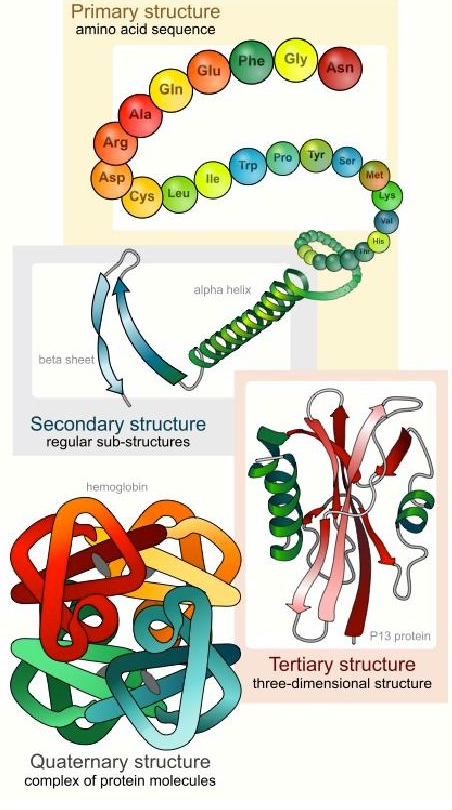
¿Cómo obtienen las proteínas tales arreglos de aminoácidos? Como veremos, las estructuras de todas las proteínas surgen en última instancia de sus secuencias de aminoácidos. La secuencia de aminoácidos se conoce como la estructura primaria y los cambios en ella pueden afectar cualquier otro nivel de estructura, así como las propiedades de una proteína. La estructura primaria de una proteína llegó a su estado actual como resultado de la mutación y selección a lo largo del tiempo evolutivo. En una escala de tiempo más inmediata, la estructura proteica 3D surge como resultado de un fenómeno llamado plegamiento. El plegamiento de proteínas resulta de tres elementos estructurales diferentes más allá de la estructura primaria. Se les conoce como estructuras secundarias, terciarias y cuaternarias, cada una de las cuales surge de interacciones entre aminoácidos progresivamente más distantes en la estructura primaria.
Estructura secundaria
Las interacciones entre aminoácidos dentro de aproximadamente diez unidades entre sí dan lugar a estructuras repetitivas regulares. Estas estructuras secundarias incluyen las conocidas cadenas de hélice alfa y beta. Ambos fueron predichos por Linus Pauling, Robert Corey y Herman Branson en 1951. Cada estructura tiene características únicas. Usamos los términos subir, repetir y tono para describir los parámetros de una hélice. La repetición es el número de residuos en una hélice antes de que comience a repetirse. El ascenso es la distancia que la hélice eleva con la adición de cada residuo. El paso es la distancia entre las vueltas de la hélice.
Hélice Alfa
La hélice alfa (Figura 3.1.3) se forma como resultado de interacciones entre aminoácidos separados por cuatro residuos. Interesante; y, las cadenas laterales de los aminoácidos en una hélice alfa están todas apuntadas hacia afuera desde el eje de la hélice. Las hélices alfa tienen una repetición de 3.6 residuos de aminoácidos por giro de la hélice, lo que significa que cuatro vueltas de la hélice tienen aproximadamente 14 residuos de aminoácidos. Los enlaces de hidrógeno ocurren entre el C=O de un aminoácido y el N-H de otro aminoácido a cuatro residuos distantes y estos ayudan a estabilizar la estructura (nótese que los C=O y N-H involucrados son parte de la cadena principal del polipéptido, no los grupos R). Algunos aminoácidos tienen altas tendencias en la formación de hélices. Incluyen metionina, alanina, leucina, glutamato sin carga y lisina. Otros, como la prolina, la glicina y el aspartato cargado negativamente, desfavorecen su formación.
Hebras Beta
Las hebras beta son la hélice más fundamental, teniendo esencialmente una columna vertebral 2D de 'pliegue' como las de los pliegues de una cortina. De hecho, las hebras beta se pueden organizar juntas para formar lo que se llaman hojas beta. También se conocen otras estructuras regulares. ¿Qué determina si un tramo dado de una proteína está en una estructura helicoidal u otra? Aquí es donde juegan un papel la forma y la química de las cadenas laterales.

Proteínas fibrosas
No todas las proteínas tienen cantidades significativas de estructura terciaria o cuaternaria. (Como veremos, estos dos últimos niveles de estructura surgen de 'curvas' en cadenas polipeptídicas e interacciones entre cadenas polipeptídicas separadas, respectivamente).
La alfa queratina, por ejemplo, es a lo que nos referimos como una proteína fibrosa (también llamada escleroproteína). La alfa queratina tiene estructura primaria y estructura secundaria, pero poca estructura terciaria o cuaternaria.
En consecuencia, la alfa queratina existe principalmente como fibras largas, como las que se encuentran en el cabello. La beta-queratina es una proteína fibrosa más dura que se encuentra en uñas, escamas y garras. Se compone principalmente de hojas beta. La prolina, que es el aminoácido menos flexible, debido a la unión de la cadena lateral al agarre alfa-amino, es menos probable que se encuentre en las hélices alfa, pero curiosamente se encuentra abundantemente en la proteína fibrosa conocida como colágeno. El colágeno (página anterior) es la proteína más abundante en el cuerpo humano y es el 'pegamento' que literalmente nos une. ¿Cómo la inflexibilidad de la prolina permite que esté en una hélice? La respuesta es probablemente la abundancia paralela en colágeno de glicina, que contiene el grupo lateral más pequeño y por lo tanto tiene la mayor flexibilidad.
Como una interesante luz lateral de la presencia de prolina en el colágeno está la modificación química de las prolinas, por la adición de grupos hidroxilo, después de que se elabora la proteína. Tales 'modificaciones postraduccionales' no son infrecuentes. La treonina, la serina y la tirosina frecuentemente tienen sus cadenas laterales de hidroxilo fosforiladas. Las lisinas en colágeno también se hidroxilan postraduccionalmente. Las prolinas y lisinas hidroxiladas juegan un papel en la formación de enlaces de hidrógeno intercatenas y la reticulación de triples hélices durante el ensamblaje de fibrillas de colágeno. Estos enlaces proporcionan integridad estructural al colágeno. Las enzimas que agregan hidroxilos a la prolina y lisina requieren vitamina C (ácido ascórbico) para su actividad. La falta de vitamina C conduce a la producción de fibrillas de colágeno debilitadas, resultando en una condición llamada escorbuto.
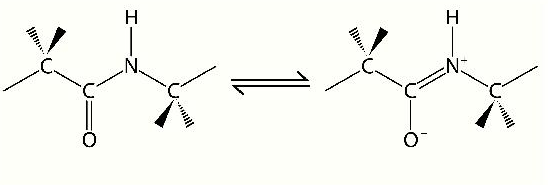
El oxígeno carbonílico del enlace peptídico puede existir en resonancia con el enlace C-N, dando al enlace peptídico características de un doble enlace e imponiendo limitaciones para la rotación alrededor del mismo. Si trat el enlace peptídico como un doble enlace, entonces las disposiciones de los enlaces de carbono adyacentes alrededor de él se pueden considerar como en las configuraciones cis o trans. En las proteínas, no es sorprendente que la disposición preferida de estos grupos sea fuertemente trans (1000/1). De los 20 aminoácidos, el que favorece los enlaces peptídicos en la configuración cis más comúnmente es la prolina, pero incluso para la prolina, se prefiere fuertemente el isómero trans.
Figura 3.2.5: Colágeno.
Parcelas Ramachandran
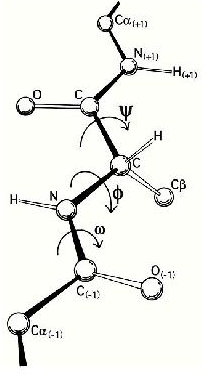
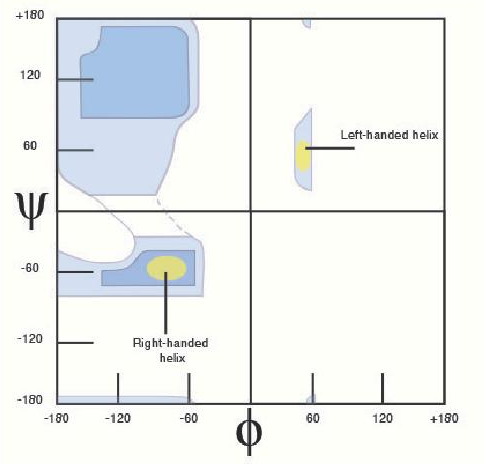
Otra consecuencia de considerar el enlace peptídico como un doble enlace es que reduce el número de ángulos rotacionales variables del esqueleto polipeptídico. Los términos phi y psi se refieren a ángulos rotacionales sobre los enlaces entre el carbono N-alfa y el carbono alfa-carbono-carbonilo respectivamente (página anterior). Dada la voluminosidad de los grupos R, el fenómeno del impedimento estérico y la tendencia de las cadenas laterales cercanas a interactuar entre sí, se podría esperar encontrar un sesgo en los valores de phi y psi. En efecto, eso es exactamente lo que se observa. El doctor G.N. Ramachandran propuso tal resultado y, en una trama que lleva su nombre, representó la probabilidad teórica de que cada ángulo apareciera en un polipéptido. Observaciones más recientes de los ángulos phi y psi reales en datos de la base de datos de proteínas PDB confirman las predicciones del Dr. Ramachandran. En la trama anterior, las hebras beta encajan muy bien en la sección azul más oscura en la parte superior y las hélices alfa encajan en la sección amarilla cerca de la mitad.
Estructura Terciaria
En contraste con las estructuras secundarias, que surgen de interacciones entre aminoácidos cercanos en la estructura primaria, la estructura terciaria surge de interacciones entre aminoácidos más distantes en la estructura primaria. Tales interacciones no son posibles en una fibra que se estira sin cesar porque cada aminoácido colocado entre dos aminoácidos hace que se alejen más el uno del otro en lo que es esencialmente las dos dimensiones de una estructura secundaria. Para que los aminoácidos distantes interactúen, deben acercarse más y esto requiere la flexión y plegamiento de la cadena polipeptídica. Las proteínas con tales estructuras se denominan “globulares” y son, con mucho, la clase de proteínas más abundante. En efecto, es en las proteínas globulares donde tenemos las imágenes más vívidas de los resultados del plegamiento. Los “pliegues” en los polipéptidos surgen como resultado de 'curvas' entre regiones de estructura secundaria (como hélice alfa o hebras beta). Tales estructuras pueden preferirse debido a la incompatibilidad de una cadena lateral de aminoácido dada para una estructura secundaria formada por los aminoácidos que la preceden. Las curvas ocurren comúnmente en las proteínas y la prolina a menudo está implicada. Las curvas no tienen la geometría predecible de hélices alfa o hebras beta y a menudo se denominan bobinas aleatorias. Por lo tanto, aunque la estructura de la proteína puede describirse fácilmente como regiones de estructura secundaria separadas por curvas, la variabilidad de las estructuras de curvatura hace que la predicción de la estructura terciaria a partir de la secuencia de aminoácidos sea enormemente más difícil que identificar/predecir regiones de estructura secundaria.
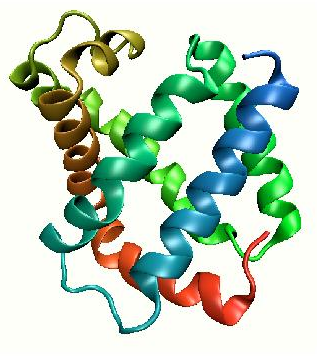
Efecto hidrofóbico
Es a nivel de estructura terciaria donde se produce la disposición característica de aminoácidos hidrófobos e hidrófilos en una proteína. En un ambiente acuoso, para que una proteína permanezca soluble, debe tener interacciones favorables con el agua que la rodea, de ahí el posicionamiento de aminoácidos hidrófilos externamente. Otro impulso para el fenómeno del plegado es un poco más difícil de entender. Se le conoce como el efecto hidrofóbico. A nivel químico, tiene sentido: los aminoácidos hidrófobos 'preferirán' interactuar entre sí internamente y lejos del agua. La fuerza impulsora de este fenómeno, sin embargo, es un poco más conceptualmente difícil. Considera una botella que contenga aceite y agua. Como todos saben, los dos líquidos no se mezclarán y en su lugar formarán capas separadas. Una pregunta razonable podría ser por qué hacen esto en lugar de uno existente como pequeños glóbulos dentro del otro. La respuesta a esa pregunta, así como el posicionamiento de los aminoácidos hidrófobos en el interior de las proteínas solubles en agua, es el efecto hidrófobo. Para entender el efecto hidrofóbico, realice el siguienteexperimento: tome la mezcla agua-aceite y agítela vigorosamente. Esto obligará a las capas a mezclarse y se observará que pequeños glóbulos tanto de agua como de aceite pueden, de hecho, encontrarse inicialmente en la capa de cada uno. Sin embargo, con el tiempo, los pequeños glóbulos se rompen y se fusionan con la capa apropiada. Esto se debe al fenómeno de entropía y consideración de la superficie. Primero, la suma del área de superficie de los pequeños globos incrustados es mucho mayor que el área de la región entre las dos capas después de que la mezcla haya terminado. Cuanto más pequeños son los glóbulos, mayor es la superficie de interacción entre el petróleo y el agua. El área superficial mínima posible de interacción ocurre cuando no hay globos en absoluto, solo dos capas y nada más.

¿Cómo se relaciona esto con la entropía? Las interacciones entre las capas hidrofóbicas hacen que las moléculas en la interfaz se organicen precisamente/regularmente para minimizar sus interacciones. Por lo tanto, el orden ocurre en las interfaces de capa. La cantidad
máxima de pedidos ocurre cuando interactúan las áreas superficiales máximas de petróleo y agua. Los pequeños glóbulos dan lugar a una mayor superficie
expuesta entre el agua y las capas hidrofóbicas y, como consecuencia, más ordenamiento. Dado que la entropía en un sistema cerrado tiende a aumentar, tenderá a reducir la cantidad de pedidos, si se deja sola. De esta manera, se puede aumentar el orden a escala nanoscópica (formando glóbulos) aplicando energía en forma de agitación. Sin embargo, cuando se deja solo, el sistema aumentará su trastorno al reducir las interacciones entre los grupos hidrófobos y los hidrófilos.
En la mezcla de aceite y agua, esto hace que los pequeños globos se rompan y produzcan las dos capas con las que estamos familiarizados porque esta es la superficie
mínima que se puede hacer entre las dos capas y, por lo tanto, la menos ordenada. En las proteínas, las cadenas laterales de aminoácidos hidrófobos se 'protegen' del agua mediante la colocación interna de la proteína, reduciendo así también las interfaces entre los residuos hidrófobos y el agua. En ambos casos se incrementa la entropía, debido a la menor organización de las capas. Una vez formadas, las interacciones entre las cadenas laterales de aminoácidos hidrófobos ayudan a estabilizar la estructura global de la proteína.
Estructura cuaternaria
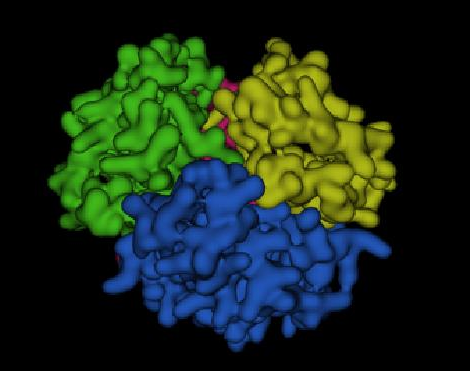
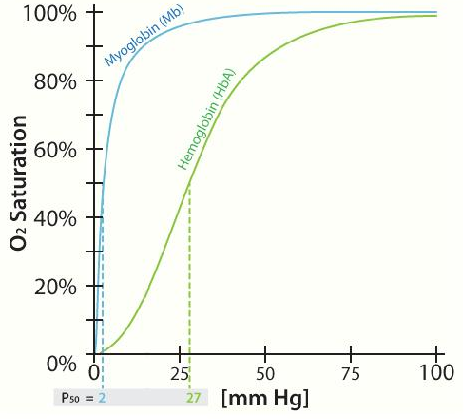
El último nivel de estructura proteica que consideraremos es el de estructura cuaternaria. Para tener estructura cuaternaria, una proteína debe tener múltiples subunidades polipeptídicas porque la estructura implica la disposición de esas subunidades entre sí. Considera la hemoglobina, la proteína portadora de oxígeno de nuestra sangre. Contiene dos subunidades idénticas conocidas como alfa y otras dos idénticas conocidas como beta. Estos están dispuestos juntos de una manera como se muestra en la página anterior. Por el contrario, la proteína de almacenamiento de oxígeno relacionada conocida como mioglobina solo contiene una sola subunidad. La hemoglobina tiene estructura cuaternaria, pero la mioglobina no. Las proteínas de subunidades múltiples son comunes en las células y dan lugar a propiedades muy útiles que no se encuentran en las proteínas de una sola subunidad. En el caso de la hemoglobina, las múltiples subunidades confieren la propiedad de cooperatividad, afinidad variable por el oxígeno dependiendo de la concentración de este último. En el caso de las enzimas, puede impartir alosterismo, la capacidad de tener la actividad de la enzima alterada por la interacción con una molécula efectora. Discutiremos el alosterismo en detalle en el próximo capítulo.
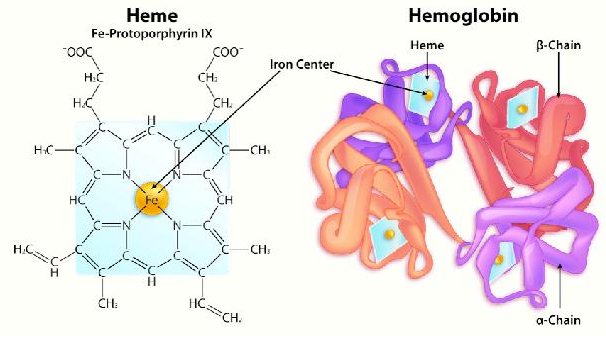
Otras características estructurales de proteínas
No todo lo que se encuentra en una proteína es un aminoácido. Las proteínas frecuentemente tienen otros grupos químicos, conocidos como grupos protésicos, unidos a ellas, que son necesarios para la función de una proteína. Los ejemplos incluyen el anillo de porfirina del hemo en la mioglobina y la hemoglobina que porta un hierro para que se pueda unir el oxígeno. Los metales son frecuentemente empleados por las enzimas en su catálisis. Varias vitaminas (denominadas coenzimas), como la tiamina (B1) y la riboflavina (B2) se modifican y se unen químicamente a las enzimas para ayudarlas a realizar funciones catalíticas específicas.
Cooperatividad
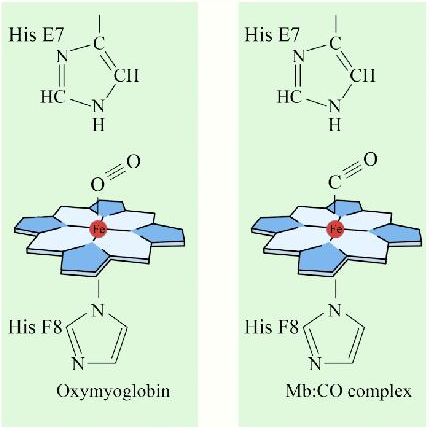
Un aspecto interesante e importante de algunas proteínas es el fenómeno de la cooperatividad. La cooperatividad se refiere al hecho de que la unión de una molécula ligando por una proteína favorece la unión de moléculas adicionales del mismo tipo. La hemoglobina, por ejemplo, exhibe cooperatividad cuando la unión de una molécula de oxígeno por el hierro del grupo hemo en una de las cuatro subunidades provoca un ligero cambio de conformación en la subunidad. Esto sucede porque el hierro hemo está unido a una cadena lateral de histidina y la unión del oxígeno 'levanta' el hierro junto con el anillo de histidina (también conocido como el anillo de imidazol).
Dado que cada subunidad de hemoglobina interactúa con las otras subunidades e influye en ellas, también son inducidas a cambiar de forma ligeramente cuando la primera subunidad se une al oxígeno (una transición que se describe como que va del estado T al estado R). Estos cambios de forma favorecen también a cada una de las subunidades restantes que se unen al oxígeno. Esto es muy importante en los pulmones donde el oxígeno es captado por la hemoglobina, ya que la unión de la primera molécula de oxígeno facilita la rápida absorción de más moléculas de oxígeno. En los tejidos, donde la concentración de oxígeno es menor, el oxígeno deja la hemoglobina y las proteínas .ips desde el estado R de nuevo al estado T.
La cooperatividad es solo uno de los muchos aspectos estructurales fascinantes de la hemoglobina que ayudan al cuerpo a recibir oxígeno donde se
necesita y recogerlo donde sea abundante. La hemoglobina también ayuda en el transporte del producto de la respiración celular (dióxido de carbono) desde los tejidos que la producen hasta los pulmones donde se exhala. Consideremos estos individualmente.
Efecto Bohr
El Efecto Bohr fue descrito por primera vez hace más de 100 años por Christian Bohr. Se muestra gráficamente (arriba a la izquierda), el efecto observado es que la afinidad de la hemoglobina por el oxígeno disminuye a medida que disminuye el pH y/o a medida que aumenta la concentración de dióxido de carbono. La unión de los protones por la histidina ayuda a facilitar los cambios estructurales en la proteína y también con la captación de dióxido de carbono. Fisiológicamente, esto tiene una gran importancia porque los tejidos que respiran activamente (como los músculos contraídos) requieren oxígeno y liberan protones y dióxido de carbono. Cuanto mayor sea la concentración de protones y dióxido de carbono, más oxígeno se libera para alimentar a los tejidos que más lo necesitan.
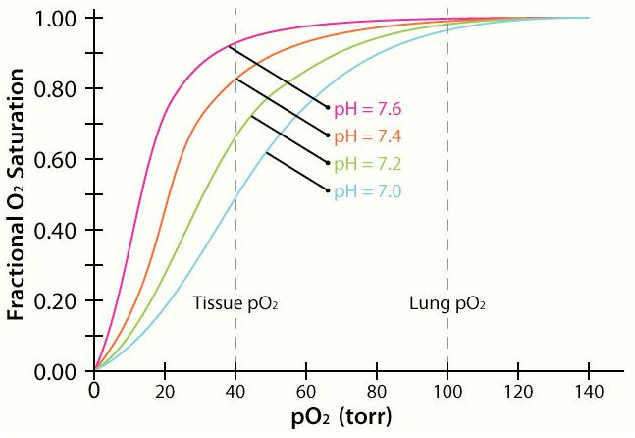
2,3 BPG
Otra molécula que afecta la liberación de oxígeno por la hemoglobina es el 2,3 bisfosfosglicerato (también llamado 2,3 BPG o simplemente BPG). Al igual que los protones y el dióxido de carbono, 2,3 BPG se produce respirando activamente los tejidos, como subproducto del metabolismo de la glucosa. La molécula 2,3 BPG encaja en el 'agujero de la donuta' de la hemoglobina adulta. Dicha unión de 2,3 BPG favorece el estado T (apretado) de la hemoglobina, que tiene una afinidad reducida por el oxígeno. En ausencia de 2,3 BPG, la hemoglobina puede existir en el estado R (relajado), el cual tiene una alta afinidad por el oxígeno.
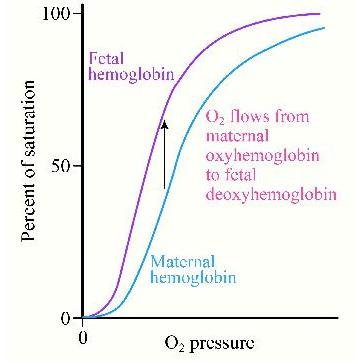
Hemoglobina fetal
La hemoglobina adulta libera oxígeno cuando se une a 2,3 BPG. Esto contrasta con la hemoglobina fetal, que tiene una configuración ligeramente diferente (α2γ2) que la hemoglobina adulta (α2ß). La hemoglobina fetal tiene una mayor afinidad por el oxígeno que la hemoglobina materna, lo que permite que el feto obtenga oxígeno de manera efectiva de la sangre de la madre. Parte de la razón de la mayor afinidad de la hemoglobina fetal por el oxígeno es que no une 2,3 BPG.
Otro dato significativo sobre 2,3 BPG es que su concentración es mayor en la sangre de los fumadores que en la de los no fumadores. En consecuencia, la hemoglobina en la sangre de un fumador pasa más tiempo en el estado T que en el estado R. Ese es un problema cuando está en los pulmones, donde estar en el estado R es necesario para cargar al máximo la hemoglobina con oxígeno. Un nivel alto en sangre de 2,3 BPG es una de las razones por las que los fumadores tienen dificultad para respirar cuando hacen ejercicio; tienen una capacidad de carga de oxígeno reducida.
Por último, aunque no está relacionado directamente con 2,3 BPG, los fumadores tienen otra razón por la que su capacidad de carga de oxígeno es menor que la de los no fumadores. El humo del cigarrillo contiene monóxido de carbono y esta molécula, que tiene dimensiones casi idénticas al oxígeno molecular, compite eficazmente con el oxígeno para unirse al átomo de hierro del hemo. Parte de la toxicidad del monóxido de carbono se debe a su capacidad para unirse a la hemoglobina y evitar que el oxígeno se una.
Desnaturalización
Para las proteínas, la función depende de una estructura precisa. La pérdida de la estructura plegada precisa de una proteína se conoce como desnaturalización y suele ir acompañada de pérdida de función. Cualquiera que alguna vez haya trabajado para purificar una enzima sabe lo fácil que es para uno perder su actividad. Algunas enzimas, como la ribonucleasa, son notablemente estables incluso en condiciones muy duras. Para la mayoría de los demás, un pequeño cambio de temperatura o pH puede afectar drásticamente la actividad. Las razones de estas diferencias varían, pero se relacionan con 1) la fuerza de las fuerzas que mantienen unida la estructura y 2) la capacidad de una proteína para replegarse después de ser desnaturalizada. Consideremos estos por separado a continuación.
Estructuras estabilizadoras de fuerzas
Los aminoácidos están unidos entre sí por enlaces peptídicos. Estos enlaces covalentes son extraordinariamente estables a pH neutros, pero pueden romperse por hidrólisis con calor en condiciones ácidas. Los enlaces peptídicos, sin embargo, solo estabilizan la estructura primaria y, de hecho, son la única fuerza relevante responsable de ello. La estructura secundaria, por otro lado, generalmente se estabiliza por fuerzas más débiles, incluidos los enlaces de hidrógeno. Los enlaces de hidrógeno se rompen fácilmente por el calor, la urea o el cloruro de guanidinio.
Las fuerzas que estabilizan la estructura terciaria incluyen interacciones iónicas, enlaces disulfuro, interacciones hidrófobas, enlaces metálicos y
enlaces de hidrógeno. De estas, las interacciones iónicas son más sensibles a los cambios de pH. Los enlaces hidrófobos son los más sensibles a los detergentes. Así,
lavarse las manos ayuda a matar las bacterias al desnaturalizar las proteínas críticas que necesitan para sobrevivir. Los enlaces metálicos son sensibles a la oxidación/reducción. Romper los enlaces disulfuro requiere un agente oxidante fuerte, como el ácido perfórmico o un agente reductor fuerte
en otro disulfuro, como mercaptoetanol o ditiotreitol.
Las estructuras cuaternarias son estabilizadas por las mismas fuerzas que la estructura terciaria y tienen las mismas sensibilidades.
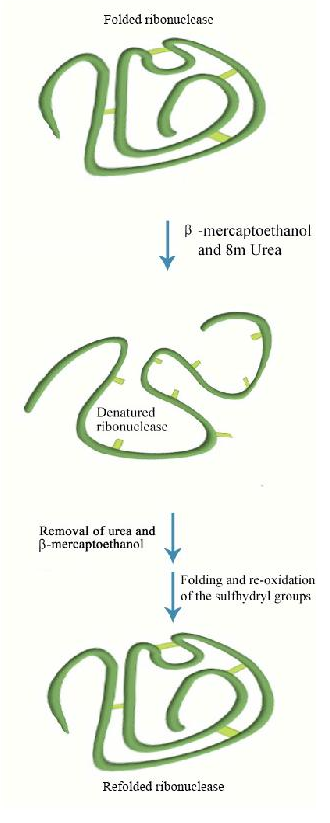
Replegamiento de Proteínas Desnaturalizadas
Toda la información para el plegamiento de proteínas está contenida en la estructura primaria de la proteína. Puede parecer curioso entonces que la mayoría de las proteínas no se replegen en su forma adecuada y completamente activa después de que se hayan desnaturalizado y se haya eliminado el desnaturalizante. Algunos, de hecho, se replegan correctamente en estas circunstancias. Un buen ejemplo es la ribonucleasa bovina (también llamada RNasa). Su actividad catalítica es muy resistente al calor y a la urea. Sin embargo, si se trata la enzima con mercaptoetanol (que rompe los enlaces disulfuro) antes del tratamiento con urea y
calentamiento, se pierde actividad, lo que indica que los enlaces disulfuro covalentes ayudan a estabilizar la estructura general de la enzima. Si se permite que la
mezcla de enzimas se enfríe de nuevo a temperatura ambiente, con el tiempo reaparece alguna actividad enzimática, lo que indica que la ribonucleasa puede replegarse en las condiciones adecuadas.
Desnaturalización irreversible
La mayoría de las enzimas, sin embargo, no se comportan como ribonucleasa. Una vez desnaturalizada, su actividad no puede ser recuperada de manera significativa. Esto puede parecer contradecir la idea de que la información de plegamiento es inherente a la secuencia de aminoácidos en la proteína. No lo hace. La razón por la que la mayoría de las enzimas no pueden replegarse correctamente se debe a dos fenómenos. En primer lugar, el plegamiento normal puede ocurrir a medida que se hacen las proteínas. Las interacciones entre aminoácidos tempranos en la síntesis no se “confunden” por interacciones con aminoácidos posteriores en la síntesis porque esos aminoácidos no están presentes cuando comienza la síntesis de proteínas. En muchos casos, el plegamiento adecuado de los polipéptidos recién hechos también es asistido por proteínas especiales llamadas chaperonas. Las chaperonas se unen a proteínas recién hechas, previniendo interacciones que podrían resultar en plegamiento erróneo. Así, el plegamiento temprano y la asistencia de las chaperonas eliminan algunas interacciones potenciales de “plegamiento erróneo” que pueden ocurrir si toda la secuencia estaba presente cuando se inició el plegamiento.
Los polipéptidos desnaturalizados de longitud completa tienen muchos más pliegues erróneos potenciales que pueden ocurrir. Una segunda razón por la que la mayoría de las proteínas no se replegan correctamente después de la desnaturalización es probablemente que el plegamiento, como cualquier otro fenómeno natural, es impulsado por la minimización de energía. Aunque la estructura plegada puede tener una energía baja, el camino que conduce a ella puede no ser todo cuesta abajo. Al igual que una reacción química que tiene energías de activación que deben superarse para que ocurra la reacción, el plegamiento probablemente tiene picos y valles de energía que no conducen automáticamente directamente al pliegue adecuado. Nuevamente, el plegamiento durante la síntesis lleva a la proteína a lo largo de un camino mejor definido a través del laberinto energético de plegamiento que las proteínas desnaturalizadas de longitud completa no pueden navegar.
Priones y Desplegamiento
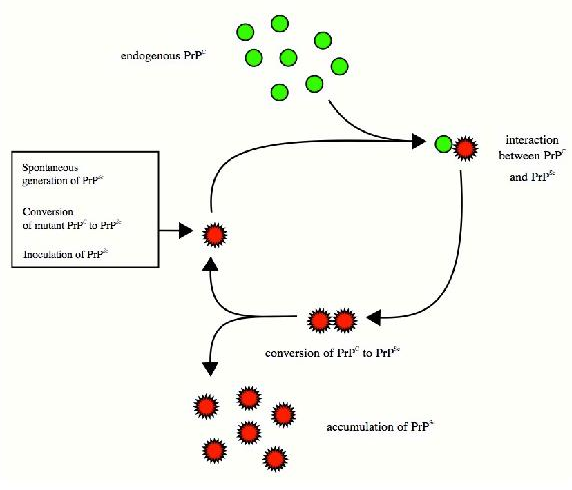
El plegamiento y la estabilidad de las proteínas plegadas es una consideración importante para las proteínas llamadas “infecciosas” conocidas como priones. Estas misteriosas proteínas, que están implicadas en enfermedades, como la enfermedad de las vacas locas y la condición humana relacionada conocida como enfermedad de Creutzfeldt-Jakob, resultan del plegamiento inadecuado de una proteína cerebral conocida como PrP. La proteína mal plegada tiene dos propiedades importantes que conducen a la enfermedad. En primer lugar, tiende a agregarse en grandes complejos llamados placas amiloides que dañan/ destruyen las células nerviosas del cerebro, conduciendo en última instancia a demencia y pérdida de la función cerebral.
Segundo, y probablemente peor, la proteína mal plegada “induce” a otras copias de la misma proteína a plegarse erróneamente también. Así, una
proteína mal plegada actúa como un centro catalítico y la enfermedad progresa rápidamente. Se plantea la pregunta de cómo la proteína PrP se despliega erróneamente para empezar, pero la respuesta a esto no está clara. Hay sugerencias de que la exposición en la dieta a proteínas mal plegadas puede
ser un factor, pero esto se disputa. Un brote de la enfermedad de las vacas locas en Gran Bretaña en la década de 1980 fue seguido por un aumento en la incidencia de una rara forma de enfermedad de Creutzfeld-Jakob humana llamada variante CJD (v-CJD), dando cierta credibilidad a la hipótesis. Es posible que el plegamiento erróneo de muchas proteínas ocurra esporádicamente sin consecuencia u observación, pero si PrP se despliega, los resultados son fácilmente evidentes. Por lo tanto, la enfermedad de Creutzfeld-Jakob puede finalmente dar una idea del proceso de plegado en sí.