8.4: Receptores Acoplados a Proteínas G (GPCR)
- Page ID
- 53843
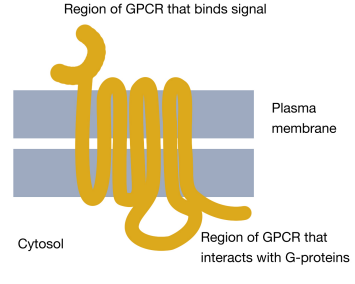
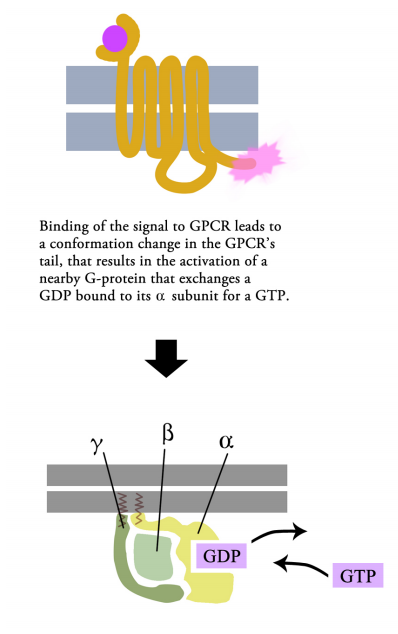
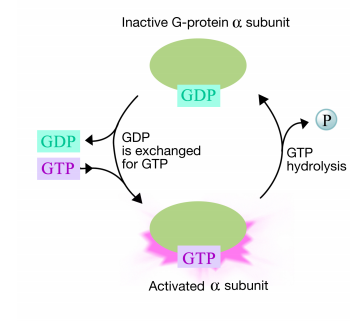
Con estos antecedentes sobre la estructura y propiedades generales de los GPCR y las proteínas G, ahora podemos observar lo que sucede cuando una señal llega a la superficie celular y se une a un GPCR. La unión de una molécula señal por la parte extracelular del receptor unido a la proteína G hace que la cola citosólica del receptor interactúe con, y altere la conformación de, una proteína G. Esto tiene dos consecuencias:
- Primero, la subunidad alfa de la proteína G- pierde su PIB y se une a un GTP en su lugar.
- Segundo, la proteína G se descompone en la parte a unida a GTP y la parte ß.
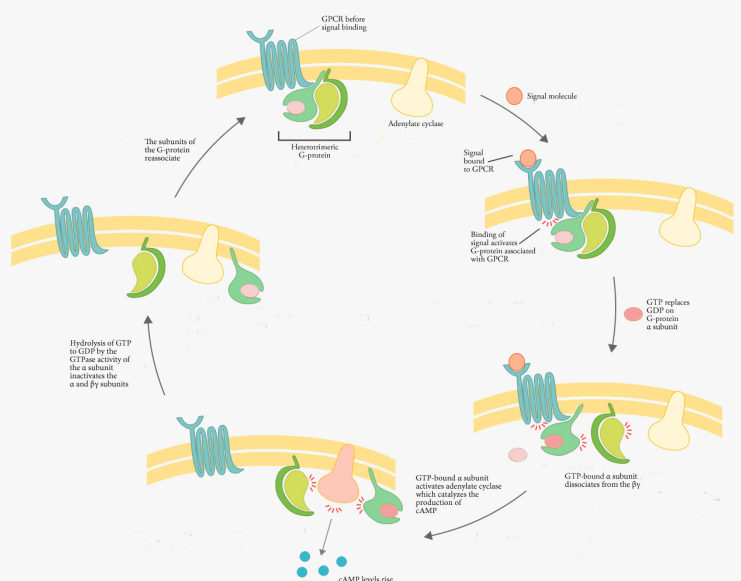
¿Qué sucede cuando las proteínas G interactúan con sus proteínas diana? Eso depende de cuál sea el objetivo. Las proteínas G interactúan con diferentes tipos de proteínas diana, de las cuales examinaremos dos categorías principales:
Canales Iónicos
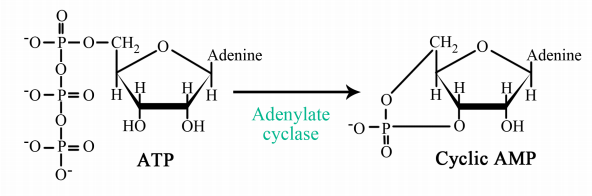
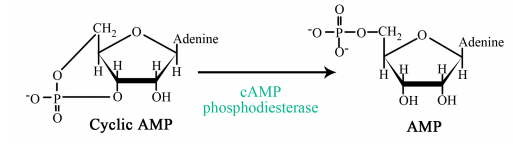
¿Cuál es el efecto de los niveles elevados de AMPc?
Por ejemplo, la unión de la epinefrina a su receptor en la superficie celular, activa, a través de la acción de las proteínas G, y la posterior activación de PKA, la fosforilación de la glucógeno fosforilasa. La activación resultante de la glucógeno fosforilasa conduce a la descomposición del glucógeno, liberando glucosa (en forma de glucosa-1-fosfato) para su uso por la célula. Los cambios en la expresión génica, asimismo, conducen a cambios en la célula al alterar la producción de proteínas particulares en respuesta a la señal.
Si bien los pasos descritos anteriormente parecen complicados, siguen el patrón simple esbozado al inicio de esta sección:
- Unión de la señal al receptor
- Varios pasos donde la señal se pasa a través de moléculas intermedias (proteínas G, adenilato ciclasa, AMPc y finalmente, PKA)
- Fosforilación de proteínas diana por la quinasa, dando lugar a cambios en la célula.
El IP3 y el DAG producidos por la fosfolipasa C activada trabajan juntos para activar una proteína quinasa. Primero, IP3 se difunde a la membrana del retículo endoplásmico donde se une a los canales de iones de calcio cerrados. Esto hace que los canales de calcio en la membrana del RE se abran y liberen grandes cantidades de calcio en el citoplasma desde la luz del RE, como se muestra en la siguiente figura.
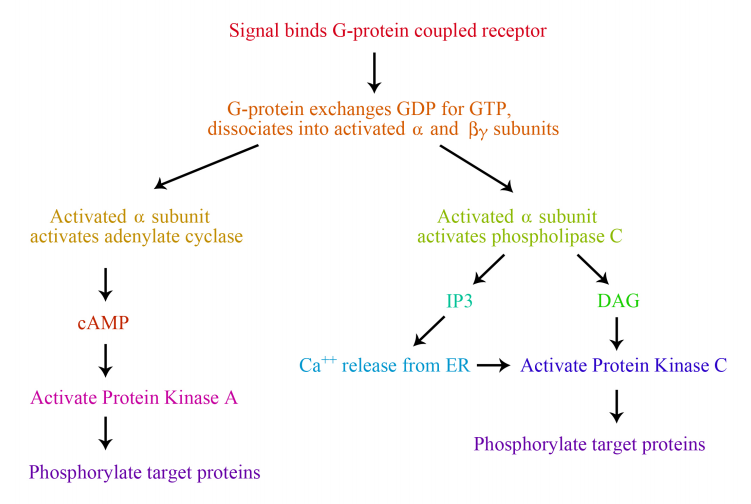
El incremento en la concentración de iones calcio citosólicos tiene diversos efectos, uno de los cuales es activar una proteína quinasa llamada proteína quinasa C (C para calcio), junto con el DAG elaborado en el paso anterior. Al igual que PKA, la Proteína quinasa C fosforila una variedad de proteínas en la célula, alterando su actividad y cambiando así el estado de la célula.
Las vías que conducen a la activación de PKC y PKA después de la unión de una señal a un GPCR se resumen en la Figura 8.4.12.


