7.9: Métodos
- Page ID
- 51530
Métodos basados en ensayos serológicos
A los efectos de realizar identificaciones más rápidas, o cuando las pruebas bioquímicas no son concluyentes, se recurre al uso de reacciones antígeno-anticuerpos. El inmunodiagnóstico es utilizado frecuentemente en el laboratorio clínico para la detección de patógenos específicos y de sus productos. Son usados para confirmar los casos de crecimiento y también para identificar los agentes etiológicos cuando no es posible el crecimiento en los medios convencionales.
Los métodos serológicos, implican la utilización de preparaciones de inmunoglobulinas específicas provenientes del suero o de un reactivo, y que pueden ser de gran utilidad en la identificación microbiana en muestras puras o en muestras biológicas. Cada uno de los métodos tiene su fundamento particular, pero en líneas generales, todos se basan en la reacción de un antígeno presente en el agente microbiano con su anticuerpo correspondiente. La solución que contiene los anticuerpos se denomina antisuero.
Los métodos serológicos son muy útiles en diversas situaciones:
- Si a través de un sistema miniaturizado basado en pruebas bioquímicas se determinó que la bacteria causante de la infección es un miembro del género Salmonella, utilizando una batería de antisueros contra el antígeno O presente en el lipopolisacárido de las bacterias Gram negativas, es posible identificar a las cepas de Salmonella aisladas hasta el nivel de serotipo (poli O, A, B, C, D, etc.).
- La inmunofluorescencia ha resultado ser sumamente útil en casos de infecciones de diferente origen. Puede utilizarse para la identificación del microorganismo aislado o presente en una muestra biológica. En el método directo se fija la muestra problema a una lámina y se pone en contacto con el antisuero específico marcado con una sustancia fluorescente (rodamina o fluoresceína). Una vez transcurrido el tiempo para que tenga lugar la reacción antígeno anticuerpo, se expone la lámina a la radiación ultravioleta para visualizar la reacción. También existe la técnica indirecta, donde en primer lugar se utiliza el anticuerpo específico no marcado y posteriormente se utiliza un anti anticuerpo marcado.
- En caso de una infección viral, los virus no pueden ser identificados mediante pruebas bioquímicas, pero si pueden ser identificados en diferentes fluidos biológicos utilizando inmunoensayos, tales como el ELISA (Enzyme-linked immunoabsorbent assay), el cual, a manera general, utiliza anticuerpos monoclonales específicos para el antígeno a identificar, marcados con una enzima.
Métodos basados en tipificación con fagos
La interacción entre un virus bacteriano (fago) y su célula bacteriana sensible es sumamente específica, ya que el proceso de adsorción se encuentra mediado por receptores específicos tanto en el virus como en la célula bacteriana. A una placa con medio de cultivo sólido inoculado con un cultivo puro de una determinada bacteria, se le añade una alícuota de un fago específico; éste puede ocasionar la lisis de las bacterias, hecho que se evidencia en el cultivo como zonas claras definidas, denominadas placas, que indican que hubo infección y lisis celular. El uso de fagos específicos permite identificar y subclasificar bacterias dentro de una misma especie.
Métodos basados en características genotípicas
La emergencia de la biología molecular ha dado lugar a nuevas herramientas moleculares que se han adaptado rápidamente al campo del diagnóstico microbiológico. Este nuevo enfoque utiliza factores genotípicos más que fenotípicos para identificar patógenos particulares.
De acuerdo a la técnica molecular utilizada podemos llegar a identificar un microorganismo en diferentes categorías:
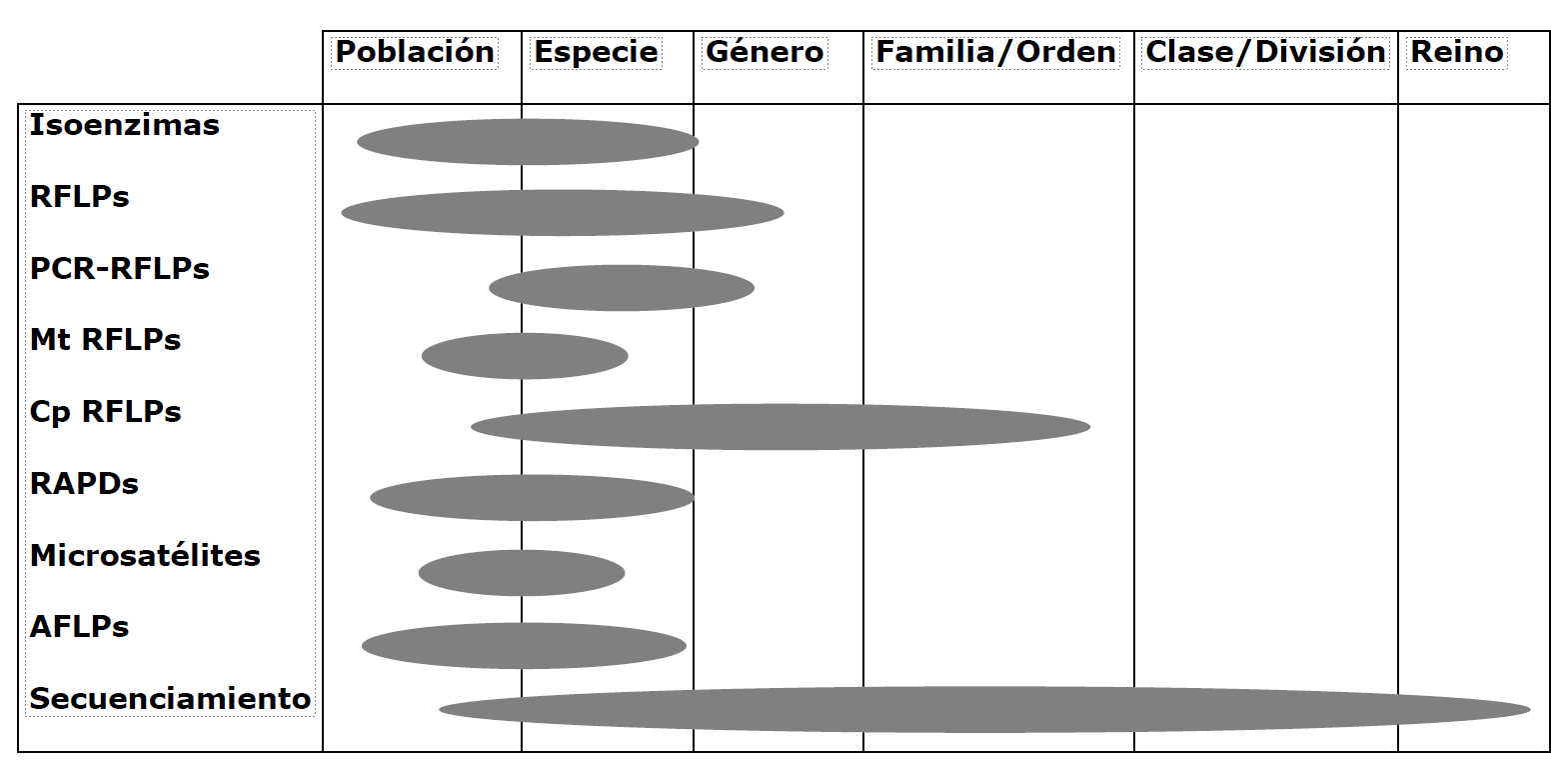
Los métodos más comúnmente utilizados son los siguientes:
Sondas de ácido nucleicos
Se fundamenta en complementariedad de bases de ácidos nucleicos. Se definen como secuencias de oligonucleotidos de ADN marcados, con un elemento radioactivo (32P,125I, 35S, 3H, 14C) o con una proteína unida a una enzima como la fosfatasa alcalina. Estas sondas se utilizan para la detección de una secuencia complementaria de ADN o ARN, presente en el agente que se desea identificar.
La muestra, que se supone que contiene un agente microbiano con una secuencia complementaria que se desea identificar, se coloca en un filtro, se trata de manera tal de liberar al ácido nucleico, se calienta para que las cadenas complementarias se separen. Posteriormente se añade la sonda, y se deja transcurrir un periodo para que tenga lugar la hibridación si existe la cadena complementaria. La unión de la sonda a la secuencia complementaria se detecta mediante la señal radiactiva que emite la sonda o mediante métodos enzimáticos.
Dos cepas cuyos ADNs muestran por lo menos el 70% de similitud bajo condiciones óptimas de hibridización y, menos del 5% de diferencia en sus Tm, generalmente se consideran miembros de la misma especie.
A continuación se presentan una lista con ejemplos de microorganismos que han sido detectados e identificados por medio de sondas de ADN:
- Bacterias Gram negativas: Neisseria gonorrhoeae, Campylobacter jejuni, Campylobacter coli, Haemophilus influenzae.
- Bacterias Gram positivas: Streptococcus Grupo A, Streptococcus Grupo B,
- Streptococcus pneumoniae, Staphylococcus aureus, Listeria monocytogenes.
- Micobacterias: Mycobacterium tuberculosis, M. Avium, M. Intracelulare.
- Hongos: Histoplasma capsulatum, Blastomyces dermatitis, Cryptococcus neoformans, Coccidiodes immitis.
Marcadores basados en la técnica de pcr (reacción en cadena de la polimerasa)
La reacción en cadena de la polimerasa (PCR) es un método poderoso con amplias aplicaciones en la biología molecular. Desde su desarrollo en 1985, la especificidad, sensibilidad y velocidad de ésta técnica ha permitido desarrollar muchos métodos para un amplio rango de áreas de investigación biológica y para toda clase de microorganismos. Dicha técnica tiene varias aplicaciones en microbiología, incluyendo genética y sistemática, ecología y microbiología de suelo, patología vegetal, microbiología médica, biotecnología y muchas otras.
Esta reacción enzimática permite la amplificación exponencial de fragmentos específicos de ADN mediante la síntesis de ADN in vitro. Para realizar una reacción de PCR es necesario contar con:
- ADN templado que contenga la región para ser amplificada,
- Primers de oligonucleótidos que flanqueen dicha región,
- ADN polimerasa termoestable aislada de Thermus aquaticus, llamada Taq polimerasa
- Nucleótidos trifosfatos (dNTPs).
Todos los componentes de la reacción se mezclan en un buffer apropiado que contiene iones Mg (MgCl2) y el procedimiento consiste de una sucesión de 3 pasos los cuales están determinados por las condiciones de temperatura: desnaturalización del templado (90 – 95° C), apareamiento de los primers (50-60° C) y extensión de la cadena de ADN (72° C) (Figura 7.2).
Los productos de la amplificación son separados por electroforesis de acuerdo a su tamaño en geles de agarosa o poliacrilamida si se requiere aumentar la resolución y, son visualizados por tinción con bromuro de etidio o plata, respectivamente.
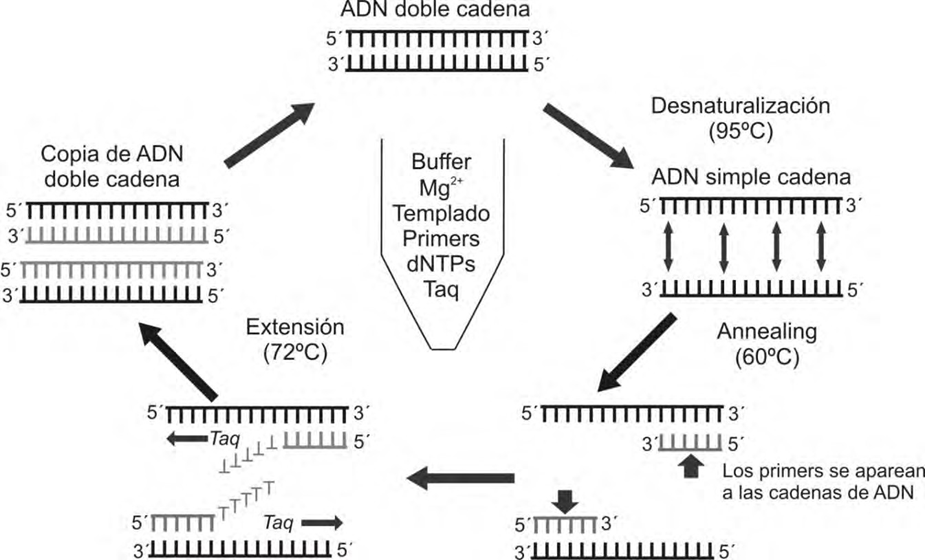
Métodos basados en PCR
RFLP (polimorfismo en la longitud de los fragmentos de restricción).
El polimorfismo en la longitud de los fragmentos de restricción se puede detectar analizando el ADN genómico digerido con endonucleasas de restricción por hibridización con sondas de ADN (“Southern blotting”). Las sondas usadas en el análisis de RFLPs se pueden generar a partir de ADN clonado, fragmentos de ADNc, ADN mitocondrial (ADNmt) o partir de segmentos específicos de ADN amplificados por PCR. De esta forma dependiendo de la sonda usada, dicha técnica se puede usar para analizar la variación del ADNmt, variación en la región del ADN ribosomal (ADNr) o variaciones en las secuencias repetidas o únicas.
RAPD (polimorfismo en el ADN amplificado al azar).
El análisis de RAPDs ha sido desarrollado para detectar polimorfismo entre organismos a pesar de la ausencia de información de la secuencia genómica, producir marcadores genéticos y construir mapas genéticos. El método se basa en la amplificación del ADN genómico por PCR usando primers cortos de secuencia arbitraria. Los ensayos RAPD-PCR han sido utilizados principalmente para definir variaciones intra y interespecíficas en las poblaciones microbianas.
AFLP (polimorfismo en la longitud de los fragmentos amplificados).
El análisis de AFLPs permite la diferenciación entre individuos, genotipos y cepas, identificación de especies con bandas conservadas, construir mapas genéticos, determinar diversidad genética en una población y además permite obtener un perfil complejo de marcadores. Los resultados son altamente reproducibles y es una técnica con resolución alta, rápida y eficiente.La técnica se basa en la amplificación selectiva de los fragmentos de restricción generados a partir de ADN genómico. El ADN es digerido con endonucleasas de restricción, generalmente una enzima de corte raro y otra de corte frecuente, tales como EcoRI y MseI, respectivamente. Los adaptadores de oligonucleótidos son ligados a los extremos del ADN genómico en los sitios de restricción específicos. La amplificación de dichos fragmentos se lleva acabo usando primers que incluyan las secuencias de los adaptadores, parte de la secuencia del sitio de restricción y entre uno y cinco nucleótidos adicionales elegidos al azar. Uno de los primers se marca radioactivamente con [g33P]ATP, por lo tanto, serán visualizados sólo los fragmentos amplificados que llevan dicha marca. El uso de extensiones selectivas tiene la función de reducir el número de fragmentos amplificados. Dichos fragmentos son analizados por electroforesis en geles de poliacrilamida.
Secuenciación
Las estructuras genómicas pueden ser directamente comparadas a través del secuenciamiento del ADN y el ARN, y aunque existen técnicas que permiten un rápido análisis de ambas moléculas, el secuenciamiento del ARN es el más extensamente usado en la taxonomía microbiana.
Una de las moléculas que ha recibido mayor atención es el ARNr 16S de la subunidad 30S de los ribosomas bacterianos. Esta molécula es ideal para estudios de evolución microbiana y filogenia ya que es esencial para una organela crítica encontrada en todas las bacterias. Además, su estructura cambia muy lentamente con el tiempo presumiblemente debido a su rol constante y crítico. Debido a que el ARNr presenta regiones constantes y regiones variables, a través de su secuencia es posible analizar tanto microorganismos estrechamente relacionados como distantes desde el punto de vista filogenético.
Además del secuenciamiento de los ácidos nucleicos también es importante el análisis de secuencias de proteínas, ya que los cambios en las secuencias de ambas moléculas con el tiempo son considerados cronómetros moleculares y resultan de importancia en la determinación de relaciones filogenéticas y evolución.


