7.5: Uso de la bioquímica para identificar microorganismos
- Page ID
- 54791
Objetivos de aprendizaje
- Describir ejemplos de productos de biosíntesis dentro de una célula que pueden detectarse para identificar bacterias
La identificación precisa de aislamientos bacterianos es esencial en un laboratorio de microbiología clínica porque los resultados a menudo informan decisiones sobre el tratamiento que afectan directamente los resultados de los pacientes. Por ejemplo, los casos de intoxicación alimentaria requieren una identificación precisa del agente causal para que los médicos puedan prescribir el tratamiento adecuado. De igual manera, es importante identificar con precisión el patógeno causante durante un brote de enfermedad para que se puedan emplear estrategias adecuadas para contener la epidemia.
Hay muchas maneras de detectar, caracterizar e identificar microorganismos. Algunos métodos se basan en características bioquímicas fenotípicas, mientras que otros utilizan identificación genotípica. Las características bioquímicas de una bacteria proporcionan muchos rasgos que son útiles para la clasificación e identificación. Analizar las capacidades nutricionales y metabólicas del aislado bacteriano es un enfoque común para determinar el género y la especie de la bacteria. Algunas de las vías metabólicas más importantes que utilizan las bacterias para sobrevivir serán discutidas en Metabolismo microbiano. En esta sección, discutiremos algunos métodos que utilizan características bioquímicas para identificar microorganismos.
Algunos microorganismos almacenan ciertos compuestos como gránulos dentro de su citoplasma, y el contenido de estos gránulos puede ser utilizado con fines de identificación. Por ejemplo, el poli-β-hidroxibutirato (PHB) es un compuesto de almacenamiento de carbono y energía que se encuentra en algunas bacterias no fluorescentes del género Pseudomonas. Diferentes especies dentro de este género pueden clasificarse por la presencia o ausencia de PHB y pigmentos fluorescentes. El patógeno humano P. aeruginosa y el patógeno vegetal P. syringae son dos ejemplos de especies fluorescentes de Pseudomonas que no acumulan gránulos de PHB.
Otros sistemas se basan en características bioquímicas para identificar microorganismos por sus reacciones bioquímicas, como la utilización de carbono y otras pruebas metabólicas. En pequeños entornos de laboratorio o en laboratorios docentes, esos ensayos se llevan a cabo utilizando un número limitado de tubos de ensayo. Sin embargo, sistemas más modernos, como el desarrollado por Biolog, Inc., se basan en paneles de reacciones bioquímicas realizadas simultáneamente y analizadas por software. El sistema de Biolog identifica las células en función de su capacidad para metabolizar ciertos bioquímicos y en sus propiedades fisiológicas, incluyendo el pH y la sensibilidad química. Utiliza todas las clases principales de bioquímicos en su análisis. Las identificaciones se pueden realizar manualmente o con los instrumentos semiautomatizados o completamente automatizados.
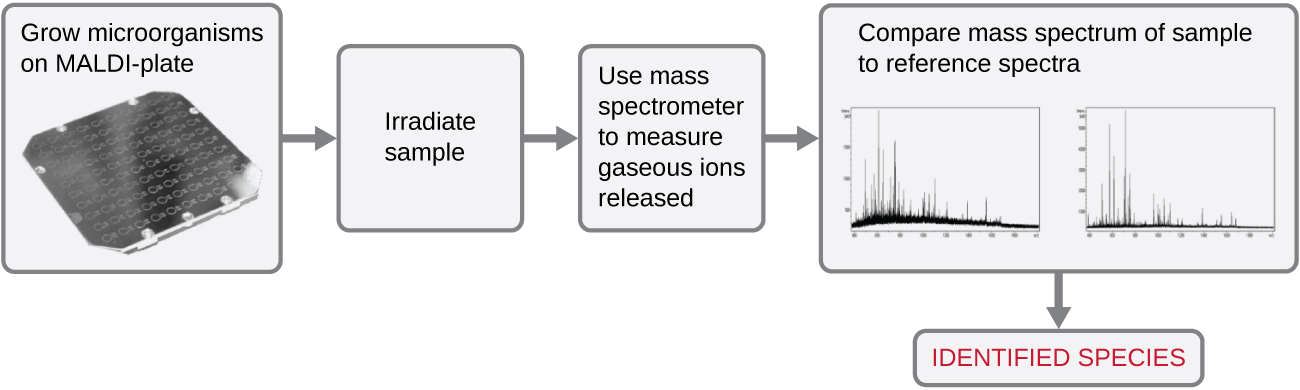
Otro sistema automatizado identifica microorganismos determinando el espectro de masas del espécimen y luego comparándolo con una base de datos que contiene espectros de masas conocidos para miles de microorganismos. Este método se basa en la espectrometría de masas de tiempo de vuelo de desorción/ionización láser asistida por matriz (MALDI-TOF) y utiliza placas MALDI desechables en las que se mezcla el microorganismo con un reactivo de matriz especializado (Figura\(\PageIndex{1}\)). La mezcla muestra/reactivo se irradia con un láser ultravioleta pulsado de alta intensidad, dando como resultado la expulsión de iones gaseosos generados a partir de los diversos constituyentes químicos del microorganismo. Estos iones gaseosos son recolectados y acelerados a través del espectrómetro de masas, con iones viajando a una velocidad determinada por su relación masa-carga (m/z), llegando así al detector en diferentes momentos. Una gráfica de señal detectora versus m/z produce un espectro de masas para el organismo que está relacionado únicamente con su composición bioquímica. La comparación del espectro de masas con una biblioteca de espectros de referencia obtenidos a partir de análisis idénticos de microorganismos conocidos permite la identificación del microbio desconocido.
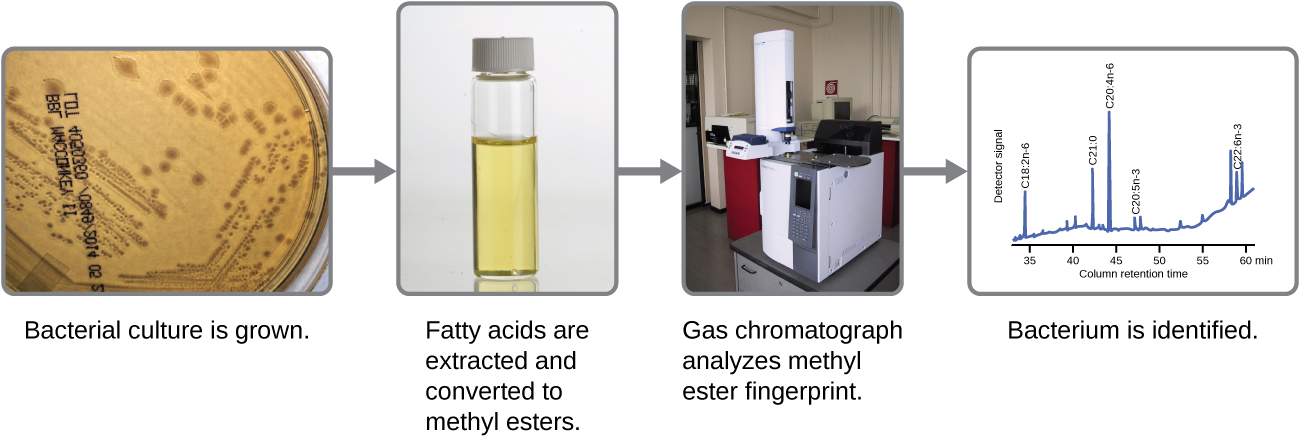
Los microbios también se pueden identificar midiendo sus perfiles lipídicos únicos. Como hemos aprendido, los ácidos grasos de los lípidos pueden variar en longitud de cadena, presencia o ausencia de dobles enlaces, y número de dobles enlaces, grupos hidroxilo, ramas y anillos. Para identificar un microbio por su composición lipídica, se analizan los ácidos grasos presentes en sus membranas. Un análisis bioquímico común utilizado para este propósito es una técnica utilizada en laboratorios clínicos, de salud pública y de alimentos. Se basa en la detección de diferencias únicas en los ácidos grasos y se llama análisis de éster metílico de ácidos grasos (FAME). En un análisis FAME, los ácidos grasos se extraen de las membranas de microorganismos, se alteran químicamente para formar ésteres metílicos volátiles, y se analizan por cromatografía de gases (GC). El cromatograma GC resultante se compara con cromatogramas de referencia en una base de datos que contiene datos de miles de aislados bacterianos para identificar el microorganismo desconocido (Figura\(\PageIndex{2}\)).
Un método relacionado para la identificación de microorganismos se llama análisis de ácidos grasos derivados de fosfolípidos (PLFA). Las membranas están compuestas principalmente por fosfolípidos, los cuales pueden saponificarse (hidrolizarse con álcali) para liberar los ácidos grasos. La mezcla de ácidos grasos resultante se somete luego a análisis FAME, y los perfiles lipídicos medidos se pueden comparar con los de microorganismos conocidos para identificar el microorganismo desconocido.
La identificación bacteriana también puede basarse en las proteínas producidas bajo condiciones de crecimiento específicas dentro del cuerpo humano. Este tipo de procedimientos de identificación se denominan análisis proteómico. Para realizar el análisis proteómico, las proteínas del patógeno se separan primero por cromatografía líquida de alta presión (HPLC), y las fracciones recolectadas se digieren para producir fragmentos peptídicos más pequeños. Estos péptidos se identifican por espectrometría de masas y se comparan con los de microorganismos conocidos para identificar el microorganismo desconocido en el espécimen original.
Los microorganismos también pueden ser identificados por los carbohidratos unidos a las proteínas (glicoproteínas) en la membrana plasmática o pared celular. Los anticuerpos y otras proteínas de unión a carbohidratos pueden unirse a carbohidratos específicos en las superficies celulares, haciendo que las células se agrupen. Se realizan pruebas serológicas (por ejemplo, las pruebas de grupos Lancefield, que se utilizan para la identificación de especies de Streptococcus) para detectar los carbohidratos únicos ubicados en la superficie de la célula.
Foco Clínico: Resolución
Penny dejó de usar su nuevo protector solar y le aplicó la crema de corticosteroides a su sarpullido según las indicaciones. No obstante, después de varios días, su sarpullido no había mejorado y en realidad parecía estar empeorando. Ella hizo una cita de seguimiento con su médico, quien observó un sarpullido rojo lleno de baches y ampollas llenas de pus alrededor de los folículos pilosos (Figura\(\PageIndex{3}\)). El sarpullido se concentró especialmente en zonas que habrían sido cubiertas por un traje de baño. Después de algunas preguntas, Penny le dijo al médico que recientemente había asistido a una fiesta en la piscina y pasó algún tiempo en un jacuzzi. A la luz de esta nueva información, el médico sospechó un caso de erupción en la bañera de hidromasaje, infección frecuentemente causada por la bacteria Pseudomonas aeruginosa, un patógeno oportunista que puede prosperar en jacuzzis y piscinas, especialmente cuando el agua no está suficientemente clorada. P. aeruginosa es la misma bacteria que se asocia con infecciones en los pulmones de pacientes con fibrosis quística.
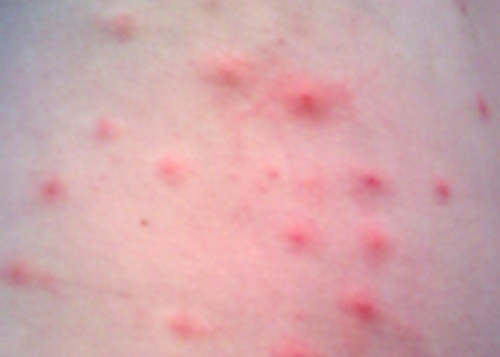
El médico recolectó un ejemplar del sarpullido de Penny para ser enviado al laboratorio de microbiología clínica. Se realizaron pruebas confirmatorias para distinguir P. aeruginosa de los patógenos entéricos que también pueden estar presentes en el agua de piscinas y bañeras de hidromasaje. La prueba incluyó la producción del pigmento azul-verde piocianina sobre agar cetrimida y crecimiento a 42 °C; la cetrimida es un agente selectivo que inhibe el crecimiento de otras especies de flora microbiana y además potencia la producción de pigmentos piocianina y fluoresceína de P. aeruginosa, los cuales son característica azul-verde y amarillo-verde, respectivamente.
Las pruebas confirmaron la presencia de P. aeruginosa en la muestra de piel de Penny, pero el médico decidió no recetarle un antibiótico. Aunque P. aeruginosa es una bacteria, las especies de Pseudomonas son generalmente resistentes a muchos antibióticos. Por suerte, las infecciones de la piel como la de Penny suelen ser autolimitantes; la erupción suele durar alrededor de 2 semanas y se resuelve por sí sola, con o sin tratamiento médico. El médico le aconsejó a Penny que lo esperara y siguiera usando la crema de corticosteroides. La crema no matará a la P. aeruginosa en la piel de Penny, pero debería calmar su sarpullido y minimizar el picor suprimiendo la respuesta inflamatoria de su cuerpo a la bacteria.
Conceptos clave y resumen
- La identificación precisa de bacterias es esencial en un laboratorio clínico para el diagnóstico y manejo de epidemias, pandemias e intoxicaciones alimentarias causadas por brotes bacterianos.
- La identificación fenotípica de microorganismos implica el uso de rasgos observables, incluyendo perfiles de componentes estructurales como lípidos, productos biosintéticos como azúcares o aminoácidos, o compuestos de almacenamiento como poli-β-hidroxibutirato.
- Un microbio desconocido puede ser identificado a partir del espectro de masas único producido cuando se analiza mediante espectrometría de masas de tiempo de vuelo de desorción/ionización láser asistida por matriz (MALDI-TOF).
- Los microbios se pueden identificar determinando sus composiciones lipídicas, utilizando ésteres metílicos de ácidos grasos (FAME) o análisis de ácidos grasos derivados de fosfolípidos (PLFA).
- El análisis proteómico, el estudio de todas las proteínas acumuladas de un organismo, también se puede utilizar para la identificación bacteriana.
- Las glicoproteínas en la membrana plasmática o las estructuras de la pared celular pueden unirse a lectinas o anticuerpos y pueden usarse para la identificación.


