3.7: Cinética enzimática con Spectrovis
- Page ID
- 56340
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Cinética enzimática de la peroxidasa de nabo
El peróxido de hidrógeno (H 2 O 2) es un agente oxidante fuerte que puede dañar las células y se forma como un subproducto del consumo de oxígeno. Afortunadamente, las células aeróbicas contienen peroxidasas que descomponen el peróxido en agua y oxígeno. Esta enzima reduce el peróxido de hidrógeno a H 2 O al oxidar un compuesto orgánico (AH 2 -> A).
Este ejercicio utiliza extracto de nabo como fuente de peroxidasa. Este extracto de nabo requiere una fuente de electrones (un agente reductor) para que se produzca la reacción. En este caso, se utiliza un compuesto orgánico incoloro llamado guayacol. El guayacol se oxida en el proceso de conversión del peróxido y se vuelve marrón. La actividad enzimática se puede rastrear usando un espectrofotómetro para medir la cantidad de marrón que se forma.
Configuración y Calibración de LabQuest con SpectroVis Plus
- Configura la cubeta para que sirva como una pieza en blanco.
- Agregar 10 gotas 0.02% de peróxido de hidrógeno.
- Agrega 5 gotas 0.2% guayacol.
- Agregar 20 gotas de tampón pH 7.
- Añadir 10 gotas de extracción tamponada.
- Conecte un LabQuest2 a un SpectroVis plus e inicie el programa de recolección de datos.
- Calibrar el SpectroVis.
- Cambiar modo.
- Seleccione “Basado en el tiempo” en el menú desplegable y presione OK cuando termine.
- Cambiar modo.
- Tasa: 0.5 muestras/s
- Intervalo: 2 s/muestra
- Duración: 200 s
2. Presione el botón Reproducir (flecha verde).
1. Realiza calentamiento por 90 segundos.
1. Elija “Calibración de acabado”.
2. Presiona “OK”.
2. Presiona el botón “Stop” (Cuadrado Rojo).
4. Seleccione “Icono de medidor” (arriba a la izquierda, la mayor parte de la pantalla).
- Presione el área roja grande que muestra la lectura de absorbancia de corriente.
- Elija “Cambiar longitud de onda”.
- Establezca la longitud de onda a 500 nm.
Efecto del pH sobre la Actividad Peroxidasa
- Configura la cubeta para pH 3.
- Agregar 10 gotas 0.02% de peróxido de hidrógeno.
- Agrega 5 gotas 0.2% guayacol.
- Agregar 20 gotas de tampón pH 3.
- Añadir 10 gotas de extracción de nabo por último.
- Invierta rápidamente la cubeta y coloque la cubeta en el Spectrovis Plus.
- Presione el botón de reproducción para comenzar a grabar datos (elija “descartar datos” si se le pide).
- Después de 200s, retire SpectroVis del USB.
- Inserte una unidad flash USB y espere 1 minuto.
- Presione Archivo → Exportar → elija el icono USB y renombra el archivo y agregue “.csv” al final del nombre del archivo.
- Presiona OK.
- Repetir secuencialmente el experimento intercambiando el tampón de pH con pH 5, 7, 10.

Efecto de la Temperatura sobre la Actividad Peroxidasa
- Configurar la cubeta a 0ºC
- Agregar 10 gotas 0.02% de peróxido de hidrógeno (incubado a 0ºC durante 10 minutos).
- Agrega 5 gotas 0.2% guayacol.
- Añadir 20 gotas de tampón de extracción a 0ºC.
- Añadir 10 gotas de extracción de nabo (incubadas a 0ºC durante 10 minutos) por último.
- Invierta rápidamente la cubeta y coloque la cubeta en el Spectrovis Plus.
- Presione el botón de reproducción para comenzar a grabar datos (elija “descartar datos” si se le pide).
- Después de 200s, retire SpectroVis del USB.
- Inserte una unidad flash USB y espere 1 minuto.
- Presione Archivo → Exportar → elija el icono USB y renombra el archivo y agregue “.csv” al final del nombre del archivo.
- Presiona OK.
- Repetir secuencialmente el experimento intercambiando los tampones almacenados a 20ºC, 40ºC, 60ºC.
Efecto de la concentración de sustrato sobre la actividad peroxidasa
- Configura la cubeta para 1X sustrato.
- Agregar 10 gotas 0.02% de peróxido de hidrógeno.
- Agrega 5 gotas 0.2% guayacol.
- Agregar 20 gotas de tampón de extracción.
- Añadir 10 gotas de extracción de nabo por último.
- Invierta rápidamente la cubeta y coloque la cubeta en el Spectrovis Plus.
- Presione el botón de reproducción para comenzar a grabar datos (elija “descartar datos” si se le pide).
- Después de 200s, retire SpectroVis del USB.
- Inserte una unidad flash USB y espere 1 minuto.
- Presione Archivo → Exportar → elija el icono USB y renombra el archivo y agregue “.csv” al final del nombre del archivo.
- Presiona OK.
- Repetir secuencialmente el experimento con diferentes cantidades de tampón y peróxido:
- Repita el experimento con sustrato 0.2X y exporte los datos a una unidad USB.
- Repita el experimento con sustrato 2X y exporte los datos a una unidad USB.
- Repita el experimento con sustrato 3X y exporte los datos a una unidad USB.

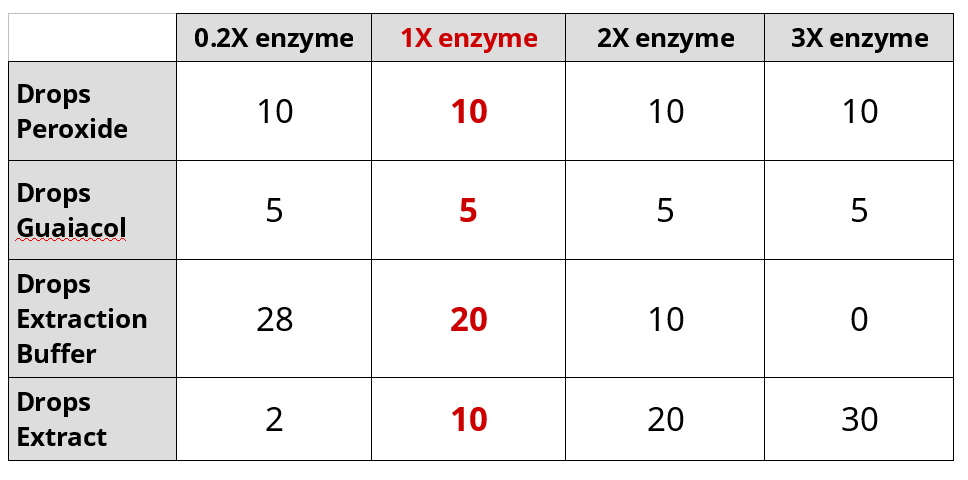
Efecto de la concentración de enzima sobre la actividad peroxidasa
- Configura la cubeta para 1X enzima.
- Agregar 10 gotas 0.02% de peróxido de hidrógeno.
- Agrega 5 gotas 0.2% guayacol.
- Agregar 20 gotas de tampón de extracción.
- Añadir 10 gotas de extracción de nabo por último.
- Invierta rápidamente la cubeta y coloque la cubeta en el Spectrovis Plus.
- Presione el botón de reproducción para comenzar a grabar datos (elija “descartar datos” si se le pide).
- Después de 200s, retire SpectroVis del USB.
- Inserte una unidad flash USB y espere 1 minuto.
- Presione Archivo → Exportar → elija el icono USB y renombra el archivo y agregue “.csv” al final del nombre del archivo.
- Presiona OK.
- Repetir secuencialmente el experimento con diferentes cantidades de tampón y extraer:
- Realice el experimento con enzima 1X y exporte los datos a una unidad USB.
- Repita el experimento con enzima 0.2X y exporte los datos a una unidad USB.
- Repita el experimento con enzima 2X y exporte los datos a una unidad USB.
- Repita el experimento con enzima 3X y exporte los datos a una unidad USB.