9.2: Sabor Amargo (Actividad)
- Page ID
- 56294
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)La genética me deja mal sabor de boca... o no
Algunas de nuestras preferencias personales surgen de la forma en que fuimos educados. La cultura juega un papel en nuestros gustos y disgustos. Así mismo, nuestras experiencias juegan un papel en la forma en que respondemos a ciertos estímulos. Otro factor importante que juega un papel en nuestras preferencias viene cableado en nuestro genoma. El ADN en nuestras células es el manual de instrucciones para quienes somos. Estamos programados para buscar cosas de valores nutritivos para adquirir materias primas como carbohidratos, proteínas y lípidos. En nuestra búsqueda de compuestos nutritivos, hemos aprendido a evitar cosas que no saben bien. Las cosas amargas tienden a asociarse con compuestos tóxicos en la naturaleza. Al comer un alimento por primera vez, las moléculas golpean nuestra lengua y estimulan múltiples sensaciones: dulce, agrio, salado, salado y amargo. A estos múltiples tipos de sabor se le atribuye una familia diversa de receptores que se unen a las moléculas que resultan en nuestra percepción de estas sensaciones. Algo amargo podría hacernos aprender a evitar este alimento en el futuro.
Un tipo de receptor amargo detecta la presencia de una sustancia química llamada feniltiocarbamida (PTC). Este químico se asemeja químicamente a compuestos tóxicos que se encuentran en las plantas pero no es tóxico. La capacidad de probar PTC proviene de un gen llamado TAS2R38. Este gen codifica una proteína que en nuestras lenguas comunica el amargor de este químico. Hay dos alelos comunes de este gen con al menos cinco variantes poco comunes más. Dentro de las dos formas comunes, un polimorfismo de un solo nucleótido (SNP) es responsable de cambiar un aminoácido en el receptor. Es esta diferencia de un aminoácido lo que da como resultado la capacidad del receptor para responder o no responder a PTC. Heredamos una copia del gen de nuestro padre y una copia de nuestra madre. Cómo se formaron los gametos de nuestros padres y qué alelos recibimos durante el evento de fertilización determina cómo respondemos a este químico. Debido a que cada uno de nosotros tiene 2 copias de este gen, podemos utilizar la genética mendeliana simple para entender qué alelo es dominante o recesivo.
- Coloca un trozo de papel “Control” en la lengua e indica si hay sabor.
- Colocar un trozo de papel “PTC” en la lengua e indicar si hay un sabor y la severidad del sabor.
- Llena la tabla de la clase para identificar cuántos no catadores, catadores o súper catadores hay.
- Indica si crees que el rasgo es dominante o recesivo (capacidad de probar o no saborear).
- Asignar un alelo descriptor para el dominante (una letra mayúscula) o el recesivo (una letra minúscula) y dibuja un cuadrado Punnet para la generación F 2 de 2 padres heterocigotos.
- Compara el recuento de la clase de catadores y no catadores en la clase y discute con tu instructor si hay un dominio claro de este rasgo.
Tabla: Contador de Cata PTC
| Fenotipos | Número | % Total |
| Catadores PTC (Dominantes o Recesivos) | ||
| PTC No Catadores (Dominantes o Recesivos) | ||
| Total |
Preguntas:
1. ¿Cómo explicas la presencia de los que no pueden probar el PTC, los que pueden probarlo y los que realmente no soportan el sabor del mismo?
2. Este químico no es tóxico y no existe en la naturaleza. ¿Crees que hay una presión selectiva que confiere una ventaja a quienes la prueban?
Ejercicio: Codificar amargura:
Previo a este ejercicio, revisar el Dogma Central.
La secuencia codificante completa de TAS2R38 tiene 1,002 bases (334 aminoácidos) de longitud. A continuación se muestra un segmento del gen donde ocurre el SNP (en rojo). La variante 1 es la versión del gen que codifica la capacidad de probar PTC. La variante 2 es la versión del gen que es incapaz de unirse a PTC. Esta mutación SNP se denomina mutación sin sentido porque cambia el aminoácido. Algunas mutaciones provocan la inserción de un codón de parada prematuro. Esta mutación sin sentido da como resultado una proteína truncada y puede ser desastrosa para la función. Ya sabemos que la simple sustitución de un nucleótido se traduce en un cambio en un aminoácido y determina la capacidad de probar PTC. Imagínese si faltara un gran grupo de aminoácidos de la proteína.
Con la información de la hebra plantilla (“Complemento”):
- Escribe la secuencia de la cadena codificante.
- Escribir la secuencia del ARNm
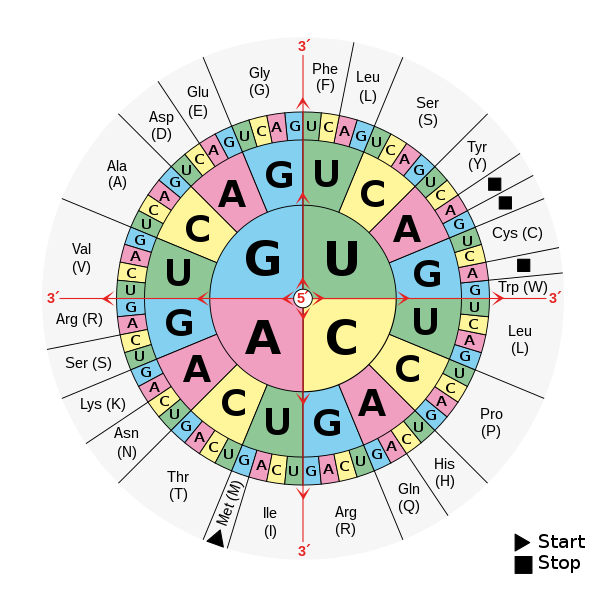
- Usar la Tabla de Código Genético para traducir la secuencia de aminoácidos
Variante 1 hebra
codificante: 5′-
Complemento: 3′-TTC TCC GTC CGT GAC TCG-5′
ARNm: 5′-
Aminoácido:
Variante 2 hebra
codificante: 5′-
Complemento: 3′-TTC TCC GTC GGT GAC TCG-5′
ARNm: 5′-
Aminoácido:
PCR Genotipado del receptor PTC TAS2R38:
- 5'-CCTTCGTTTTTGGTGAATTTTTGGGATGTAGTGAAGAGGCGG-3' (Imprimación hacia adelante)
- 5′-AGGTTGGCTTGGTTTGCAATCATC-3′ (Imprimación inversa)
- PCR de las muestras de ADN extraídas de las células de las mejillas usando las Perlas de PCR.
- Vierta 2% de agarosa en el aparato de fundición en refrigerador.
- Se necesitan hacer 2 geles por clase → 100ml de TBE con agarosa 2g.
- Agregue 5μl de solución segura SYBR en la agarosa fundida antes de colar.
- Coloca 2 juegos de peines en el gel → en un extremo y en el medio.
- Digerir el producto de PCR con Hae III.
- Retirar 10μl de producto de PCR en un tubo nuevo.
- Agrega 1μl de enzima Hae III en el tubo.
- Incubar por 10 minutos a 37°C.
- Cargar gel con escalera de ADN, digerido y no digerido.
- la muestra no digerida es de la PCR original.
- Ejecute el gel a 120V durante 20 minutos.
- Visualice en el transiluminador UV.
Detección de SNP:
El cebador más largo termina con la secuencia “GG”. Ambos alelos en este locus se amplificarán igualmente bien con este conjunto de cebadores, sin embargo, un alelo tendrá la secuencia “GGGC” y otro “GGCC”. “GGCC” es el sitio de restricción para la enzima Hae III. La digestión de este ADN amplificado será digerible para un alelo y producirá un fragmento de ADN del tamaño del cebador grande (44 pb) así como el resto del amplicón. Debido a esta diferencia en el perfil de digestión del amplicón, podemos identificar los 2 alelos en este locus.
Preguntas de análisis:
- ¿Cuál es el tamaño del producto de PCR?
- El cebador largo es de 44 pb. Si el amplicón del alelo digiere, ¿cuáles son los tamaños de los fragmentos que se esperan después de Hae III?
- ¿Qué alelo es el que se puede identificar a través de la digestión con Hae III?
- Utilice los resultados de la prueba de papel PTC.
- Algunos carriles contienen 3 bandas en lugar de 1 o 2. ¿Puedes explicar esto?


