4.1: Introducción: El panorama general
- Page ID
- 52930
Ya comenzamos a examinar Genética del Desarrollo y probamos la complejidad de las interacciones genéticas. Por ejemplo, el gen de Drosophila Bicoid se transcribe en ARNm en las células maternas y se bombea al embrión. Allí su expresión está regulada por señales de localización en el ARNm, así como la degradación y localización de la proteína. La proteína bicoide en sí misma actúa como regulador tanto de la transcripción como de la traducción de genes aguas abajo. El efecto final es un embrión con diferentes genes expresados a lo largo del eje A/P.
Ahora daremos un paso atrás y veremos un panorama más amplio de la Genética del Desarrollo, desde el punto de vista de las vías de señalización. Una vía de señalización permite a las células comunicarse con su entorno externo. En Biología del Desarrollo, esto suele ser interacciones célula-célula. Este tipo de interacciones son increíblemente importantes porque cada célula necesita seguir su propia trayectoria de desarrollo en coordinación con todas las células que la rodean. Por ejemplo, imagina un brote de miembro de mamífero en crecimiento. Cada célula en la yema de la extremidad necesita saber si está en el pulgar o en el lado meñique de la extremidad y qué tan cerca del cuerpo está. No sólo eso, también necesita saber cuánto tiempo ha avanzado el desarrollo. Imagínese si una célula osteogénica comenzó a formar matriz ósea temprano en el desarrollo de la extremidad. No sufriría suficientes divisiones celulares para hacer el número correcto de osteoblastos, dando lugar a muy poco hueso y a un animal con una extremidad malformada.
El kit de herramientas genéticas
(Un PDF introductorio se puede encontrar aquí: Redescubriendo la biología www.learner.org/courses/biology/pdf/7_gendev.pdf)
Uno de los principales hallazgos de Evo-Devo es que los organismos comparten un conjunto común de genes para construir sus cuerpos muy diversos. En particular, los animales comparten un conjunto de genes de “plan corporal” que realizan funciones similares en el desarrollo. Mezclar y emparejar diferentes patrones de expresión génica a través de tejidos y tipos celulares parece ser el mecanismo principal para generar los cuerpos únicos que vemos en cada filo animal. Las características evolutivas novedosas (como espinas, plumas, corazones con cámaras, tentáculos, etc.) parecen depender de la reutilización de genes de herramientas en nuevos momentos y lugares en desarrollo (ver Formas infinitas para más detalles). A continuación, comenzaremos a echar un vistazo a la genética detrás de este kit de herramientas genéticas: ¿cómo interactúan estos genes entre sí?
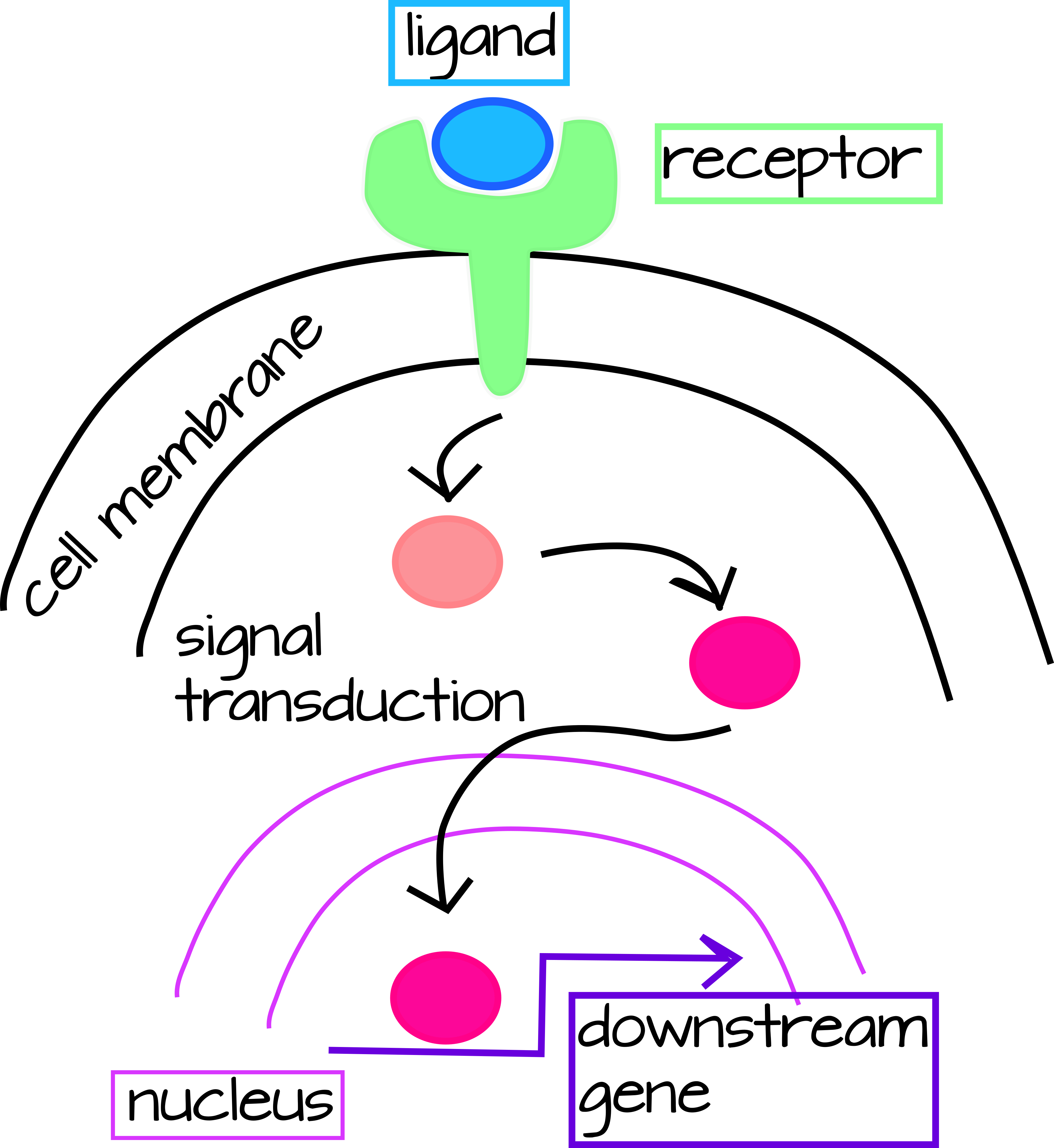
En Evo/Devo, hay dos formas principales en las que podemos pensar sobre las interacciones genéticas, pero como están interconectadas por lo que consideraremos ambas simultáneamente. Una de ellas son las vías de transducción de señales, las cuales tienen una señal externa (en Biología del Desarrollo esta suele ser una proteína secretada) que suele ser recibida por un receptor de membrana celular que solo expresan ciertas células. Cuando el ligando (proteína secretada) y el receptor se unen, esto desencadena una cascada de modificaciones proteicas que conducen a la activación transcripcional de genes aguas abajo (Figura 1).
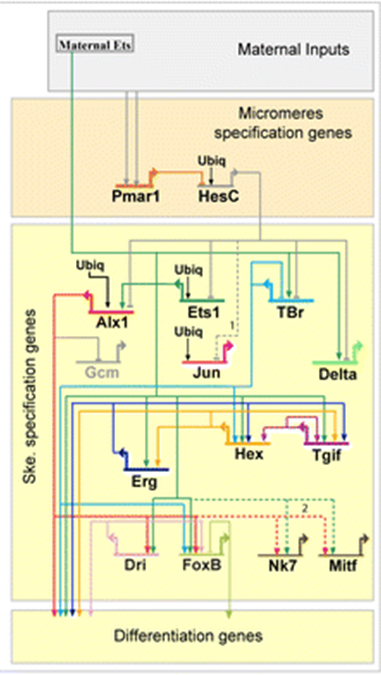
La segunda forma en que Evo/Devo piensa sobre la señalización es Redes Reguladoras de Genes (GRN). Esto incluye la vía de transducción de señales y sus efectos aguas abajo sobre la expresión génica. Si subimos la expresión de un gen, ¿a qué otros genes afecta? Los GRN a menudo se representan como diagramas de cableado con flechas y barras que muestran el efecto de las proteínas en la expresión de genes aguas abajo (Figura 2).
 |
.png) |
| Figura 1: Vía genérica de transducción de señales. Un ligando extracelular se une a un receptor unido a membrana desencadenando la activación de una cascada de tranducción de señal en el citoplasma. Esta cascada puede ser simple (como en el caso de la ruta Notch a continuación) o muy compleja requiriendo múltiples pasos y tipos de entrada. El paso final de la cascada de transducción es la translocación de un factor de transcripción citoplásmico al núcleo donde activará o inhibirá la transcripción de un gen aguas abajo. Un archivo svg editable de esta figura se puede descargar en https://scholarlycommons.pacific.edu/open-images/18/ | Figura 2: Diagrama de cableado de la red reguladora génica parcial para la especificación del mesodermo formador de esqueleto en el erizo de mar, Strongylocentrotus purpuratus. Cada gen está representado por una línea horizontal unida a una flecha del mismo color único. La línea horizontal indica la región cis-reguladora del gen y la flecha indica la transcripción de la región codificante del ARNm. Las líneas que emanan de la región codificante de ARNm indican cómo el producto proteico del gen regula otros genes aguas abajo. Las entradas en las regiones reguladoras cis por los productos proteicos de otros genes pueden ser activantes (flecha) o inhibidoras (bar). “Ubiq” son activadores desconocidos expresados en todas las células. Figura de Dylus et al 1 Publicado bajo licencia CC BY 4.0. La figura ha sido recortada del original. |
El panorama general: Los destinos celulares se restringen progresivamente a medida que avanza el desarrollo
En el transcurso del desarrollo deben especificarse todos los órganos, tejidos y tipos de células. En los animales esto se logra mediante la restricción repetida del destino 2. Por ejemplo, el embrión totipotente se somete a varias rondas de división para formar una blástula (bola de células). Algunas de estas células tomarán un destino mesodérmico. En este punto, pueden convertirse en cualquier tipo de célula mesodérmica. En algunos animales se han realizado experimentos para demostrar que una célula mesodérmica genérica puede ser inducida por sus vecinos para convertirse en una gran variedad de tipos de células mesodérmicas 3. Sin embargo, con el tiempo, estas células mesodérmicas se dividen, experimentan cambios de forma, migran y se diferencian. A medida que reciben señales de su entorno (principalmente las células que los rodean) se diferencian en tipos celulares más específicos. Por ejemplo, la Figura 3 muestra una célula mesodérmica que se diferencia en células musculares, óseas y sanguíneas. A medida que avanza el tiempo, diferentes células progenitoras musculares (miogénicas) se diferenciarán en diferentes tipos de músculo, por ejemplo liso y estriado. Cada una de estas decisiones de destino celular depende del patrón de expresión génica en la célula, que depende de la historia de la célula y sus vecinos.

Llamamos a este tipo de diferenciación progresiva “jerárquica” con tipos celulares más genéricos (como el mesodermo, Figura 3) en la parte superior de la jerarquía y tipos celulares más específicos (como los neutrófilos) en la parte inferior 4. A medida que avanza el desarrollo, bajamos por la jerarquía. El desarrollo comienza la especificación del eje, estos ejes pronto se leen en regiones, por ejemplo, cabeza, tronco/abdomen y cola. Dentro de estas diferentes regiones se especifican los tejidos, primero a nivel de capas germinales y posteriormente a niveles más específicos. Por ejemplo, el ectodermo se divide en neuroectodermo y ectodermo epidérmico dependiendo de la ubicación del tejido. A continuación, se especifican los tipos de células dentro de los tejidos. Por ejemplo, el neuroectodermo puede convertirse en neuronas o células gliales. Ambos tipos de células posteriormente se diferencian en los muchos tipos específicos de neuronas y glía. En este punto, las células se diferencian más o menos terminalmente: se especifican sus destinos adultos y no pueden regresar a formas pluripotentes. Sin embargo, la mayoría de las células siguen siendo receptivas al ambiente y pueden responder a las señales ambientales incluso una vez que se diferencian terminalmente. Nos referimos a esto como plasticidad celular.
El GRN en la Figura 2 refleja esta jerarquía, con genes de acción temprana en la parte superior especificando tejidos más generales y genes de actuación posterior en la parte inferior especificando tipos celulares específicos. Sin embargo, es importante señalar que los genes que actúan en estos diferentes niveles jerárquicos se reutilizan en otros niveles de la jerarquía y en otros tejidos en desarrollo. De hecho, las partes centrales de los GRN y las vías de transducción de señales se reutilizan a lo largo del desarrollo y tienen diferentes resultados dependiendo de su contexto.


