4.2: Señalización y restricción del destino: comunicación célula-célula - Dentro de la célula
- Page ID
- 52953
Comunicación célula-célula: vías de transducción de señales
Las vías de transducción de señales acoplan señales externas a cambios en la expresión génica dentro de una célula. Es decir, una señal externa desencadena una cascada de cambios bioquímicos en la célula dando como resultado una transcripción mayor o menor de un conjunto de genes. Menos de una docena de vías principales de transducción de señales suelen regular el desarrollo animal 5,6. Aquí, nos centraremos en cuatro de estos que llevan el nombre de su receptor o ligando: Notch, Hedgehog (Hh), TGF-b (también llamado Dpp o BMP) y Wnt (Figura 4). Estas vías de transducción de señales actúan como interruptores para las Redes Reguladoras de Genes (discutidas más adelante) que encienden usando diferentes mecanismos. Dos factores principales afectan los tipos de patrones y procesos de especificación para los que sirve una Vía de Transducción de Señal en particular.
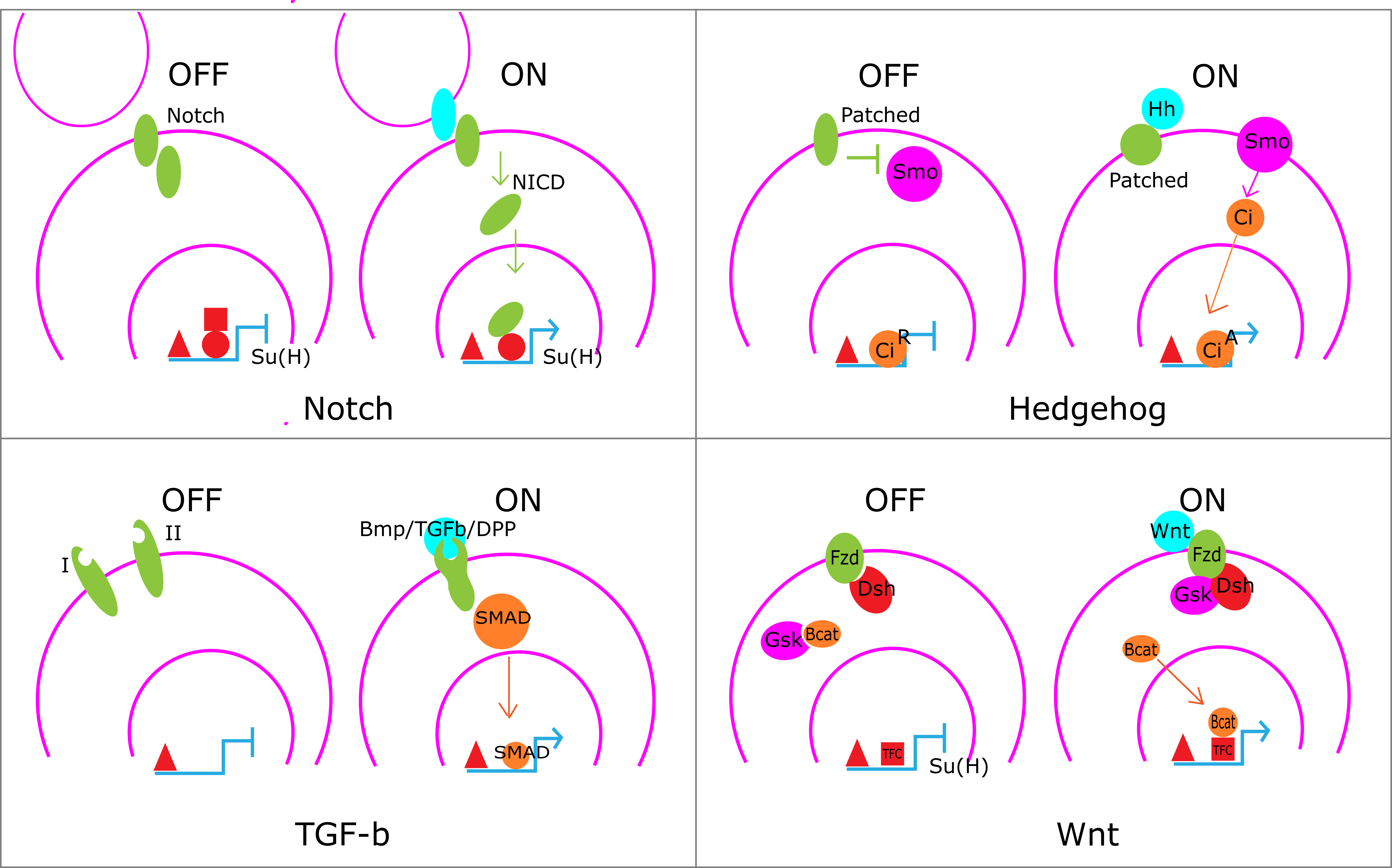
Primero, ¿cómo se comunica la señal? ¿Utiliza un ligando de largo alcance (señalización paracrina), requiere contacto célula a célula (señalización yuxtacrina), o el ligando actúa sobre la misma célula de la que fue secretada (señalización autocrina)? Las vías paracrinas de largo alcance, como TGF-b y Wnt, son excelentes para formar gradientes a través de un tejido grande y a menudo se usan como morfógenos tempranos que diseñan ejes corporales o múltiples tipos de células a través de un tejido. Las vías paracrinas de corto alcance, como Hh, a menudo actúan como morfógenos a menor escala, ajustando patrones de regionalización. Otros factores modifican el rango de señalización de estos ligandos, por ejemplo la cantidad de ligando secretado, la neutralización por proteínas de la matriz extracelular, el número de células sensibles y si las células sensibles están expresando inhibidores o coactivadores de los genes diana 6. Las señales yuxtacrinas, al igual que la vía Notch, implican la asociación de dos receptores unidos a membrana. En el caso de Notch, este es típicamente un ligando de clase Delta/Serrate/Lag-12 (DSL) y una proteína Notch glicosilada de longitud completa. La vía Notch se usa a menudo en las decisiones de encendido/apagado de destino celular, famoso en la inhibición lateral donde una señal de “encendido” en una célula desencadena una señal de “apagado” en todas las células circundantes.
El segundo factor importante que rige la utilidad de una vía de transducción de señales es su regulación. La mayoría de las vías pueden regularse hasta cierto punto por sus genes diana aguas abajo, ya sea a través de retroalimentación negativa o positiva. En retroalimentación negativa, los genes diana aguas abajo eventualmente apagan la vía. Por ejemplo, una vía de señalización podría aumentar la transcripción de un inhibidor de la ruta. En retroalimentación positiva, los objetivos descendentes mantienen el camino. Por ejemplo, una vía de señalización podría aumentar la transcripción de su propio receptor. Por lo tanto, la retroalimentación negativa es buena para promover una señal transitoria de una sola vez, mientras que la retroalimentación positiva convierte una señal transitoria en una decisión permanente de destino celular Las vías también pueden ser reguladas entre sí y su salida puede ser modificada por factores de transcripción locales, que pueden diferir entre los tipos de células. Este tema se trata más extensamente en Tres hábitos de vías de señalización altamente efectivas. de Borolo y Posakony.
Dentro de la célula: Redes reguladoras génicas
A principios de la década de 2000, Eric Davidson e Isabelle Peter escribieron una serie de artículos proponiendo un marco filosófico para comprender las Redes Reguladoras de Genes (GRN). En 2011 escribieron un trabajo seminal sobre la evolución de estas redes. Definían tres tipos básicos de interacción genética central que se utilizan para especificar los tipos de células:
- núcleos: son interacciones evolutivamente inflexibles que especifican una parte del cuerpo.
- baterías: Estas están involucradas en la diferenciación celular o tisular y son más flexibles evolutivamente que los granos.
- plug-ins: se trata de pequeños subcircuitos que se utilizan en muchos contextos de desarrollo diferentes 7.
Como madre de los entusiastas de Lego, utilizo kits de Lego como analogía para entender esto. Un núcleo serían piezas especializadas que van juntas, como un par de ruedas y un eje. Especifican una función particular y son inflexibles en su interacción - una rueda debe ser encajada a cada lado del eje para que sea útil. Una “batería” es más flexible pero todavía se usa generalmente para un conjunto similar de propósitos. Un ejemplo de esto sería una batería literal en un kit de Lego: se usa para impulsar el movimiento pero es flexible ya que puede alimentar muchos tipos de construcciones. Un plug-in es similar a los ladrillos estándar, estos se pueden utilizar para construir cualquier objeto cuboide que sugiera tu imaginación Para hacer construcciones elegantes, sin embargo, también requieres granos y baterías.
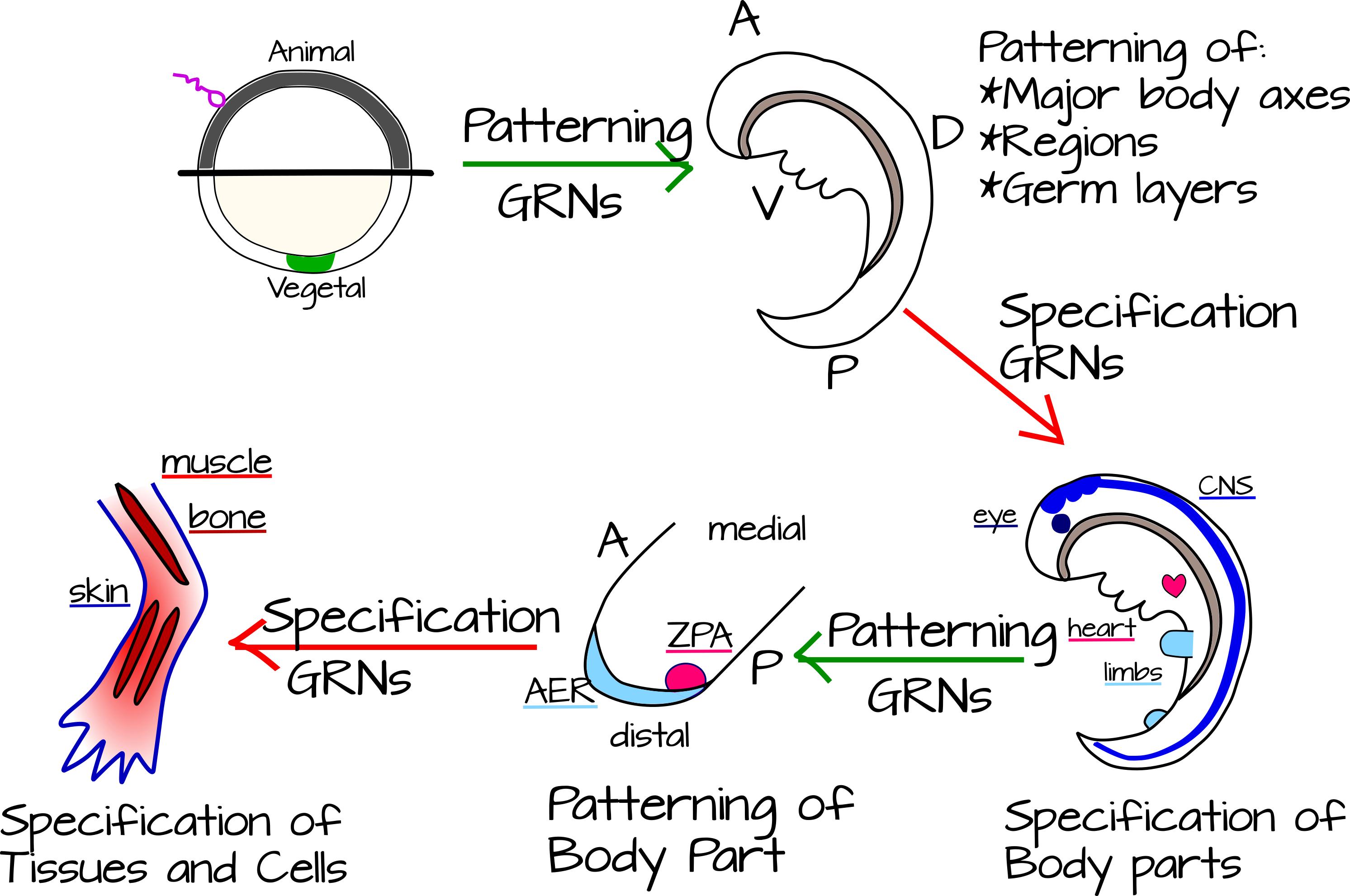
¿Cómo vinculamos este tipo de interacciones con un embrión en desarrollo? Peter y Davidson conciben el desarrollo animal como alternando pasos de modelado y especificación 7. El modelado consiste en dividir un cuerpo o un conjunto de tejidos en partes más pequeñas. La especificación es una elección de destino para la célula o tejido que fue modelado. En esta concepción, los ejes corporales y las capas germinales se definen primero usando redes genéticas de patrones corporales. A continuación, se especifica cada sección del cuerpo: neuroectodermo, intestino, arcos faríngeos (en cordados), sistema circulatorio, etc. Luego, ocurre otra ronda de patrones de estas unidades especificadas, cada parte del cuerpo ahora se modula a lo largo de sus propios ejes. Por ejemplo, las puntas de los dedos vs un codo o la anterior vs. la posterior del corazón (Figura 5). 8