5.2: Del banco de investigación a la base de datos
- Page ID
- 53815
La última fuente de información en las bases de datos es la comunidad investigadora, que envía sus datos experimentales a bases de datos primarias. Las bases de datos primarias piden a los investigadores información básica sobre su envío. Se acepta un registro que cumpla con los estándares de la base de datos y se le asigna un número de acceso único que permanecerá permanentemente asociado con el registro. Cada base de datos tiene su propio sistema de números de acceso, lo que permite identificar la fuente de un registro a partir de su número de acceso. Una vez que un registro es aceptado en una base de datos primaria, los curadores profesionales toman el relevo. Los curadores son científicos profesionales que agregan valor
a un registro al proporcionar vínculos entre registros en diferentes bases de datos. Los curadores también organizan
la información de formas novedosas para generar bases de datos derivadas. Las bases de datos derivadas, como las bases de datos de organismos, a menudo están diseñadas para satisfacer las necesidades de comunidades de investigación particulares. La base de datos del genoma de Saccharomyces (www.yeastgenome.org), por ejemplo, vincula información sobre genes con información sobre las proteínas codificadas por los genes y experimentos genéticos que exploran la función génica. En este curso, estaremos utilizando bases de datos tanto primarias como derivadas.
La figura de la siguiente página resume el flujo de información desde el banquillo hasta las bases de datos. La información en bases de datos se origina en experimentos. Cuando los investigadores completan un experimento, analizan sus datos y compilan los resultados para su comunicación a la comunidad investigadora. Estas comunicaciones pueden adoptar diversas formas.
PubMed indexa publicaciones en ciencias biomédicas
Los investigadores suelen escribir un artículo para su publicación en una revista científica. Los revisores
de la revista juzgan si los resultados son precisos y representan un hallazgo novedoso que avanzará en el campo. Estos artículos revisados por pares son aceptados por la revista, que luego publica los resultados en forma impresa y/o en línea. Como parte del proceso de publicación, las revistas biomédicas envían automáticamente la cita y el resumen del artículo a PubMed, una base de datos bibliográfica mantenida por el NCBI. A las entradas de PubMed se les asigna un número de acceso PMID. Los números PMID se asignan secuencialmente y los números han crecido bastante. ¡PubMed actualmente contiene más de 23 millones de registros! Los usuarios de PubMed pueden restringir sus búsquedas a campos como título, autor, revista, año de publicación, reseñas y más. La usabilidad de PubMed sigue creciendo. Los usuarios pueden
pegar citas en un portapapeles, guardar sus búsquedas y organizar fuentes RSS cuando los nuevos resultados de búsqueda ingresan a PubMed. Los estudiantes de ciencias biomédicas necesitan ser competentes en el uso de PubMed. Puede acceder a PubMed en pubmed.gov o a través del portal de base de datos de la Biblioteca BC. Una ventaja de usar el portal de la biblioteca es que podrás usar el potente botón “Encuéntralo” de la biblioteca para acceder a los artículos reales.
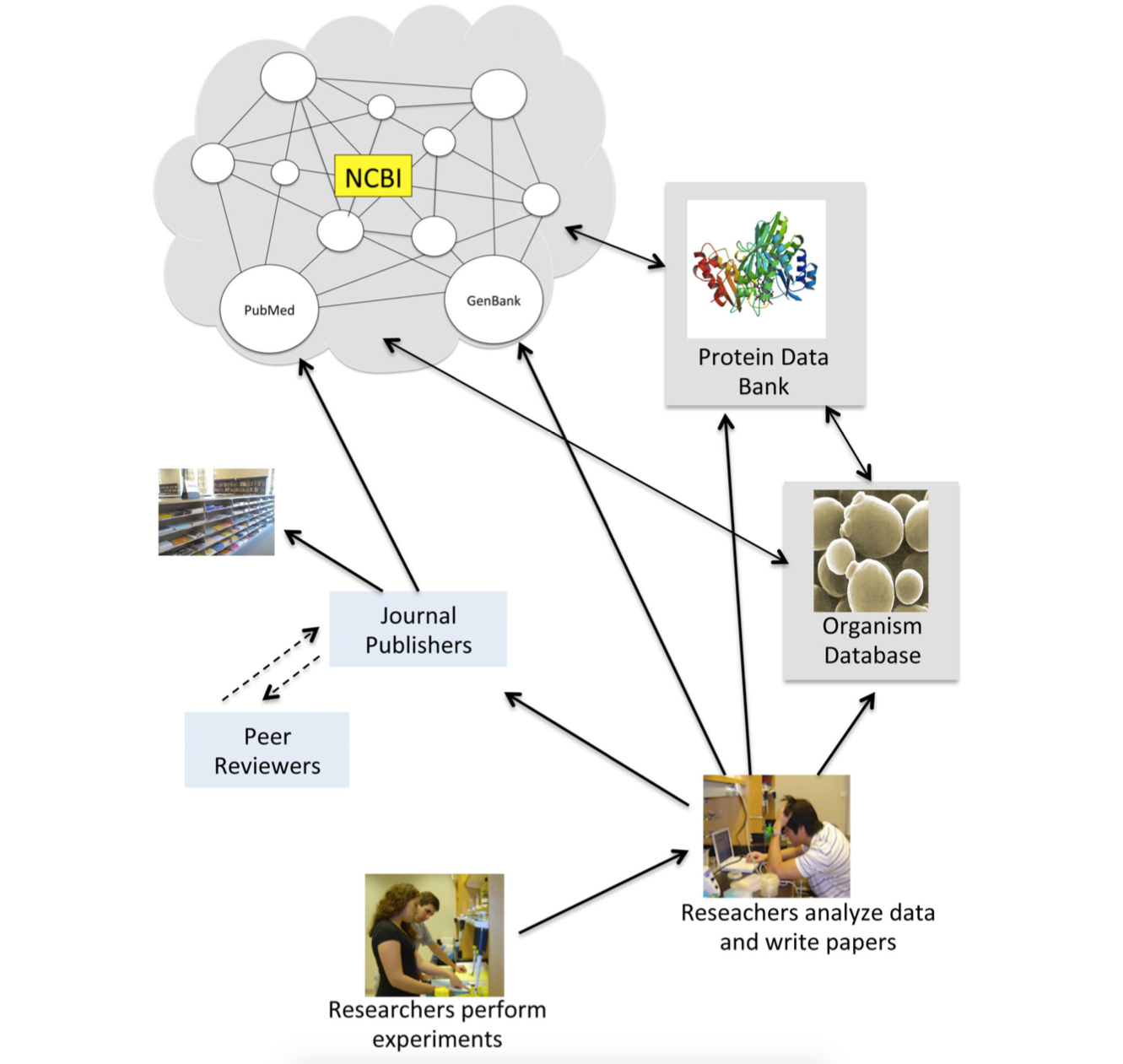
Flujo de información de experimentos a bases de datos. Los investigadores analizan sus datos y preparan manuscritos para su publicación. Las citas de revistas se envían automáticamente a PubMed. Los investigadores también envían datos a bases de datos más especializadas e interconectadas.
Investigadores envían datos experimentales a bases de datos de investigación especializadas
Dependiendo del experimento, los investigadores enviarán sus datos a una serie de bases de datos diferentes. Consideremos el ejemplo hipotético de un investigador que ha aislado una variante novedosa de un gen MET a partir de una cepa silvestre de S. cerevisiae con un sofisticado tamiz genético. El investigador ha secuenciado el gen, clonado el gen en un plásmido de sobreexpresión bacteriana y cristalizado la proteína sobreexpresada, que posee propiedades reguladoras únicas. El investigador está preparando un manuscrito sobre los experimentos. En preparación para el envío del manuscrito (los revisores del manuscrito querrán ver los números de acceso), el investigador planea enviar datos a tres bases de datos diferentes: una base de datos de nucleótidos, una base de datos de estructura y una base de datos de organismos.
Si nuestro investigador está trabajando en una institución en Estados Unidos, probablemente enviará la secuencia de nucleótidos al GenBank de NCBI, una subdivisión de la base de datos de Nucleotides más grande. GenBank se fundó en 1982, cuando acababan de desarrollarse métodos de secuenciación de ADN y los investigadores individuales secuenciaban manualmente un gen a la vez. La tasa de presentaciones de GenBank ha aumentado en ritmo con los avances en las tecnologías de secuenciación de ADN. Hoy en día, GenBank acepta envíos generados computacionalmente de grandes proyectos de secuenciación
, así como presentaciones de investigadores individuales. GenBank actualmente contiene más de 300 millones de registros de secuencias, incluyendo genomas completos, genes individuales, transcritos, plásmidos y más. No es sorprendente que haya una redundancia considerable en los registros de GenBank. Para eliminar esta redundancia, los curadores del NCBI construyeron la base de datos derivada RefSeq. RefSeq considera que las secuencias genómicas completas producidas por proyectos de secuenciación son las secuencias de referencia para un organismo. RefSeq actualmente contiene registros no redundantes de genoma, transcritos y secuencias de proteínas de más de 36,000 organismos.
El investigador en nuestro ejemplo hipotético también querrá presentar las coordenadas atómicas y los modelos estructurales para la proteína cristalizada al Banco de Datos de Proteínas (PDB). El PDB forma parte de un consorcio internacional que acepta datos para proteínas y ácidos nucleicos. La gran mayoría de los registros de PDB se han obtenido por difracción de rayos X, aunque la base de datos también acepta modelos obtenidos con resonancia magnética nuclear (RMN), microscopía electrónica y otras técnicas. El número de entradas en las bases de datos de PDB es órdenes de magnitud menor que el número de proteínas predichas en GenBank, reflejando las dificultades inherentes a la determinación de estructuras de macromoléculas. PDB ofrece herramientas para visualizar macromoléculas en tres dimensiones, lo que permite a los investigadores sondear interacciones de aminoácidos que son importantes para la función de las proteínas.
Finalmente, nuestro investigador querrá enviar datos sobre el fenotipo del nuevo mutante e información sobre su regulación a la Base de Datos del Genoma de Saccharomyces (SGD). El SGD
sirve como un recurso central para la comunidad de investigación de S. cerevisiae, que ahora te incluye a ti. El SGD es solo una de las muchas bases de datos específicas del organismo. Existen bases de datos similares para otros organismos modelo como la mosca de la fruta Drosophila, la planta Arabidopsis thaliana, pez cebra y más. Además de proporcionar información, estas bases de datos especializadas también facilitan la investigación al proporcionar enlaces a recursos importantes como colecciones de cepas y plásmidos.


