8.1: Antecedentes
- Page ID
- 54172
Los geles de agarosa proporcionan un método sencillo para analizar preparaciones de ADN. Las moléculas de ADN comparten la misma relación carga/masa, lo que imparte propiedades electroforéticas similares a las moléculas de ADN lineales de longitudes ampliamente variables. Utilizaremos “molécula” para referirnos a una pieza lineal de ADN, pero hay que tener en cuenta que una sola “molécula” de ADN puede contener las secuencias de múltiples genes o partes de genes.
Los geles de agarosa son matrices porosas
La agarosa es un polisacárido purificado a partir de algas rojas o algas marinas. La agarosa está más altamente purificada (¡y significativamente más cara!) que el agar, el cual se obtiene de la misma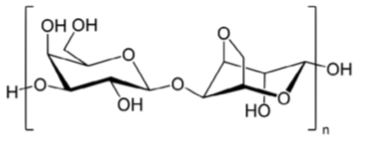 alga marina. Las moléculas de agarosa son polímeros largos y lineales de un disacárido repetido D-galactosa y 3,6-anhidro-α-L-galactopiranosa (derecha). Una molécula de agarosa típica contiene más de cien subunidades. La agarosa utilizada para electroforesis ha sido altamente purificada. El proceso de purificación elimina contaminantes que interferirían con las enzimas utilizadas en la clonación molecular, como las endonucleasas de restricción. El proceso también genera una preparación de agarosa con propiedades electroforéticas deseables y mínima fluorescencia de fondo, lo cual es importante para visualizar moléculas de ADN.
alga marina. Las moléculas de agarosa son polímeros largos y lineales de un disacárido repetido D-galactosa y 3,6-anhidro-α-L-galactopiranosa (derecha). Una molécula de agarosa típica contiene más de cien subunidades. La agarosa utilizada para electroforesis ha sido altamente purificada. El proceso de purificación elimina contaminantes que interferirían con las enzimas utilizadas en la clonación molecular, como las endonucleasas de restricción. El proceso también genera una preparación de agarosa con propiedades electroforéticas deseables y mínima fluorescencia de fondo, lo cual es importante para visualizar moléculas de ADN.
Las moléculas de agarosa son capaces de formar geles con tamaños de poro relativamente definidos debido a
las propiedades químicas de las moléculas de agarosa. La agarosa demuestra histéresis: su temperatura de fusión es mayor que su temperatura de gelificación. Las moléculas de agarosa se disuelven a aproximadamente 90°C, formando bobinas aleatorias en solución. Los geles se forman cuando la temperatura cae a aproximadamente 40°C. A medida que se forma el gel, las moléculas de agarosa primero se ensamblan en fibras helicoidales, que luego se agregan para formar redes de hélices superenrolladas estabilizadas por enlaces de hidrógeno. Los tamaños de los poros, que típicamente oscilan entre 50 y 200 nm, dependen de la concentración de agarosa. A medida que aumenta la concentración de agarosa, el diámetro promedio del poro disminuye.
Varios factores afectan la migración del ADN a través de geles de agarosa
Debido a la carga negativa de los residuos de fosfato en la cadena principal del ADN, las moléculas de ADN se mueven hacia el polo positivo (ánodo) del aparato de electroforesis. La tasa de migración real de las moléculas de ADN en un experimento particular se ve afectada por múltiples factores.
Algunos de estos factores son intrínsecos a las moléculas de ADN, mientras que otros factores se relacionan con las condiciones electroforéticas. Los factores intrínsecos incluyen tanto la longitud como la conformación de
las moléculas de ADN que se están analizando. Dentro de un cierto rango de tamaño dictado por las condiciones del gel, la tasa de migración de las moléculas lineales de ADN es inversamente proporcional al log10 (número de pares de bases). La migración de moléculas de ADN más estructuradas, como los plásmidos superenrollados, es mucho menos predecible. Las tasas de migración de estos ADN más altamente estructurados están influenciadas por la densidad de bobinas, la presencia de mellas y otras características estructurales.
Las tasas de migración de las moléculas de ADN en geles de agarosa también se ven afectadas por la composición del gel. La tasa de migración de una molécula de ADN disminuye a medida que aumenta la concentración de agarosa en el gel. Los investigadores suelen ajustar la concentración de agarosa para optimizar la resolución de las moléculas de ADN dentro de un rango de tamaño particular. Los dos tampones comúnmente utilizados en laboratorios, TAE (Tris: acetato: EDTA) y TBE (Tris: borato: EDTA), también afectan las tasas de electroforesis.
Debido a esta variabilidad inherente, los investigadores SIEMPRE incluyen un carril de patrones de ADN con tamaños conocidos en el mismo gel que las muestras que se analizan. Es importante destacar que estos estándares necesitan tener una estructura similar (por ejemplo, lineal o superenrollada) y ser sometidos a las mismas modificaciones químicas que las muestras de ADN que se analizan.
Se utilizan agentes intercalantes fluorescentes para visualizar moléculas de ADN en geles
Los ácidos nucleicos son visualizados por colorantes fluorescentes que se unen fuertemente al ADN. Los colorantes son agentes intercalantes que se insertan en la hélice de ADN y en regiones estructuradas de ácidos nucleicos monocatenarios. La fluorescencia de estos colorantes aumenta en un orden de magnitud cuando se unen a los ácidos nucleicos, por lo que la fluorescencia de fondo en los geles de agarosa suele ser baja. En esta clase, utilizaremos bromuro de etidio (EtBr) para visualizar fragmentos de ADN. ETBr absorbe la luz en el rango ultravioleta (UV) y emite luz naranja. Los geles se ven con transiluminadores especiales que contienen luces UV. El EtBr es un compuesto sensible a la luz, por lo que las existencias se almacenan en la oscuridad.
Se utilizan estándares para estimar los tamaños de las moléculas de ADN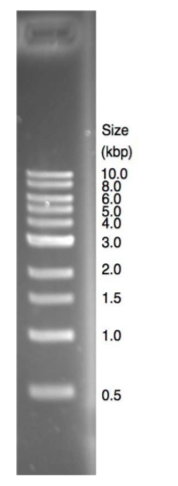
La figura de la derecha muestra un carril de un gel de agarosa al 1% teñido con EtBR que contiene patrones de tamaño de ADN. La escalera de 1 kb que se muestra en este gel estudiantil
es una mezcla patentada de fragmentos de ADN lineales que varían en tamaño de 0.5 a 10 kilobases (kb). La intensidad de tinción de las bandas en geles de agarosa refleja la cantidad de ADN en la banda, porque EtBr se intercala de manera bastante uniforme a lo largo de la longitud de las moléculas de ADN lineales. Como puede ver, esta mezcla en particular contiene cantidades similares de cada fragmento de ADN, con la excepción del fragmento de 3 kb, que tiene ~2.5 más ADN que los otros fragmentos. La mayor intensidad del fragmento de 3 kb sirve como marcador de orientación útil en situaciones en las que fragmentos más pequeños podrían haberse escurrido del extremo del gel o cuando algunos marcadores no están bien resueltos entre sí. (¡Estas son ocurrencias comunes!)
Tenga en cuenta que las moléculas más cortas en el gel están más bien separadas entre sí que las moléculas más largas. Los productos de PCR que analizará en este laboratorio están en su mayoría en el rango de 400-1000 pb. Para incrementar la resolución de estas moléculas, utilizaremos geles de agarosa al 1.25%. (Esto reducirá la resolución de moléculas de ADN más grandes). Obsérvese también que las bandas más pequeñas aparecen más borrosas en el gel teñido, debido a que se han visto más afectadas por la difusión aleatoria ya que han migrado a través de la red de polímeros de agarosa.


