16.3: Micro-informe 2- PCR de colonias de levadura
- Page ID
- 53477
En este reporte, describirás los resultados del experimento PCR de colonias de levadura que diseñaste para identificar las deleciones met en tus tres cepas YMP. Los productos de su reacción PCR se separarán en geles de agarosa similares a éste preparado por alumnos de otra clase.
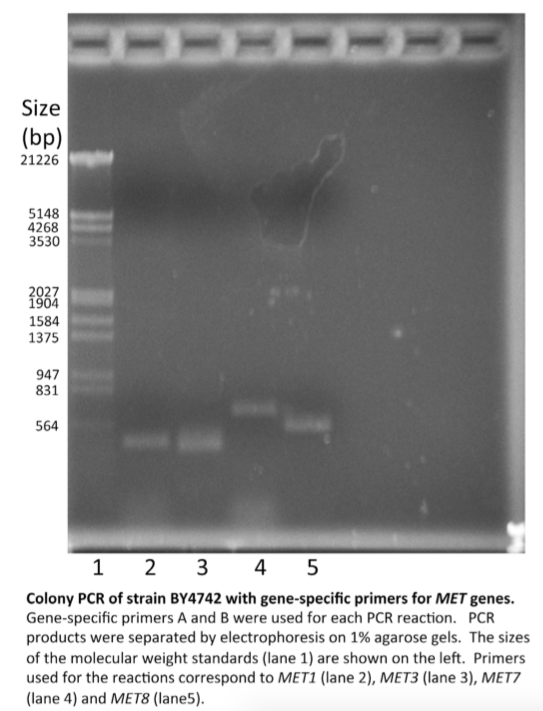
Tenga en cuenta las siguientes características de esta figura:
- El gel se muestra con los pozos de carga en la parte superior.
- Los carriles están claramente marcados. En esta figura, los carriles están numerados. Las cepas e iniciadores utilizados para cada reacción están descritas en la leyenda. Alternativamente, los carriles podrían etiquetarse con los nombres de cepas e iniciadores, cuidando que permanezcan legibles. Las conclusiones NO están incluidas en la leyenda.
- Se indican los tamaños de los marcadores. (Tus marcadores serán diferentes.)
El lector deberá consultar el M&M para obtener detalles adicionales sobre los genotipos de cepas y el procedimiento de PCR.
Lineamientos específicos micro-informe 2 seguir.
Materiales y Métodos: Proporcione información sobre las cepas y cebadores específicos que utilizó, así como los procedimientos para PCR y electroforesis en gel de agarosa.
Cepas: Ver micro-reporte 1 lineamientos.
Cebadores: En una publicación, los autores suelen incluir las secuencias de sus cebadores de PCR en el texto o en una tabla. Debe incluir los nombres de los genes MET, pero NO es necesario incluir las secuencias reales, ya que éstas están listadas en el Capítulo 7 del manual. No obstante, asegúrese de citar el manual. Utilice la terminología correcta cuando se refiera a los cebadores. Los cebadores GSP-A son cebadores sentido que corresponden a secuencias flanqueantes 5' de los genes MET. Los cebadores GSP-B son cebadores antisentido que son complementarios a las secuencias ORF.
PCR: Los métodos de PCR se describen en el manual. “Las reacciones de PCR se realizaron como described (ref.)” es adecuado. Si realizó alguna modificación, sin embargo, es necesario describirlas por ejemplo...” con las siguientes modificaciones”...
Electroforesis en gel de agarosa: Los productos de la reacción PCR se analizaron en geles de agarosa que se tiñeron con bromuro de etidio. Describa las condiciones que utilizó para analizar los productos de PCR. Incluya la concentración de agarosa, el nombre del búfer y el voltaje. En términos muy generales describen este análisis. Refiérase a los lectores al manual para obtener más detalles.
Resultados y Discusión: La figura con tu gel es el punto focal de esta sección. Describa brevemente su diseño experimental y la lógica que subyace a su elección de cebadores.
Al comentar sus resultados, recuerde que las bandas que ve en el gel de agarosa son productos de PCR. El tamaño es muy importante en la interpretación de los resultados de un experimento de PCR. Mientras te preparabas para el experimento, calculaste los tamaños de los productos de PCR que confirmarían o no confirmarían tus predicciones. ¿Tus resultados experimentales confirmaron tus predicciones?
Si tus resultados no confirmaron tus predicciones, debes considerar si las predicciones fueron incorrectas O si las reacciones no funcionaron. ¿Viste bandas en algún carril? Si es así, los re- agentes de PCR probablemente no tuvieron la culpa. ¿Qué más pudo haber pasado?
Sería útil una sola tabla de datos resumida con los tamaños pronosticados y observados de los productos de PCR. Los tamaños pronosticados deben ser muy precisos, ya que se basan en la secuencia de ge- nome real, la cual tiene resolución nucleotídica. Sus tamaños estimados son mucho menos precisos. Es posible que pueda determinar si un producto de PCR es más pequeño que otro, pero solo podrá colocar los productos dentro de un cierto rango de tamaño.


