6.1: Tipos de Selección: Comienzo - Generación de Mutantes
- Page ID
- 52960
Mutaciones y selección
La evolución a nivel genético comienza con mutaciones que generan variantes genéticas. Las variantes se pueden seleccionar a favor o en contra si afectan a la aptitud y así aumentar o disminuir la frecuencia en una población. Si no hay selección en la variante, decimos que es “neutral”. En este caso, está sujeto a deriva genética y aumentará o disminuirá de frecuencia solo por casualidad. La mayoría de las mutaciones en animales se encuentran en regiones intergénicas, pero algunas ocurrirán naturalmente en los genes. Generalmente pensamos en estas mutaciones génicas como las que más probablemente se vean por selección. Si estamos buscando largos periodos de tiempo evolutivo, es probable que no veamos muchas mutaciones que reduzcan la condición física ya que éstas deben ser desyerbadas por selección natural o sexual. En cambio, esperamos ver mutaciones que son:
- neutral (sin efecto en el estado físico)
- casi neutral (muy poco efecto sobre el fitness)
- amortiguado (invisible debido a efectos epistáticos) o
- positivo (efecto positivo en el estado físico)
Cuando comparamos secuencias de genes homólogos (genes que provienen del mismo gen ancestral común) podemos realizar análisis para decirnos si la secuencia está bajo selección “positiva” o “purificadora”. Los genes bajo selección purificadora han tenido mutaciones “purificadas”. En este caso, hay una “mejor” secuencia y la mayoría de las especies en un taxón tendrán secuencias muy similares de este gen. Cuando surgen mutaciones en el gen, los individuos portadores de las mutaciones tienden a tener menor aptitud y no producirán muchos descendientes portadores de esas mutaciones. Comparando las secuencias de especies distantemente relacionadas vemos menos diferencias de las que esperaríamos ver por casualidad (o deriva genética neutra).
Los genes bajo selección positiva muestran lo contrario: más diferencias en la secuencia genética en un taxón de lo que esperaríamos ver por casualidad. Un gen seleccionado positivamente ha tenido antecedentes de mutaciones que dieron a los individuos una mayor aptitud. Estas mutaciones fueron diferentes en diferentes linajes, dando lugar a especies con diferentes conjuntos de mutaciones seleccionadas positivamente. En resumen, si la mayoría de las mutaciones en un gen lo hacen menos funcional y dan como resultado un efecto negativo en la aptitud, podríamos esperar ver una selección purificadora en ese gen (es decir, la mayoría de las mutaciones serán retiradas por selección natural, Figura 1). Pero si hay mutaciones específicas que funcionan mejor en diferentes ecosistemas, podríamos esperar ver una selección positiva en ese gen (es decir, diferentes mutaciones prevalecerán en diferentes ambientes).
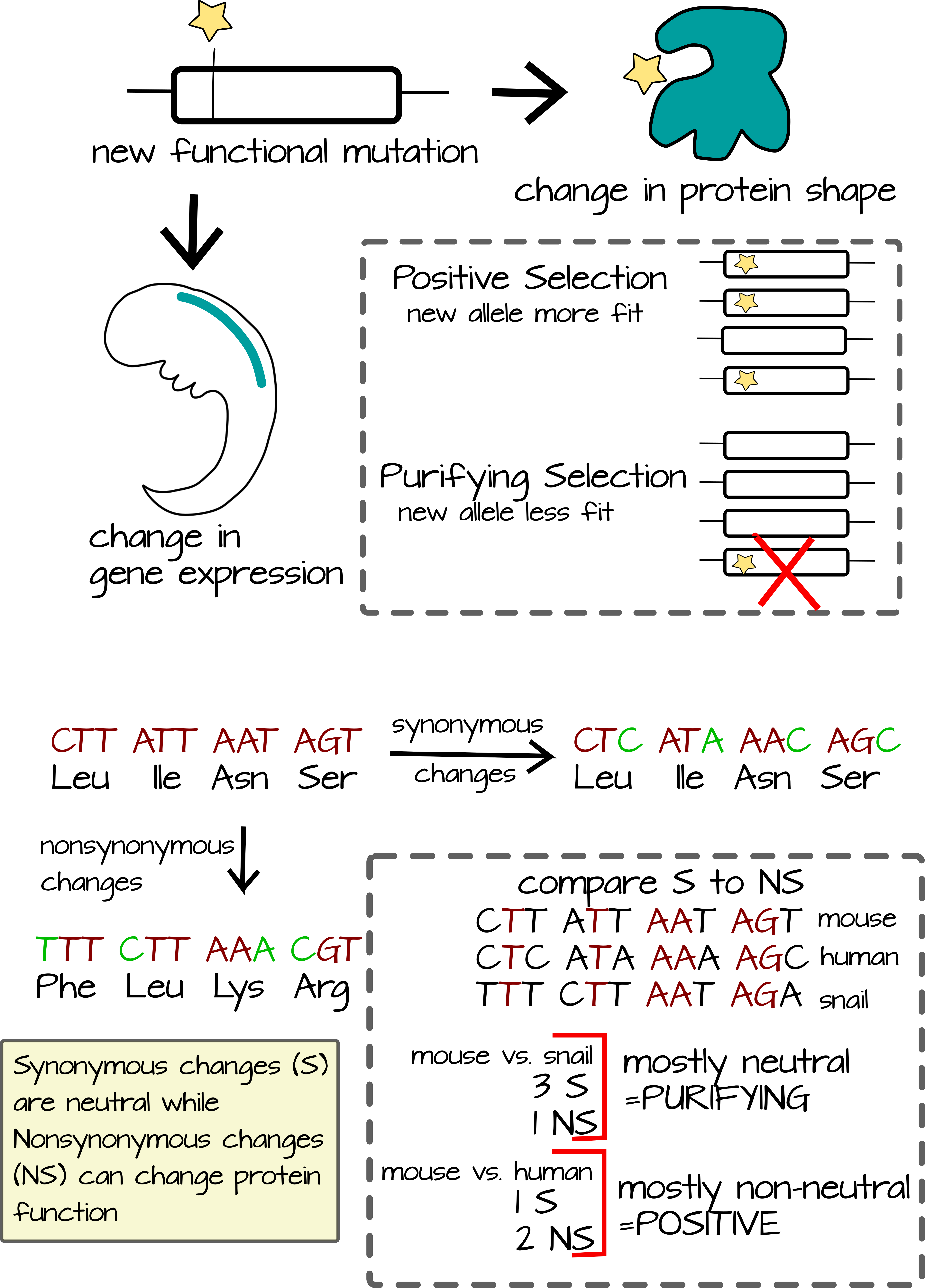
Para saber si un gen está bajo selección positiva o purificante, comparamos la secuencia génica entre organismos. Si una especie o linaje tiene muchos cambios funcionales en un gen en comparación con sus parientes, decimos que el gen está bajo selección positiva. Si hay muy pocos cambios funcionales entre especies distantemente relacionadas, decimos que el gen está bajo selección purificadora. Bajo selección purificante nuevas variantes génicas (alelos) tienden a disminuir la aptitud. Esto podría deberse a que el gen ya está en su punto máximo de aptitud o porque el gen afecta múltiples procesos (pleiotropía).
La “tasa de fondo” de mutación a lo largo del tiempo evolutivo puede estimarse mediante la acumulación de mutaciones neutras (o casi neutras). Se trata de mutaciones que no deben cambiar la función del gen. El lugar más fácil para buscar estos son los cambios sinónimos en la secuencia de ADN. Los cambios sinónimos son mutaciones en el ADN codificante de proteínas que no afectan la secuencia proteica. Estos suelen estar en el tercer codón. Los cambios no sinónimos pueden afectar la función al cambiar la secuencia de la proteína Debido a que los cambios sinónimos no afectan la función de la proteína, podemos usarlos para estimar la tasa de deriva neutra.
En el ejemplo mostrado, se comparan las secuencias de ratón, humano y caracol en una región codificante de proteínas. La secuencia de ADN que es la misma para todas las especies se muestra en rojo. Las comparaciones por pares que cuentan el número de mutaciones que resultan en un cambio de aminoácidos (NS para no sinónimos) se comparan con el número de mutaciones que no dan como resultado un cambio de aminoácidos (S para sinónimo). Cuando el número de cambios de S es mayor que el número de cambios de NS, es probable que el gen esté bajo selección purificadora. Es decir, el número de cambios funcionales es mucho menor que el número de cambios neutros. Cuando el número de cambios de S es menor que el número de cambios de NS, es probable que el gen esté bajo selección positiva.
Ubicación de la mutación en un gen
En la sección anterior, categorizamos las mutaciones por su efecto sobre el fenotipo. También podemos categorizar las mutaciones por su posición dentro de un gen. Puede haber mutaciones en una región codificante o mutaciones en una región cis-reguladora (también puede haber mutaciones en una región “basura” pero no necesitamos considerarlas aquí).
Las mutaciones reguladoras de CIS pueden cambiar el patrón de expresión de un gen y causar pérdida de expresión, disminución de la expresión, aumento de la expresión o expresión ectópica (expresión en un nuevo lugar y/o en un nuevo momento). Si el gen mutado es un gen regulador en sí mismo, esto también puede tener efectos aguas abajo sobre la expresión de sus dianas. En general, las mutaciones cis-reguladoras pueden cambiar la “huella celular”, que es el conjunto de genes expresados en una determinada célula que le da a esa célula su identidad y función. Esto puede cambiar la forma en que una célula “se comporta”, tanto en términos de su propia anatomía y fisiología como en la forma en que interactúa con las células que la rodean, potencialmente incluso cambiando su destino a través de la señalización célula-célula.
Las mutaciones en regiones codificantes pueden cambiar la estructura y función de una proteína o ARN. Estos cambios pueden ser estrictamente estructurales ya que afectan la anatomía celular, la fisiología y/o la señalización celular. Por ejemplo, una mutación en la proteína funcional Mioglobina puede cambiar lo bien que una célula muscular almacena oxígeno. Por otro lado, una mutación en el receptor de la vía Wnt Frizzled puede cambiar si una célula responde a la señalización de Wnt. La mutación Frizzled no es solo estructural, ya que también podría afectar la expresión del gen diana, activando o desactivando las dianas de la vía Wnt en un nuevo patrón.
Generando mutantes
Una tercera forma de categorizar las mutaciones es examinando su efecto físico sobre el genoma. ¿El evento mutacional borró una gran parte del genoma? ¿Convertió un nucleótido en otro? Los tres tipos principales de mutación que consideraremos son:
- Mutaciones puntuales: convertir un par de nucleótidos en otro
- Indels: Inserciones y deleciones que agregan o eliminan nucleótidos del genoma. Estos pueden ser causados por transposones, deslizamiento durante la replicación o enzimas reparadoras del ADN.
- Duplicaciones: Se trata de un tipo específico de mutación de inserción que inserta una pieza de secuencia genética existente en un nuevo locus sin eliminarlo del locus original.
De estos tres, las duplicaciones e inserciones pueden agregar complejidad genómica. Aquí estoy definiendo la complejidad como el número de piezas únicas en un sistema. La complejidad genómica se incrementaría si añadimos nueva información genética. Parece sencillo imaginar un caso en el que un aumento en la complejidad genómica resultaría en un aumento de la complejidad del organismo. Por ejemplo, agregamos un gen extra para digerir la amilosa y aumentamos la cantidad de grano que podemos comer, diversificando nuestra dieta y haciéndola más compleja. Sin embargo, también podemos aumentar la complejidad genómica sin aumentar la complejidad del organismo e incluso podemos aumentar la complejidad del organismo sin aumentar la complejidad genómica.


