4.1: Enzimas
- Page ID
- 56852
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Las enzimas son catalizadores. La mayoría son proteínas. (Se han descubierto algunas enzimas ribonucleoproteínicas y, para algunas de ellas, la actividad catalítica está en la parte de ARN más que en la parte de proteína. Enlace a la discusión de estas ribozimas). Las enzimas se unen temporalmente a uno o más de los reactivos, el sustrato o sustratos, de la reacción que catalizan. Al hacerlo, disminuyen la cantidad de energía de activación necesaria y así aceleran la reacción.

Ejemplos:
- La catalasa cataliza la descomposición del peróxido de hidrógeno en agua y oxígeno. \[2H_2O_2 \rightarrow 2H_2O + O_2\]Una molécula de catalasa puede romper 40 millones de moléculas de peróxido de hidrógeno cada segundo.
- La anhidrasa carbónica se encuentra en los glóbulos rojos donde cataliza la reacción. \[CO_2 + H_2O \rightleftharpoons H^+ + HCO_3^−\]Permite que los glóbulos rojos transporten dióxido de carbono desde los tejidos hasta los pulmones. Una molécula de anhidrasa carbónica puede procesar un millón de moléculas de CO 2 por segundo.
- La acetilcolinesterasa cataliza la descomposición del neurotransmisor acetilcolina en varios tipos de sinapsis así como en la unión neuromuscular, la sinapsis especializada que desencadena la contracción del músculo esquelético. Una molécula de acetilcolinesterasa descompone 25.000 moléculas de acetilcolina cada segundo. Esta velocidad hace posible el rápido “restablecimiento” de la sinapsis para la transmisión de otro impulso nervioso.
La actividad enzimática se puede analizar cuantitativamente. Para hacer su trabajo, una enzima debe unirse —aunque sea tan brevemente— con al menos uno de los reactivos. En la mayoría de los casos, las fuerzas que mantienen el sustrato en el sitio activo de la enzima son no covalentes, una variedad de enlaces de hidrógeno, interacciones iónicas e interacciones hidrofóbicas.
La mayoría de estas interacciones son débiles y especialmente si los átomos involucrados están más alejados de aproximadamente un angstrom entre sí. Por lo tanto, la unión exitosa del sustrato en el sitio activo de la enzima requiere que las dos moléculas sean capaces de acercarse entre sí de cerca sobre una superficie bastante amplia. De ahí la analogía de que una molécula sustrato une su enzima como una llave en una cerradura. Este requisito de complementariedad en la configuración de sustrato y enzima explica la notable especificidad de la mayoría de las enzimas. Generalmente, una enzima dada es capaz de catalizar solo una única reacción química o, como máximo, algunas reacciones que implican sustratos que comparten la misma estructura general.
Inhibición competitiva
La necesidad de un ajuste cercano, aunque breve, entre la enzima y el sustrato explica el fenómeno de la inhibición competitiva. Una de las enzimas necesarias para la liberación de energía dentro de la célula es la deshidrogenasa succínica. Cataliza la oxidación (por la eliminación de dos átomos de hidrógeno) del ácido succínico. Si se agrega ácido malónico a las células, o a una mezcla de tubo de ensayo de ácido succínico y la enzima, la acción de la enzima se inhibe fuertemente. Esto se debe a que la estructura del ácido malónico le permite unirse al mismo sitio en la enzima. Pero no hay oxidación por lo que no hay liberación rápida de productos. La inhibición se llama competitiva porque si aumentas la proporción de ácido succínico a ácido malónico en la mezcla, gradualmente restablecerás la velocidad de catálisis. En una proporción de 50:1, las dos moléculas compiten en términos aproximadamente iguales por el sitio de unión (=catalítico) en la enzima.
Cofactores enzimáticos
Muchas enzimas requieren la presencia de una no proteína adicional, un cofactor.
- Algunos de estos son iones metálicos como Zn 2+ (el cofactor de la anhidrasa carbónica), Cu 2+, Mn 2+, K + y Na +.
- Algunos cofactores son pequeñas moléculas orgánicas llamadas coenzimas. Las vitaminas B tiamina (B1), riboflavina (B2) y nicotinamida
son precursoras de coenzimas.
Las coenzimas pueden unirse covalentemente a la parte proteica (llamada apoenzima) de las enzimas como un grupo protésico. Otros se unen de manera más floja y, de hecho, pueden unirse solo transitoriamente a la enzima a medida que realiza su acto catalítico.
Varias lisozimas se encuentran en la naturaleza; en lágrimas humanas y clara de huevo, por ejemplo. La enzima es antibacteriana porque degrada el polisacárido que se encuentra en las paredes celulares de muchas bacterias. Esto lo hace catalizando la inserción de una molécula de agua en la formación de un enlace glicosídico. Esta hidrólisis rompe la cadena en ese punto.
El polisacárido bacteriano consiste en cadenas largas de aminoazúcares alternos:
- N-acetilglucosamina (NAG)
- Ácido N-acetilmurámico (NAM)
Estas unidades de hexosa se asemejan a la glucosa excepto por la presencia de las cadenas laterales que contienen grupos amino.
La lisozima es una proteína globular con una hendidura profunda en parte de su superficie. Seis hexosas del sustrato encajan en esta hendidura. Con tantos átomos de oxígeno en los azúcares, se forman hasta 14 enlaces de hidrógeno entre los seis aminoazúcares y ciertos grupos de aminoácidos R como Arg-114, Asn-37, Asn-44, Trp-62, Trp-63 y Asp-101. Algunos enlaces de hidrógeno también se forman con los grupos C=O de varios enlaces peptídicos. Además, las interacciones hidrofóbicas pueden ayudar a mantener el sustrato en posición.
La cristalografía de rayos X ha demostrado que a medida que la lisozima y su sustrato se unen, cada uno se deforma ligeramente. La cuarta hexosa en la cadena (anillo #4) se torsiona fuera de su posición normal. Esto impone una tensión en el enlace C-O en el lado anillo-4 del puente de oxígeno entre los anillos 4 y 5. Es justo en este punto que se rompe el polisacárido. Una molécula de agua se inserta entre estas dos hexosas, lo que rompe la cadena. Aquí, entonces, hay una visión estructural de lo que significa bajar la energía de activación. La energía necesaria para romper este enlace covalente es menor ahora que los átomos conectados por el enlace han sido distorsionados desde su posición normal.
En cuanto a la lisozima misma, la unión del sustrato induce un pequeño movimiento (~0.75Å) de ciertos residuos de aminoácidos por lo que la hendidura se cierra ligeramente sobre su sustrato. Entonces la “cerradura” así como la “llave” cambian de forma a medida que los dos se juntan. (Esto a veces se llama “ajuste inducido”).
Los residuos de aminoácidos en las proximidades de los anillos 4 y 5 proporcionan un mecanismo plausible para completar el acto catalítico. El residuo 35, ácido glutámico (Glu-35), es aproximadamente 3Å del puente -O- que se va a romper. El grupo carboxilo libre del ácido glutámico es un donante de iones de hidrógeno y está disponible para transferir H + al átomo de oxígeno. Esto rompería el enlace ya tenso entre el átomo de oxígeno y el átomo de carbono del anillo 4.
Ahora habiendo perdido un electrón, el átomo de carbono adquiere una carga positiva. El carbono ionizado es normalmente muy inestable, pero la atracción del ion carboxilo cargado negativamente de Asp-52 podría estabilizarlo el tiempo suficiente para que un ion -OH (de una molécula de agua disociada espontáneamente) se uniera con el carbono. Incluso a pH 7, el agua se disocia espontáneamente para producir iones H + y OH -. El ion hidrógeno (H +) sobrante puede reemplazar el perdido por Glu-35. En cualquier caso, la cadena se rompe, los dos fragmentos se separan de la enzima, y la enzima es libre de unirse a una nueva ubicación en la pared celular bacteriana y continuar su trabajo de digestión.
Factores que afectan la acción enzimática
La actividad de las enzimas se ve fuertemente afectada por los cambios en el pH y la temperatura. Cada enzima funciona mejor a un cierto pH (gráfico izquierdo) y temperatura (gráfico derecho), disminuyendo su actividad a valores por encima y por debajo de ese punto. Esto no es sorprendente considerando la importancia de la estructura terciaria (es decir, la forma) en la función enzimática y las fuerzas no covalentes, por ejemplo, interacciones iónicas y enlaces de hidrógeno, para determinar esa forma. Ejemplos:
- la proteasa pepsina funciona mejor como un pH de 1—2 (que se encuentra en el estómago)
- la proteasa tripsina es inactiva a un pH tan bajo pero muy activa a un pH de 8 (que se encuentra en el intestino delgado ya que el bicarbonato del fluido pancreático neutraliza el contenido estomacal que llega).
Los cambios en el pH alteran el estado de ionización de los aminoácidos cargados (por ejemplo, Asp, Lys) que pueden desempeñar un papel crucial en la unión al sustrato y/o la acción catalítica misma. Sin el grupo -COOH sindicalizado de Glu-35 y el -COO ionizado - de Asp-52, cesaría la acción catalítica de la lisozima. Los enlaces de hidrógeno se rompen fácilmente al aumentar la temperatura. Esto, a su vez, puede alterar la forma de la enzima para que su afinidad por su sustrato disminuya.
Regulación de la actividad enzimática
Varios mecanismos trabajan para hacer que la actividad enzimática dentro de la célula sea eficiente y bien coordinada.
Enzimas de anclaje en membranas
Muchas enzimas se insertan en las membranas celulares, por ejemplo,
- la membrana plasmática
- las membranas de mitocondrias y cloroplastos
- el retículo endoplásmico
- la envolvente nuclear
Estos están encerrados en relaciones espaciales que les permiten interactuar de manera eficiente.
Precursores inactivos
Las enzimas, como las proteasas, que pueden atacar a la propia célula son inhibidas mientras están dentro de la célula que las sintetiza. Por ejemplo, la pepsina se sintetiza dentro de las células principales (en las glándulas gástricas) como un precursor inactivo, el pepsinógeno. Solo cuando se expone al pH bajo fuera de la célula se elimina la porción inhibidora de la molécula y se produce pepsina activa.
Inhibición de retroalimentación

Si el producto de una serie de reacciones enzimáticas, por ejemplo, un aminoácido, comienza a acumularse dentro de la célula, puede inhibir específicamente la acción de la primera enzima involucrada en su síntesis (barra roja). Así se detiene la producción adicional de la enzima.
Activación de Precursores
La acumulación de una sustancia dentro de una célula puede activar específicamente (flecha azul) una enzima que pone en movimiento una secuencia de reacciones para las cuales esa sustancia es el sustrato inicial. Esto reduce la concentración del sustrato inicial.
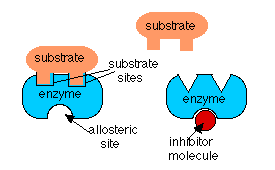
En el caso de la inhibición por retroalimentación y la activación del precursor, la actividad de la enzima está siendo regulada por una molécula que no es su sustrato. En estos casos, la molécula reguladora se une a la enzima en un sitio diferente al que se une el sustrato. Cuando el regulador se une a su sitio, altera la forma de la enzima para que se cambie su actividad. Esto se llama efecto alostérico. En la inhibición por retroalimentación, el efecto alostérico disminuye la afinidad de la enzima por su sustrato y en la activación precursora, la molécula reguladora aumenta la afinidad de la enzima en la serie por su sustrato.
Regulación de la síntesis enzimática
Los cuatro mecanismos descritos anteriormente regulan la actividad de las enzimas ya presentes dentro de la célula. ¿Qué pasa con las enzimas que no son necesarias o que son necesarias pero que no están presentes? Aquí, también, están en funcionamiento mecanismos de control que regulan la velocidad a la que se sintetizan nuevas enzimas. La mayoría de estos controles funcionan activando o apagando, la transcripción de genes.
Si, por ejemplo, ya están disponibles para la célula grandes cantidades de un aminoácido a partir de su fluido extracelular, se cierra la síntesis de las enzimas que permitirían a la célula producir ese aminoácido por sí misma. Por el contrario, si se pone a disposición de la célula un nuevo sustrato, puede inducir la síntesis de las enzimas necesarias para hacerle frente. Las células de levadura, por ejemplo, normalmente no metabolizan la lactosa, y no se puede detectar lactasa en ellas. Sin embargo, si se cultivan en un medio que contiene lactosa, pronto comienzan a sintetizar lactasa —transcribiendo y traduciendo el (los) gen (s) necesario (s )— y así pueden comenzar a metabolizar el azúcar. E. coli también tiene un mecanismo que regula la síntesis enzimática mediante el control de la traducción de un ARN mensajero necesario.


