5.12: Pirosecuenciación
- Page ID
- 57099
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)En los laboratorios de todo el mundo hay un intenso deseo de secuenciar más genomas.
- los de una amplia variedad de organismos para ayudar a establecer relaciones evolutivas;
- los de poblaciones agrupadas de microorganismos en, por ejemplo, agua de mar, suelo, intestino grueso;
- otros humanos para buscar genes que predispongan a enfermedades y patrones genéticos en diversos grupos étnicos.
Todos los genomas secuenciados enumerados en Tamaños del genoma se determinaron utilizando el método didesoxi-inventado por Frederick Sanger y descrito en otros casos. Sin embargo, ahora se está haciendo un gran esfuerzo para encontrar formas de secuenciar el ADN de manera más rápida (y más barata).
El secuenciador del genoma
Se están desarrollando varios métodos nuevos y uno ya está disponible comercialmente (el Genome Sequencer 20 System). Su método se llama pirosecuenciación o secuenciación por síntesis. Funciona así.
- El ADN que se va a secuenciar se divide en fragmentos de ~100 pares de bases y se desnaturaliza para formar ADN monocatenario (ADNss).
- Los fragmentos de ADNmc individuales se unen a perlas microscópicas, las cuales están separadas entre sí.
- La reacción en cadena de la polimerasa (PCR) se ejecuta en cada perla de manera que cada una se recubre con ~ 10 millones de copias idénticas de ese fragmento.
- Las perlas se colocan individualmente en pocillos microscópicos separados (~200,000 de ellos).
- Cada pozo recibe un cóctel de reactivos:
- ADN polimerasa — para añadir desoxirribonucleótidos al ssDNA
- adenosina fosfosulfato (APS)
- ATP sulfurilasa: una enzima que forma ATP a partir de adenosina fosfosulfato (APS) y pirofosfato (PPi)
- luciferina
- luciferasa - una ATPasa que cataliza la conversión de luciferina en oxilociferina con la liberación de luz
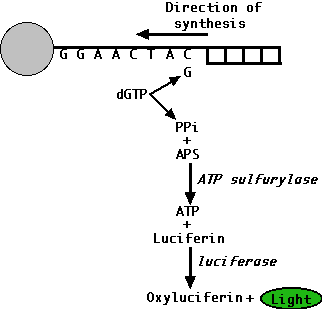

La ejecución de secuenciación:
- Cada uno de los miles de pozos está inundado con cuatro desoxirribonucleótidos, dTTP, dCTP y dGTP, pero en lugar de dATP (que desencadenaría la reacción de luciferina), se usa desoxiadenosina alfa-tiotrifosfato (dATP αS) en su lugar. La ADN polimerasa ignora la diferencia y la usa cada vez que se encuentra una T en el molde de ssDNA, pero la luciferasa no la reconoce.
- En cualquier pozo donde el nucleótido complementario esté presente en el extremo 3' del molde, se añade el nucleótido y se libera pirofosfato.
- La cantidad de luz es proporcional al número de ese nucleótido agregado. Entonces, si, por ejemplo, el nucleótido entrante es dGTP, y hay una cadena de 3 Cs en la plantilla, la luz emitida será 3 veces más brillante que si solo está presente una C.
- Un detector capta la luz (si la hay) de cada pozo y se registran los datos.
- Después, cada uno de los 3 nucleótidos restantes se agregan en secuencia.
- Después se repite la secuencia de 4 adiciones hasta que se completa la síntesis.
El diagrama anterior también muestra el tipo de datos producidos en un solo pozo. La altura del pico de producción de luz da el número de adiciones que ocurrieron cuando se agregó un nucleótido particular (parte inferior). Luego, el software de computadora muestra la secuencia de plantillas (arriba) para cada uno de los miles de fragmentos diferentes secuenciados. Con esta tecnología, se pueden aprender hasta 20 millones de pares de bases de secuencia genómica en una ejecución de instrumentos de menos de 6 horas.


