18.11: Endosimbiosis
- Page ID
- 57324
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)La teoría de la endosimbiosis postula que las mitocondrias de eucariotas evolucionaron a partir de una bacteria aeróbica (probablemente relacionada con las rickettsias) que vive dentro de una célula hospedadora arqueal y los cloroplastos de algas rojas, algas verdes y plantas evolucionaron a partir de una cianobacteria endosimbiótica que viven dentro de una célula hospedadora eucariota que contiene mitocondrias.
La Evidencia
- Tanto las mitocondrias como los cloroplastos pueden surgir solo de mitocondrias y cloroplastos preexistentes. No se pueden formar en una célula que carece de ellos porque los genes nucleares codifican sólo algunas de las proteínas de las que están hechos.
- Tanto las mitocondrias como los cloroplastos tienen su propio genoma, y se asemeja al de las bacterias no al del genoma nuclear.
- Ambos genomas consisten en una sola molécula circular de ADN.
- No hay histonas asociadas con el ADN.
- Tanto las mitocondrias como los cloroplastos tienen su propia maquinaria sintetizadora de proteínas, y se parece más a la de las bacterias que a la que se encuentra en el citoplasma de los eucariotas.
- El primer aminoácido de sus transcritos es siempre fMet como lo es en bacterias (no metionina [Met] que es el primer aminoácido en proteínas eucariotas).
- Varios antibióticos (por ejemplo, estreptomicina) que actúan bloqueando la síntesis de proteínas en bacterias también bloquean la síntesis de proteínas dentro de mitocondrias y cloroplastos. No interfieren con la síntesis de proteínas en el citoplasma de los eucariotas.
- Por el contrario, los inhibidores (por ejemplo, la toxina diftérica) de la síntesis de proteínas por ribosomas eucariotas no tienen -suficientemente sensatamente- ningún efecto sobre la síntesis de proteínas bacterianas ni sobre la síntesis de proteínas dentro de mitocondrias y cloroplastos.
- El antibiótico rifampicina, que inhibe la ARN polimerasa de bacterias, también inhibe la ARN polimerasa dentro de las mitocondrias. No tiene tal efecto sobre la ARN polimerasa dentro del núcleo eucariota.
El Genoma Mitocondrial
El genoma de las mitocondrias humanas contiene 16.569 pares de bases de ADN organizados en un círculo cerrado (Figura\(\PageIndex{1}\)). Estos codifican 2 ARN ribosómicos (ARNr), moléculas, 22 moléculas de ARN de transferencia (ARNt) y 13 polipéptidos. Los 13 polipéptidos participan en la construcción de varios complejos proteicos incrustados en la membrana mitocondrial interna e incluyen 7 subunidades que conforman la NADH deshidrogenasa mitocondrial, 3 subunidades de citocromo c oxidasa, 2 subunidades de ATP sintasa y citocromo b.
Todos estos productos génicos se utilizan dentro de la mitocondria, pero la mitocondria también necesita >900 proteínas diferentes, así como algunos ARNm y ARNt codificados por genes nucleares. Las proteínas (por ejemplo, el citocromo c y las ADN polimerasas utilizadas dentro de la mitocondria) se sintetizan en el citosol y luego se importan a la mitocondria.
El genoma del cloroplasto
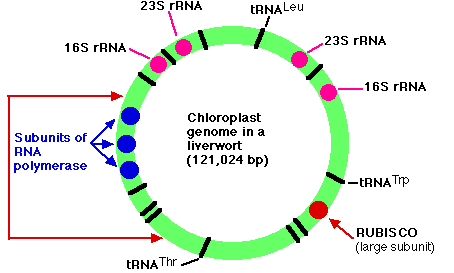
El genoma de los cloroplastos encontrados en Marchantia polymorpha (una hepática, una de las briófitas) contiene 121,024 pares de bases en círculo cerrado. Estos conforman unos 128 genes que incluyen:
- genes duplicados que codifican cada una de las cuatro subunidades (23S, 16S, 4.5S y 5S) del ARN ribosómico (ARNr) utilizado por el cloroplasto
- 37 genes que codifican todas las moléculas de ARN de transferencia (ARNt) utilizadas para la traducción dentro del cloroplasto. Algunos de estos están representados en la figura por barras negras (algunas de las cuales están etiquetadas).
- 4 genes que codifican algunas de las subunidades de la ARN polimerasa utilizadas para la transcripción dentro del cloroplasto (3 de ellos se muestran en azul)
- un gen que codifica la subunidad grande de la enzima RUBISCO (ribulosa bisfosfato carboxilasa oxigenasa)
- 9 genes para componentes de fotosistemas I y II
- 6 genes que codifican partes de la ATP sintasa de cloroplasto
- genes para 19 de las ~60 proteínas utilizadas para construir el ribosoma del cloroplasto
- Todos estos productos génicos se utilizan dentro del cloroplasto, pero todas las estructuras de cloroplastos también dependen de las proteínas RUBISCO, por ejemplo, la enzima que agrega CO 2 al bifosfato de ribulosa para iniciar el ciclo Calvino, consiste en múltiples copias de dos subunidades:
- Codificado por genes nucleares traducidos en el citosol e importados al cloroplasto.
- Una grande codificada en el genoma del cloroplasto y sintetizada dentro del cloroplasto, y una pequeña subunidad codificada en el genoma nuclear y sintetizada por ribosomas en el citosol. La subunidad pequeña debe entonces importarse al cloroplasto.
- La disposición de los genes que se muestra en la figura se encuentra no sólo en los briófitos (musgos y hepáticas) sino también en los licópsidos (e.g., Lycopodium y Selaginella). En todas las demás plantas, sin embargo, se invierte la porción de ADN entre corchetes por las flechas rojas de la izquierda. Los mismos genes están presentes pero en orden invertido. La cifra se basa en el trabajo de Ohyama, K., et al., Nature, 322:572, 7 de agosto de 1986; y Linda A. Raubeson y R. K. Jansen, Science, 255:1697, 27 de marzo de 1992.
- La evolución del cloroplasto eucariota por la endosimbiosis de una cianobacteria en una célula hospedadora eucariota que contiene mitocondrias condujo a la evolución de las algas verdes y plantas como se describió anteriormente, algas rojas y glaucófitas; un pequeño grupo de algas unicelulares.
Endosimbiosis Secundaria: Eucariotas Envolvente a Eucariotas
El Nucleomorfo
Una vez que ambos eucariotas heterótrofos y fotosintéticos habían evolucionado, los primeros envolvieron repetidamente a los segundos para explotar su forma de vida autotrófica. Muchos animales que viven hoy en día engullan algas para este propósito. Por lo general, los socios en estas relaciones mutualistas se pueden cultivar por separado. Sin embargo, un creciente cuerpo de evidencia indica que los cloroplastos de algunas algas no se han derivado envolviendo cianobacterias en una endosimbiosis primaria como las discutidas anteriormente, sino envolviendo eucariotas fotosintéticos (Figura 18.11.3). Esto se llama endosimbiosis secundaria. Ocurrió hace tanto tiempo que estos endosimbiontes no pueden cultivarse lejos de su huésped.
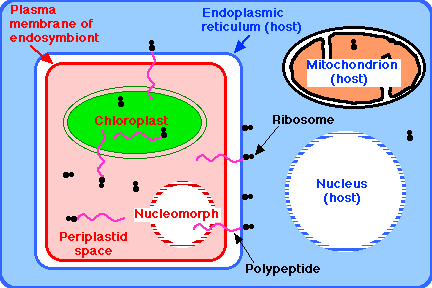
En dos grupos, la naturaleza eucariota del endosimbionte se puede observar por su retención de un vestigio de un núcleo (llamado su nucleomorfo). Un grupo de algas unicelulares y móviles llamadas criptomonadas parecen ser el resultado evolutivo de un flagelado eucariota no fotosintético (es decir, un protozoo) que envuelve una alga roja por endocitosis. Otro pequeño grupo de algas unicelulares, llamadas cloraracniofitos, parece ser el resultado de que un protozoo flagelado haya engullido una alga verde.
El resultado en ambos casos: una célula móvil autotrófica que contiene su propio núcleo, sus propias mitocondrias y su propio retículo endoplásmico. Este último contiene el endosimbionte con:
- su propia membrana plasmática
- su propio citoplasma, el espacio periplastidio
- sus propios ribosomas
- su propio cloroplasto, y
- su nucleomorfo, solo vestigio de su núcleo original, pero aún rodeado por una envoltura nuclear perforada con complejos de poros nucleares y que contiene un genoma minúsculo pero que aún funciona.
Los Cuatro Genomas de Guillardia theta
La criptomonada Guillardia theta contiene cuatro genomas diferentes:
- su propio genoma nuclear; con mucho el más grande con 87.2x10 6 pares de bases (pb) de ADN
- el genoma de sus mitocondrias (48.000 pb)
- el genoma del cloroplasto en su endosimbionte (121,000 pb)
- el genoma del nucleomorfo (551,264 pb)
Susan Douglas y sus colegas informaron (en la edición del 26 de abril de 2001 de Nature) el genoma completamente secuenciado del nucleomorfo.
- Contiene 3 pequeños cromosomas con
- 47 genes para ARN no mensajeros (ARNr, ARNt, ARNs)
- 464 genes para ARN mensajero; es decir, que codifican proteínas como
- 65 proteínas para sus propios ribosomas
- 30 por su cloroplasto (una pequeña fracción de los cientos necesarios)
- una variedad de proteínas necesarias dentro del nucleomorfo, incluyendo
- Factores de licencia de ADN
- histonas
- proteínas necesarias para la replicación del ADN (pero no genes para ADN polimerasas, que deben traducirse e importarse de los ribosomas del huésped)
Los genes están abarrotados de cerca en los tres cromosomas. De hecho, 44 de ellos se superponen entre sí. Sólo 17 genes contienen intrones, y estos son muy pequeños.
Interacciones del genoma en Guillardia theta
Millones de años de evolución han resultado en una compleja pero precisa orquestada gama de interacciones entre los 4 genomas. Por ejemplo:
- El cloroplasto necesita proteínas sintetizadas por 3 genomas diferentes: los propios, los nucleomorfos y los del huésped.
- El genoma nucleomorfo ha renunciado a todos (menos uno) de sus genes que codifican enzimas para funciones metabólicas generales; el endosimbionte ahora depende de los codificados por el núcleo huésped.
- El propio nucleomorfo también depende de genes (por ejemplo, para las ADN polimerasas) que residen en el núcleo del huésped.
El Apicoplast
El apicoplasto (abreviatura de “plastidio apicomplexano”) es un orgánulo solitario que se encuentra en los protistas apicomplexanos: “esporozoos” como Plasmodium falciparum (y los otros agentes de la malaria) y Toxoplasma gondii.
Características:
- Esencial: los organismos no pueden sobrevivir sin él.
- Encerrado por 4 membranas.
- Contiene su propio genoma, una molécula circular de ADN (35.000 pares de bases) que codifica
- ~ 30 proteínas
- un conjunto completo de ARNt más algunos otros ARN
- Solo se han descubierto algunas funciones, pero estas incluyen
- metabolismo anabólico como la síntesis de ácidos grasos
- reparación, replicación, transcripción y traducción de sus genes
- Claramente 30 proteínas no son suficientes para cumplir tantas funciones, por lo que el apicoplasto tiene que importar del citosol ~500 proteínas codificadas con el núcleo.
El apicoplasto es producto de una antigua endosimbiosis en la que el antepasado eucariota envolvió una alga unicelular -probablemente una alga roja- con un cloroplasto solitario. Con el tiempo, el núcleo se perdió (sin nucleomorfo residual) así como muchas características del cloroplasto (incluida su capacidad para realizar fotosíntesis).
Dos científicos japoneses han descubierto un flagelado heterótrofo que envuelve una alga verde unicelular que vive libremente en el agua circundante. Una vez dentro, el alga pierde sus flagelos y citoesqueleto; el huésped pierde su aparato de alimentación. Además, el hospedador cambia de nutrición heterotrófica a autotrófica (fotosíntesis) y el huésped se vuelve capaz de fototaxis. Cuando el huésped se divide por mitosis, solo una célula hija obtiene el plastidio. La otra célula vuelve a crecer el aparato de alimentación y está lista para engullir otra alga.


