10.3: Control del Ciclo Celular
( \newcommand{\kernel}{\mathrm{null}\,}\)
Habilidades para Desarrollar
- Entender cómo el ciclo celular es controlado por mecanismos tanto internos como externos a la célula
- Explicar cómo ocurren los tres puntos de control interno al final de G 1, en la transición G 2 /M y durante la metafase
- Describir las moléculas que controlan el ciclo celular mediante regulación positiva y negativa
La duración del ciclo celular es muy variable, incluso dentro de las células de un solo organismo. En humanos, la frecuencia de recambio celular va desde unas pocas horas en el desarrollo embrionario temprano, hasta un promedio de dos a cinco días para las células epiteliales, y a toda una vida humana gastada en G 0 por células especializadas, como las neuronas corticales o las células del músculo cardíaco. También hay variación en el tiempo que una célula pasa en cada fase del ciclo celular. Cuando las células de mamífero de división rápida se cultivan en cultivo (fuera del cuerpo en condiciones óptimas de crecimiento), la duración del ciclo es de aproximadamente 24 horas. En células humanas que se dividen rápidamente con un ciclo celular de 24 horas, la fase G 1 dura aproximadamente nueve horas, la fase S dura 10 horas, la fase G 2 dura aproximadamente cuatro horas y media, y la fase M dura aproximadamente media hora. En embriones tempranos de moscas de la fruta, el ciclo celular se completa en aproximadamente ocho minutos. El tiempo de eventos en el ciclo celular está controlado por mecanismos que son tanto internos como externos a la célula.
Regulación del Ciclo Celular por Eventos Externos
Tanto el inicio como la inhibición de la división celular son desencadenados por eventos externos a la célula cuando está a punto de comenzar el proceso de replicación. Un evento puede ser tan simple como la muerte de una célula cercana o tan barrido como la liberación de hormonas promotoras del crecimiento, como la hormona de crecimiento humana (HGH). La falta de HGH puede inhibir la división celular, resultando en enanismo, mientras que demasiada HGH puede resultar en gigantismo. El hacinamiento de células también puede inhibir la división celular. Otro factor que puede iniciar la división celular es el tamaño de la célula; a medida que una célula crece, se vuelve ineficiente debido a su decreciente relación superficie-volumen. La solución a este problema es dividir.
Cualquiera que sea la fuente del mensaje, la celda recibe la señal, y una serie de eventos dentro de la celda le permite proceder a la interfase. Avanzando desde este punto de inicio, todos los parámetros requeridos durante cada fase del ciclo celular deben cumplirse o el ciclo no puede progresar.
Regulación en los puntos de control internos
Es esencial que las células hijas producidas sean duplicados exactos de la célula parental. Los errores en la duplicación o distribución de los cromosomas conducen a mutaciones que pueden pasar hacia adelante a cada nueva célula producida a partir de una célula anormal. Para evitar que una célula comprometida continúe dividiéndose, existen mecanismos de control interno que operan en tres puntos de control del ciclo celular principal. Un punto de control es uno de varios puntos en el ciclo celular eucariota en los que se puede detener la progresión de una célula a la siguiente etapa del ciclo hasta que las condiciones sean favorables. Estos puntos de control ocurren cerca del final de G 1, en la transición G 2 /M, y durante la metafase (Figura10.3.1).
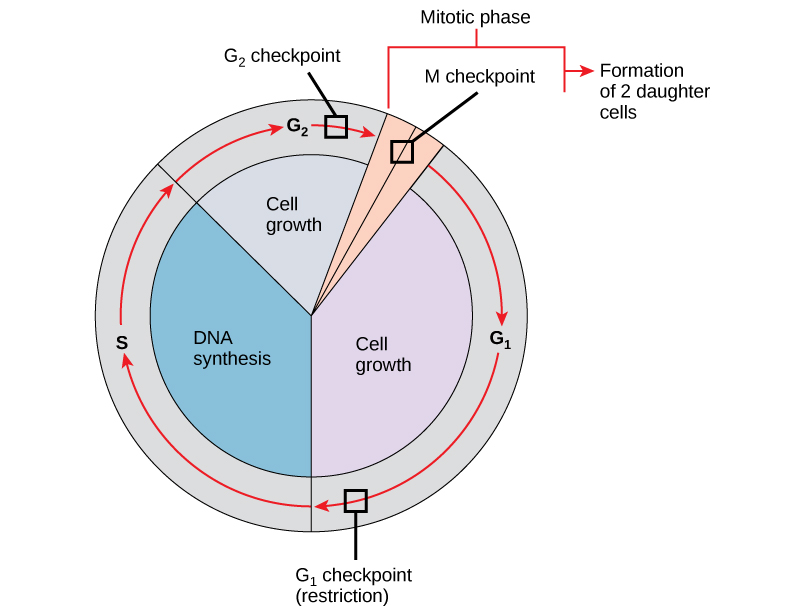
El puesto de control G 1
El punto de control G 1 determina si todas las condiciones son favorables para que continúe la división celular. El punto de control G 1, también llamado punto de restricción (en levadura), es un punto en el que la célula se compromete irreversiblemente con el proceso de división celular. Las influencias externas, como los factores de crecimiento, juegan un papel importante en llevar la célula más allá del punto de control G 1. Además de las reservas adecuadas y el tamaño celular, se realiza un chequeo de daños en el ADN genómico en el punto de control G 1. No se permitirá que una célula que no cumpla con todos los requisitos, avance a la fase S. La célula puede detener el ciclo e intentar remediar la condición problemática, o la célula puede avanzar a G 0 y esperar más señales cuando las condiciones mejoren.
El puesto de control G 2
El punto de control G 2 barre la entrada a la fase mitótica si no se cumplen ciertas condiciones. Al igual que en el punto de control G 1, se evalúan el tamaño celular y las reservas de proteínas. Sin embargo, el papel más importante del punto de control G 2 es asegurar que todos los cromosomas se hayan replicado y que el ADN replicado no esté dañado. Si los mecanismos de punto de control detectan problemas con el ADN, se detiene el ciclo celular y la célula intenta completar la replicación del ADN o reparar el ADN dañado.
El puesto de control M
El punto de control M ocurre cerca del final de la etapa metafásica de la cariocinesis. El punto de control M también se conoce como punto de control del huso, ya que determina si todas las cromátidas hermanas están correctamente unidas a los microtúbulos del huso. Debido a que la separación de las cromátidas hermanas durante la anafase es un paso irreversible, el ciclo no procederá hasta que los cinetocoros de cada par de cromátidas hermanas estén firmemente anclados a al menos dos fibras de huso que surgen de polos opuestos de la célula.
Enlace al aprendizaje

Mira lo que ocurre en los puntos de control G 1, G 2 y M visitando este sitio web para ver una animación del ciclo celular.
Moléculas Reguladoras del Ciclo Celular
Además de los puntos de control controlados internamente, existen dos grupos de moléculas intracelulares que regulan el ciclo celular. Estas moléculas reguladoras o bien promueven el progreso de la célula a la siguiente fase (regulación positiva) o detienen el ciclo (regulación negativa). Las moléculas reguladoras pueden actuar individualmente, o pueden influir en la actividad o producción de otras proteínas reguladoras. Por lo tanto, la falla de un solo regulador puede no tener casi ningún efecto sobre el ciclo celular, especialmente si más de un mecanismo controla el mismo evento. Por el contrario, el efecto de un regulador deficiente o que no funciona puede ser amplio y posiblemente fatal para la célula si se ven afectados múltiples procesos.
Regulación Positiva del Ciclo Celular
Dos grupos de proteínas, llamadas ciclinas y quinasas dependientes de ciclina (Cdks), son responsables del progreso de la célula a través de los diversos puntos de control. Los niveles de las cuatro proteínas ciclinas fluctúan a lo largo del ciclo celular en un patrón predecible (Figura10.3.2). Increases in the concentration of cyclin proteins are triggered by both external and internal signals. After the cell moves to the next stage of the cell cycle, the cyclins that were active in the previous stage are degraded.
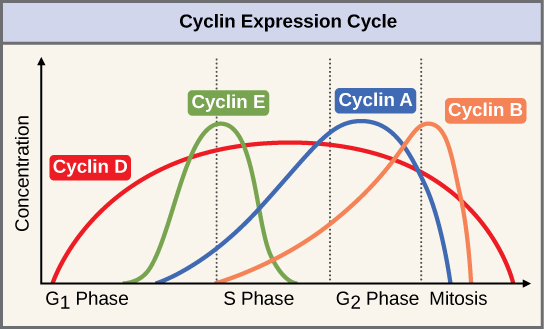
Cyclins regulate the cell cycle only when they are tightly bound to Cdks. To be fully active, the Cdk/cyclin complex must also be phosphorylated in specific locations. Like all kinases, Cdks are enzymes (kinases) that phosphorylate other proteins. Phosphorylation activates the protein by changing its shape. The proteins phosphorylated by Cdks are involved in advancing the cell to the next phase. (Figure 10.3.3). The levels of Cdk proteins are relatively stable throughout the cell cycle; however, the concentrations of cyclin fluctuate and determine when Cdk/cyclin complexes form. The different cyclins and Cdks bind at specific points in the cell cycle and thus regulate different checkpoints.
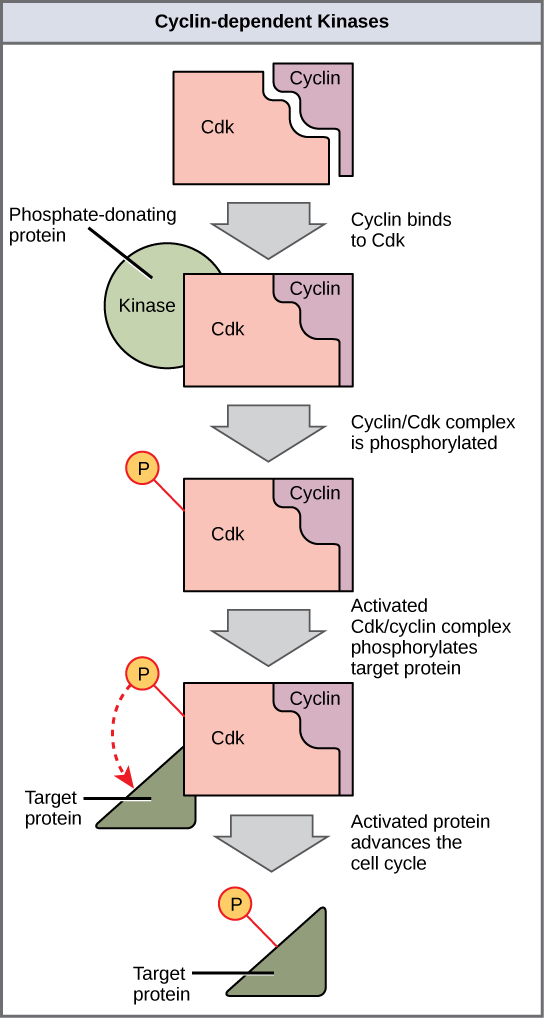
Since the cyclic fluctuations of cyclin levels are based on the timing of the cell cycle and not on specific events, regulation of the cell cycle usually occurs by either the Cdk molecules alone or the Cdk/cyclin complexes. Without a specific concentration of fully activated cyclin/Cdk complexes, the cell cycle cannot proceed through the checkpoints.
Although the cyclins are the main regulatory molecules that determine the forward momentum of the cell cycle, there are several other mechanisms that fine-tune the progress of the cycle with negative, rather than positive, effects. These mechanisms essentially block the progression of the cell cycle until problematic conditions are resolved. Molecules that prevent the full activation of Cdks are called Cdk inhibitors. Many of these inhibitor molecules directly or indirectly monitor a particular cell cycle event. The block placed on Cdks by inhibitor molecules will not be removed until the specific event that the inhibitor monitors is completed.
Negative Regulation of the Cell Cycle
The second group of cell cycle regulatory molecules are negative regulators. Negative regulators halt the cell cycle. Remember that in positive regulation, active molecules cause the cycle to progress.
The best understood negative regulatory molecules are retinoblastoma protein (Rb), p53, and p21. Retinoblastoma proteins are a group of tumor-suppressor proteins common in many cells. The 53 and 21 designations refer to the functional molecular masses of the proteins (p) in kilodaltons. Much of what is known about cell cycle regulation comes from research conducted with cells that have lost regulatory control. All three of these regulatory proteins were discovered to be damaged or non-functional in cells that had begun to replicate uncontrollably (became cancerous). In each case, the main cause of the unchecked progress through the cell cycle was a faulty copy of the regulatory protein.
Rb, p53, and p21 act primarily at the G1 checkpoint. p53 is a multi-functional protein that has a major impact on the commitment of a cell to division because it acts when there is damaged DNA in cells that are undergoing the preparatory processes during G1. If damaged DNA is detected, p53 halts the cell cycle and recruits enzymes to repair the DNA. If the DNA cannot be repaired, p53 can trigger apoptosis, or cell suicide, to prevent the duplication of damaged chromosomes. As p53 levels rise, the production of p21 is triggered. p21 enforces the halt in the cycle dictated by p53 by binding to and inhibiting the activity of the Cdk/cyclin complexes. As a cell is exposed to more stress, higher levels of p53 and p21 accumulate, making it less likely that the cell will move into the S phase.
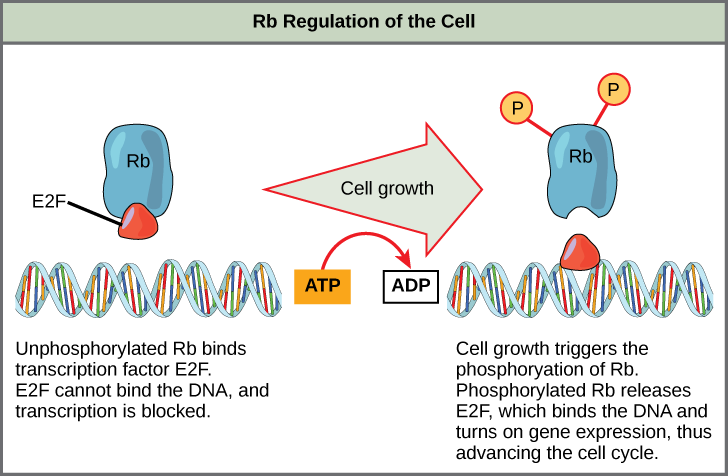
Rb exerts its regulatory influence on other positive regulator proteins. Chiefly, Rb monitors cell size. In the active, dephosphorylated state, Rb binds to proteins called transcription factors, most commonly, E2F (Figure 10.3.4). Transcription factors “turn on” specific genes, allowing the production of proteins encoded by that gene. When Rb is bound to E2F, production of proteins necessary for the G1/S transition is blocked. As the cell increases in size, Rb is slowly phosphorylated until it becomes inactivated. Rb releases E2F, which can now turn on the gene that produces the transition protein, and this particular block is removed. For the cell to move past each of the checkpoints, all positive regulators must be “turned on,” and all negative regulators must be “turned off.”
Exercise 10.3.1
Rb and other proteins that negatively regulate the cell cycle are sometimes called tumor suppressors. Why do you think the name tumor suppressor might be appropriate for these proteins?
- Answer
-
Rb and other negative regulatory proteins control cell division and therefore prevent the formation of tumors. Mutations that prevent these proteins from carrying out their function can result in cancer.
Summary
Each step of the cell cycle is monitored by internal controls called checkpoints. There are three major checkpoints in the cell cycle: one near the end of G1, a second at the G2/M transition, and the third during metaphase. Positive regulator molecules allow the cell cycle to advance to the next stage. Negative regulator molecules monitor cellular conditions and can halt the cycle until specific requirements are met.
Glossary
- cell cycle checkpoint
- mechanism that monitors the preparedness of a eukaryotic cell to advance through the various cell cycle stages
- cyclin
- one of a group of proteins that act in conjunction with cyclin-dependent kinases to help regulate the cell cycle by phosphorylating key proteins; the concentrations of cyclins fluctuate throughout the cell cycle
- cyclin-dependent kinase
- one of a group of protein kinases that helps to regulate the cell cycle when bound to cyclin; it functions to phosphorylate other proteins that are either activated or inactivated by phosphorylation
- p21
- cell cycle regulatory protein that inhibits the cell cycle; its levels are controlled by p53
- p53
- cell cycle regulatory protein that regulates cell growth and monitors DNA damage; it halts the progression of the cell cycle in cases of DNA damage and may induce apoptosis
- retinoblastoma protein (Rb)
- regulatory molecule that exhibits negative effects on the cell cycle by interacting with a transcription factor (E2F)


