1.2: Estructura de ADN y ARN
- Page ID
- 57191
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)La Doble Hélice
El ADN (ácido desoxirribonucleico) y el ARN (ácido ribonucleico) están compuestos por dos clases diferentes de bases que contienen nitrógeno: las purinas y las pirimidinas. Las purinas más comunes en el ADN son adenina y guanina:
.png)
Figura 1.2.1: Purinas
Las pirimidinas más comunes en el ADN son la citosina y la timina:
.png)
Figura 1.2.2: Piramidinas
El ARN contiene las mismas bases que el ADN con la excepción de la timina. En cambio, el ARN contiene la pirimidina uracilo:
.png)
Figura 1.2.3: Timina vs. Uracilo
Adenina, guanina, citosina, timina y uracilo suelen abreviarse utilizando los códigos de una sola letra A, G, C, T y U, respectivamente.
Las purinas y pirimidinas pueden formar enlaces químicos con azúcares de pentosa (5 carbonos). Los átomos de carbono en los azúcares se designan 1', 2', 3', 4' y 5'. Es el carbono 1' del azúcar que se une al átomo de nitrógeno en la posición N1 de una pirimidina o N9 de una purina. Los precursores de ADN contienen la pentosa desoxirribosa. Los precursores de ARN contienen la pentosa ribosa (que contiene un grupo OH adicional en la posición 2'):
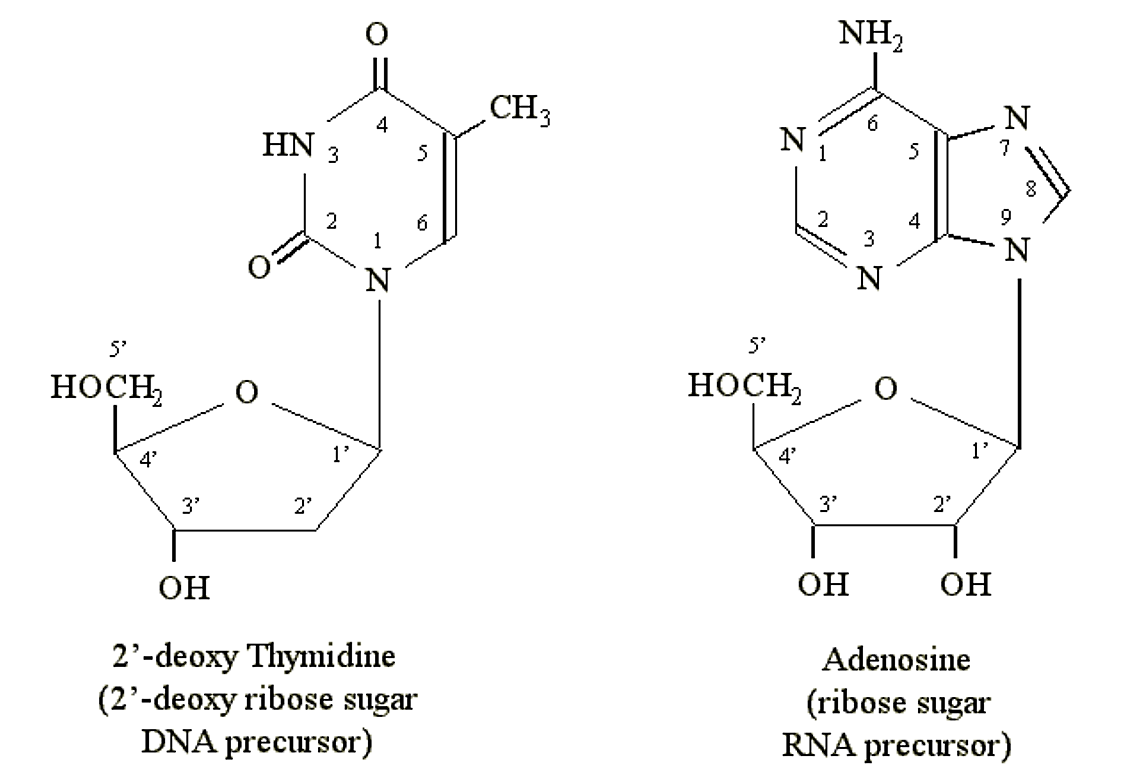.png)
Figura 1.2.4: Nucleósidos
Antes de que un nucleósido pueda formar parte de una molécula de ADN o ARN, debe complejarse con un grupo fosfato para formar un nucleótido (ya sea un desoxirribonucleótido o ribonucleótido). Los nucleótidos pueden poseer 1, 2 o 3 grupos fosfato, por ejemplo los nucleótidos adenosina monofosfato (AMP), adenosido difosfato (ADP) y adenosina trifosfato (ATP). Los grupos fosfato están unidos al carbono 5' del resto de azúcar de ribosa. Comenzando con el grupo fosfato unido al carbono de ribosa 5', se etiquetan como fosfato a, b y g. Es el nucleótido trifosfato que se incorpora en el ADN o ARN.
.png)
Figura 1.2.5: Nucleótido
El ADN y el ARN son simplemente polímeros largos de nucleótidos llamados polinucleótidos. Solo el fosfato a está incluido en el polímero. Se une químicamente al carbono 3' del resto de azúcar de otro nucleótido:
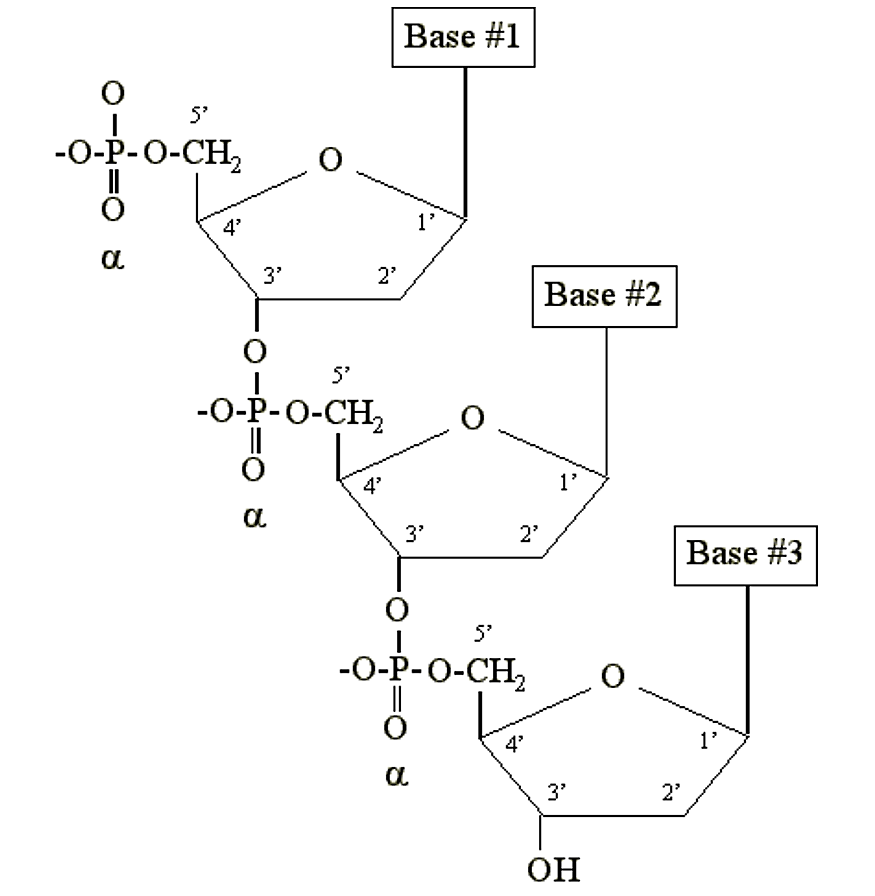.png)
.png)
Resumen de términos:
|
|
|
|
|
|
|
|---|---|---|---|---|---|
| Adenina | Adenosina | (Acido adenílico) |
|
|
|
| Guanina | Guanosina | (Ácido guanílico) |
|
|
|
| Citosina | Citidina | (Ácido citidílico) |
|
|
|
| Timina | Timidina | (Ácido timidílico) |
|
|
|
| Uracilo | Uridina | (Ácido uridílico) |
|
|
¿Cuál es la estructura del ADN? ¿Cómo se relaciona la estructura con la función?
1950's
Se conocía la estructura química primaria de los polinucleótidos (es decir, el enlace fosfato 3'-5').
1951 E. Chargaff
El experimento:
Tomar ADN de una variedad de especies e hidrolizarlo para producir pirimidinas y purinas individuales. Determinar las concentraciones relativas de las bases A, T, C y G.
Resultado:
Aunque diferentes especies tenían proporciones de pirimidinas o purinas singularmente diferentes, las concentraciones relativas de adenina siempre fueron iguales a las de timina, y la guanina equivalía a citosina.
1950 R.E. Franklin
Los estudios de difracción de rayos X de fibras de ADN demostraron que el ADN adoptó una estructura helicoidal altamente ordenada. Franklin concluyó que dos o más cadenas deben arrollarse una alrededor de la otra para formar una hélice. Algunas dimensiones básicas de la hélice se calcularon a partir de los datos de difracción de rayos X.
1953 L. Pauling y R.B. Corey
Proponer una estructura helicoidal de tres cadenas para el ADN con el esqueleto de fosfato en el centro y las bases en el exterior.
1953 J.D. Watson y F.H.C. Crick
Identificó un arreglo de enlaces de hidrógeno entre modelos de bases de timina y adenina, y entre bases de citosina y guanina que llenaron la regla de Chargaff:
.png)
Figura 1.2.7: Vinculación de reglas de Chargaff
Tenga en cuenta que el par “TA” puede superponer al par “GC” con los enlaces a los grupos de azúcar en yuxtaposición similar. En el modelo de “doble hélice” de Watson y Crick las cadenas polinucleotídicas interactúan para formar una doble hélice con las cadenas corriendo en direcciones opuestas. Las bases se dirigen hacia el centro (y se apilan una encima de la otra) y las cadenas principales de azúcar se enfrentan al exterior de la hélice.
El modelo de Watson y Crick tuvo las siguientes dimensiones físicas:
- 34 Å por repetición helicoidal
- 10 pares de bases por repetición (es decir, por giro de la hélice)
- Distancia de apilamiento entre bases de 3.4 Å
- 20 Å de diámetro para la anchura helicoidal
Las características físicas del modelo coincidieron con las determinadas por los estudios de difracción de rayos X de Rosalind Franklin.
Consecuencias del modelo de información genética:
El artículo de Watson y Crick fue un ejercicio de brevedad (1 página sólo en Nature). La estructura era tan rica de implicación que se podía escribir bastante. Los autores, sin embargo, optaron únicamente por decir “No ha escapado a nuestro aviso que el emparejamiento específico que hemos postulado sugiere de inmediato un posible mecanismo de copia para el material genético”.
- Si G siempre se emparejó con C, y T siempre se emparejó con A, entonces cualquiera de las cadenas podría regenerarse a partir de la información complementaria en la otra hebra.
- La base de la complementariedad fueron los enlaces de hidrógeno, es decir, interacciones no covalentes que podrían romperse y reformarse fácilmente.
- La información que transportaba el ADN estaba dentro de la secuencia de bases única del ADN.
- De la ubicación interior general de las bases, parecería que la doble hélice tendría que disociarse para poder acceder a la información.
- La ubicación no equitativa de los restos de azúcar (ver arriba) sugirió que la hélice de ADN tendría un surco mayor y un surco menor.
Notación general del ADN bicatenario:
.png)


