8.4: Propagación de ADN en bacterias
- Page ID
- 56282
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Complementación
George Beadle y Edward Tatum describieron por primera vez el concepto de que cada gen correspondía a una enzima en una vía metabólica al exponer la levadura Neurospora crassa a condiciones mutagénicas (Beadle & Tatum, 1941). Siguiendo estos procedimientos, Joshua Lederberg continuó estos estudios con Tatum donde generaron dos cepas mutantes en Escherichia coli. Estas bacterias fueron auxótrofas, incapaces de generar algunos nutrientes básicos necesarios para sostener su crecimiento. Las dos cepas fueron descritas como met − bio − Thr + Leu + Thi + (Cepa A) y Met + Bio + thr − leu − thi − (Cepa B). La cepa A puede sintetizar suficientemente los aminoácidos treonina, leucina y el cofactor tiamina mientras que es deficiente en la producción del cofactor biotina y el aminoácido metionina, mientras que lo contrario fue cierto para la cepa B. Cuando cualquiera de estas dos cepas se sembró en medio mínimo, no se produjo crecimiento alguno. Complementar medios mínimos con metionina y biotina permitió que la Cepa A crezca de forma normal. Cuando las dos cepas se mezclaron y se sembraron en medio mínimo, hubo un crecimiento de bacterias. Las dos cepas fueron capaces de complementarse entre sí de alguna manera como si hubiera ocurrido un intercambio sexual de material genético (Lederberg & Tatum, 1946).
Las bacterias están equipadas con todas las capacidades necesarias para replicar el ADN. Las especies bacterianas comunes tienen abejas adaptadas para su uso en el laboratorio para portar ADN y propagarlo para usos en biotecnología. Además del ADN cromosómico del genoma bacteriano, las bacterias también tienen ADN extracromosómico llamado plásmidos. Estos plásmidos se replican independientemente del cromosoma bacteriano y pueden presentarse en una copia alta. Estas piezas circulares de ADN se modifican en laboratorios para portar piezas específicas de ADN para que puedan ser estudiadas o utilizadas para la expresión en proteínas. Los plásmidos pueden portar naturalmente rasgos importantes, incluida la resistencia a los antibióticos. Los plásmidos son relativamente pequeños, variando en tamaño desde 1000 bases hasta 1,000,000 bases de largo (1kb-1000kb).
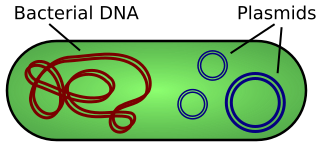
El ADN bacteriano suele existir como un cromosoma circular grande (rojo). Los plásmidos son fragmentos extracromosómicos y de replicación autónoma de ADN (azul).
A través de un proceso llamado conjugación, las bacterias pueden transferir “sexualmente” material genético a otra pasando plásmidos a través de una estructura llamada pilus de conjugación.

Proceso de conjugación entre un donante portador de plásmido y un receptor sin plásmido. El donante crea un pilus de conjugación para crear un puente citosólico con el donante donde el plásmido se replica en el receptor a través del método de círculo rodante de replicación. El receptor se vuelve entonces competente para actuar como donante.
Características de los plásmidos
Los plásmidos que son diseñados por los biólogos para transportar piezas de ADN para su estudio se denominan vectores porque mueven una pieza de ADN.

Estos vectores plasmídicos tienen las mismas características que los plásmidos tradicionales con la capacidad de replicarse independientemente del genoma bacteriano. La característica que permite que estos ADN se repliquen se denomina origen de replicación (ori) que suele ser rico en A y T. Sin embargo, estos vectores plasmídicos tienen las propiedades adicionales que los hacen fáciles de trabajar y distinguirlos de los plásmidos bacterianos; un marcador de selección y un sitio de clonación múltiple. Un marcador de selección generalmente viene en forma de un gen que codifica la resistencia a un antibiótico específico. En el plásmido de la foto, resistencia a la ampicilina otorgada por el gen β-lactamasa. El sitio de clonación múltiple (MCS), también conocido como el polienlazador, es la ubicación en la que el ADN de interés se incorpora al vector. Las MCS se definen por un conjunto de sitios únicos donde el ADN puede ser cortado por endonucleasas de restricción (RE). Como su nombre lo indica, las enzimas de restricción están “restringidas” en su capacidad para cortar o digerir el ADN. La restricción que es útil para los biólogos suele ser secuencias de ADN palindrómico. Las secuencias palindrómicas son la misma secuencia hacia adelante y hacia atrás. Algunos ejemplos de palíndromos: CARRERA, CIVIC, UN HOMBRE UN PLAN A CANAL PANAMA. Con respecto al ADN, hay 2 hebras que corren antiparalelas entre sí. Por lo tanto, el complemento inverso de una hebra es idéntico al otro.


EcoRI genera pegajoso de extremos cohesivos SmaI genera extremos romos
Las enzimas de restricción hidrolizan enlaces fosfodiéster covalentes del ADN para dejar extremos “adherentes/cohesivos” o extremos “romos”. Esta distinción en el corte es importante porque un extremo pegajoso EcoRI se puede usar para emparejar un trozo de ADN cortado con la misma enzima para pegarlos o ligarlos nuevamente. Mientras que las endonucleasas cortan el ADN, las ligasas las unen de nuevo. El ADN digerido con EcoRI puede ligarse de nuevo junto con otro trozo de ADN digerido con EcoRI, pero no a un trozo digerido con SmaI. Otro cortador romo es EcoRV con una secuencia de reconocimiento de GAT | ATC.
Al “cortar y pegar” ADN en vectores, podemos introducir ADN extraño o exógeno en bacterias. Este tipo de ADN ahora se llama ADN recombinante y es el corazón de la biotecnología.
Tecnología de ADN recombinante
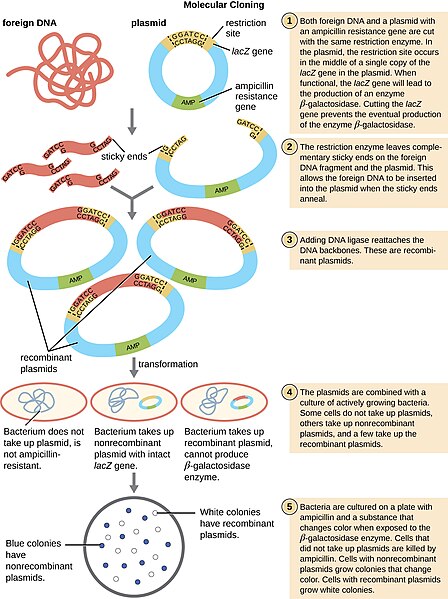

Recursos adicionales
Preguntas para el Pensamiento
1. ¿Por qué crees que los orígenes de la replicación están hechos de A y T?
2. ¿Qué hay de diferente en los tipos de enlaces que mantienen unidas las cadenas dobles frente a los enlaces fosfodiéster de la cadena principal del ADN?
3. ¿Se puede digerir el ADN con SmaI ser ligado al ADN digerido con EcoRV?
4. Si es así, ¿qué enzima podrá digerir este nuevo ADN?
- Beadle, G. W.; Tatum, E. L. (1941). “Control Genético de Reacciones Bioquímicas en Neurospora”. Actas de la Academia Nacional de Ciencias. 27 (11): 499—506. doi: 10.1073/pnas.27.11.499. PMC 1078370. PMID 16588492
- Lederberg J, Tatum EL (1946). “Recombinación génica en E. coli”. Naturaleza. 158 (4016): 558. doi: 10.1038/158558a0


