10.1: Introducción
- Page ID
- 56270
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Historia de la Transformación Genética
Cualquier captación de información genética del entorno externo hacia las células que resulte en la expresión de nuevos rasgos se denomina transformación genética. Este proceso puede ocurrir de forma natural. Algunas bacterias son referidas como “competentes” para indicar que son capaces de tomar ADN a la célula desde el ambiente. Esto se conoce como competencia natural. Las bacterias también son capaces de recibir ADN a través del proceso de conjugación donde los plásmidos de una bacteria se envían a otra a través del pilus de conjugación. Otros métodos de introducción de ADN extraño incluyen la inyección directa en el citosol o mediante el uso de virus en un proceso llamado transducción. En las células eucariotas, nos referimos a la introducción del ADN como transfección.
Frederick Griffith y el agente transformador
Al inicio de la biología moderna, no se sabía que la fuente de material genético fuera el ADN. De hecho, muchos científicos pensaban que el ADN era demasiado sencillo para realizar este trabajo. Los científicos creían que las proteínas, con sus 20 aminoácidos variados, eran las portadoras de información genética. En un intento de desarrollar una vacuna para la neumonía inducida por bacterias, Frederick Griffith fue el primero en describir el proceso de transformación genética por accidente en 1928. Griffith tomó una cepa virulenta de bacterias (de apariencia suave) que causó neumonía y las inyectó en ratones. Esto resultaría en la muerte de los ratones. También observó que la inyección de una bacteria áspera no causó ninguna enfermedad. Después de matar por calor a las bacterias lisas, descubrió que se requerían bacterias vivas de la cepa virulenta para que la enfermedad progresara. Por último, observó que inyectar a las bacterias virulentas muertas por calor bacterias vivas de la cepa no virulenta resultó en neumonía y muerte en los ratones. A partir de este experimento, se encontró que un agente transformante con la capacidad de transmitir un rasgo estaba dentro del contenido de esas células muertas. Pero nadie sabía que este agente fuera ADN en ese momento.

Hershey y Chase
Hershey y Chase estudiaron bacteriófagos (fago=devorador). Los fagos son virus bacterianos que infectan bacterias y causan lisis de las células. Tienen una estructura muy simple de una cabeza/collar/cola proteinácea y un núcleo de ADN. Se sabía que las bacterias infectadas con fagos eran resistentes a infecciones adicionales. En 1952 Hershey y Chase cultivaron bacteriófagos en condiciones que marcarían específicamente el ADN o la proteína con radiactividad. Posteriormente tomaron fagos con ADN radiomarcada e infectaron bacterias. Paralelamente, tomaron fagos con proteína radiomarcada e infectaron a otro conjunto de bacterias. Después del tiempo justo para la infección, los cultivos bacterianos se colocaron en una licuadora para separar el bacteriófago de la bacteria.
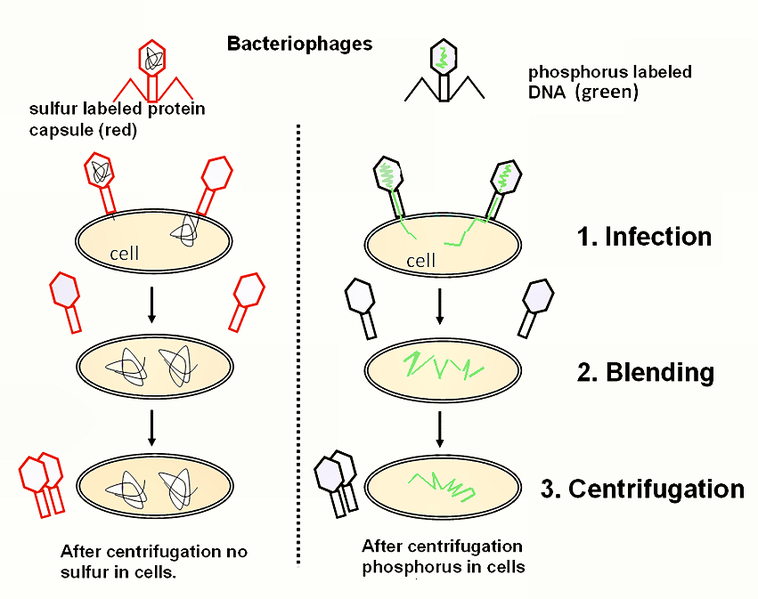
Las soluciones se centrifugaron para aislar bacterias del fago. Las bacterias fueron radiactivas solo cuando el fago cultivado en condiciones para radiomarcar el ADN infectó a la bacteria para indicar que el ADN podría ser el agente transformante.
Preguntas para pensar
- ¿Qué isótopo se usaría para etiquetar proteína?
- ¿Qué isótopo se usaría para etiquetar solo ADN?
Avery, McCarty y MacCleod
En 1944, Oswald Avery, Colin MacCleod y Maclyn McCarty repitieron el experimento de Griffith. En lugar de usar bacterias muertas por calor, estos científicos aislaron proteínas, carbohidratos, lípidos y ácidos nucleicos de la cepa virulenta y coinyectaron con las bacterias no virulentas. Los extractos de carbohidratos fueron ineficaces para transformar bacterias. Los extractos proteicos fueron incapaces de causar transformación. Las inyecciones lipídicas no pudieron dar lugar a virulencia. Solo las muestras de ácido nucleico tratadas con RNasa fueron capaces de transformar bacterias. Al coinyectarse con DNasa, las bacterias no se transformaron. Junto con Hershey-Chase, esto ilustró definitivamente que el ADN era el agente transformador capaz de transferir información genética.



