11.2: Transcripción procariota
- Page ID
- 56156
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)La intolerancia a la lactosa de las bacterias
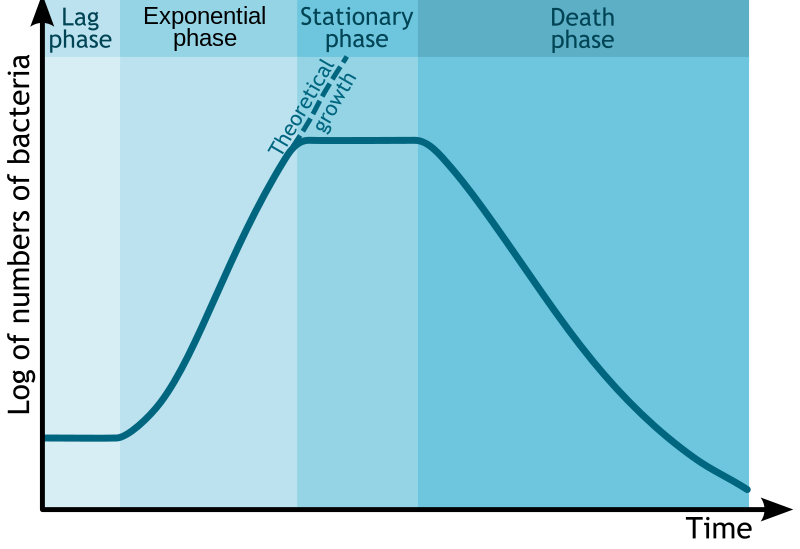
La cinética de crecimiento estándar de E. coli se describe por la curva. Crédito: Michał Komorniczak (CC-BY-SA 3.0)
La glucosa es la fuente de energía preferida de las células. François Jacob y Jacques Monod buscaron comprender cómo las bacterias tomaban decisiones para cambiar entre diferentes azúcares como fuentes de energía. Jacob y Monod encontraron que si la glucosa y la lactosa se presentaran como alimento para bacterias, habría un patrón de crecimiento bifásico.


Crédito: CNX OpenStax (CC-BY 4.0)
Jacob y Monod llegaron a entender que primero se utilizaría la glucosa (fuente preferida) y la lactosa se digiriría después del agotamiento de la glucosa. Esto ocurrió porque en situaciones normales la bacteria no tendría acceso a la lactosa y desperdiciaría energía creando enzimas para digerirla. La enzima β-galactosidasa, que se encarga de digerir la lactosa a los monómeros galactosa y glucosa solo se induciría en condiciones de glucosa baja y lactosa alta. Monod encontró que cuando la lactosa era el único azúcar, se indujo la expresión de la enzima β-galactosidasa y mostró un crecimiento monofásico con retraso.
El Operón Lac
Jacob y Monod más tarde encontraron que los genes involucrados en la utilización de lactosa se agruparon en proximidad bajo un mecanismo de control coordinado. Esto se conoció como el operón Lac.

Un esquema del Operón Lac. LacZ, LacY y LacA se transcriben como un solo ARNm.
El uso de lactosa como fuente de energía es preferido por las bacterias cuando la glucosa no está presente. En presencia de abundante glucosa, sería un desperdicio de energía y recursos celulares comprometerse a sintetizar el ARNm y la proteína para β-galactosidasa. A menos que esté presente lactosa, una proteína se une a una porción del promotor Lac al que se hace referencia como el operador. Esta proteína represora está codificada por otro gen (lacI) fuera del grupo génico. Ocasionalmente, el represor se desune al operador y se permite que la ARN polimerasa transcriba el gen lacZ (β-galactosidasa), el gen lacY (permeasa) y el gen lacA (acetilasa). Esta “pérdida” de expresión es importante ya que la proteína permeasa es necesaria en la superficie de la célula para permitir que la lactosa entre en la célula si está presente en el ambiente. La presencia de lactosa hace que el represor se caiga del operador para otorgar acceso a ARN pol al ADN. Cuando la glucosa es baja, una proteína llamada CAP (Catabolite Activated Protein) se une al promotor Lac y funciona como reclutador de ARN pol. Los efectos coordinados de la activación de CAP y la inactivación del represor Lac producen una alta transcripción del operón.
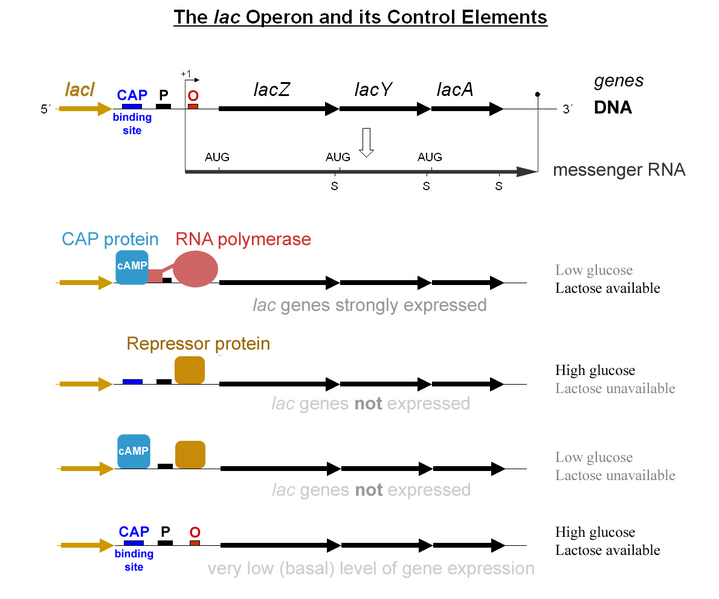
Crédito: G3pro (CC-BY 2.0)

LacI se enlazó a 2 secuencias operadoras de ADN.
Crédito: SocratesJedi (CC-BY-SA 3.0)

Simulación de Operón Lac
Inicie la simulación a continuación para explorar la activación coordinada del Operón Lac.
LacZ como gen reportero
pUC19 contiene ADN lacZ como gen reportero para ilustrar la presencia del gen funcional. La transcripción de este gen es impulsada por el sitio de unión para la subunidad de la ARN polimerasa llamado factor σ. El sitio de unión al factor σ determina la direccionalidad de la ARN polimerasa ya que existe la opción de transcribir en 2 direcciones. El sitio estándar de unión al factor σ a menudo se denota como -35 TTGACA... TATAAT -10 desde el inicio de la transcripción.
El sitio de clonación múltiple dentro del plásmido proporciona una ubicación conveniente para transportar una pieza extraña de ADN. Cuando no se inserta ADN extraño en este espacio, el producto del gen lacZ β-galactosidasa es funcional. La interrupción del marco de lectura para este gen también inhabilita la traducción del producto funcional. Mediante el uso de reporteros químicos, la integridad de este gen se puede confirmar a través de la actividad enzimática.
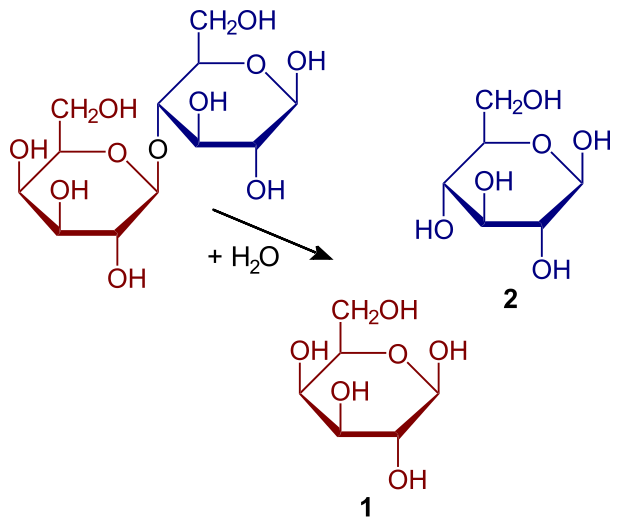
Hidrólisis de lactosa a galactosa y glucosa
Dos reporteros químicos utilizados para revelar la presencia de un LacZ funcional son X-Gal (5-Bromo-4-cloro-3-indolil-β-D-galactosido5-bromo-4-cloro-3-indolil-β-D-galactósido) y ONPG (ortonitrofenol).
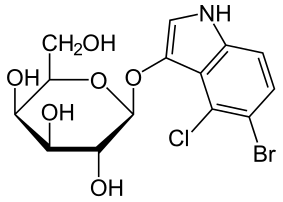
X-Gal (5-Bromo-4-cloro-3-indolil-β-D-galactósido) produce un color azul cuando se escinde por β-galactosidasa

ONPG (ortonitrofenol) produce un color amarillo tras la escisión por β-galactosidasa
Como en el caso del operón Lac, la LacI (proteína represora) ocupará al operador. Este operador pasa a estar superponiendo las secuencias -35 y -10. Para activar completamente estos genes, se debe eliminar el represor Lac uniéndose a un análogo de lactosa. En este caso, se utiliza el químico IPTG (Isopropil β-D-1-tiogalactopiranósido) ya que es un análogo no fragmentable que se unirá perpetuamente al represor Lac.

IPTG
Cribado Azul-Blanco

Blue-White Screening revela un fenotipo de bacterias transformadas basado en la capacidad de conversión de X-Gal por β-lactamasa Crédito: Stefan Walkowski (CC-BY-SA 4.0)
Referencias
- JACOB F, MONOD J. Mecanismos reguladores genéticos en la síntesis de proteínas. J Mol Biol. 1961 jun; 3:318-56.


