12.5: Transposones
- Page ID
- 56387
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Elementos Transposables
Los elementos genéticos móviles llamados elementos transponibles o transposones se localizan en todo el genoma. Estos elementos fueron descritos por primera vez en maíz por Barbara McClintock en el Laboratorio Cold Spring Harbor donde observó una alteración de la coloración en los granos de maíz que no siguió la simple herencia mendeliana.
Granos de Maíz McClintock
Cada núcleo representa un nuevo organismo individual distinto. El color del grano se describe a través de la simple herencia mendeliana donde el púrpura es dominante sobre el amarillo. El Dr. McClintock notó que algunos granos contenían manchas. Ella notó que la alteración de la coloración podría revertirse más tarde en generaciones posteriores. Describió el fenómeno de esta ruptura en las características mendelianas como una “inestabilidad genética”. Con el tiempo, llegaría a darse cuenta de que las manchas en estos granos surgieron de la inserción de ADN en el área de genes que estaban involucrados en el control de la coloración del núcleo.
La descripción del Dr. McClintock sobre este fenómeno y los mecanismos subyacentes fue extremadamente impopular ya que violó lo que ya se sabía sobre la fijedad de la genética. Aunque inicialmente escéptica, la biología ha descubierto que estos “genes saltarines” se encuentran en todos los taxones, incluidos los procariotas (donde a menudo se asocian con genes que confieren resistencia a los antibióticos) y luego fue galardonada con el Premio Nobel. Aproximadamente la mitad del genoma humano consiste en transposones, constituyendo la mayor parte de lo que antes se denominaba “ADN basura”. (Ver Animación: http://www.dnaftb.org/32/animation.html )
Video Avanzado de Disrupción Génica al Saltar Genes
Clases de Transposones
Los transposones pueden ser autónomos o no autónomos. Los transposones autónomos codifican su propia enzima transposasa que facilita el salto del gen mientras que los transposones no autónomos requieren la actividad transposasa de otro elemento transponible. Los transposones funcionales de ADN son autónomos y funcionan a través de un mecanismo de “cortar y pegar”. Los transposones de ADN son delineados por repeticiones terminales flanqueantes que marcan la ubicación en la que la transposasa extirpa el ADN. Estos elementos de ADN luego se reintegran en una ubicación diferente dentro del genoma. La escisión del ADN deja marcas de estas repeticiones flanqueantes que pueden ser utilizadas para estudiar la tasa y el nivel de eventos de transposición de ADN dentro de un genoma. La inserción de estos transposones puede afectar la expresión de genes cercanos y puede alterar completamente los genes en los que aterrizan como se evidencia en los granos de maíz moteados que McClintock describió.
Los transposones de ARN se denominan retrotransposones porque se transcriben en un ARNm y requieren una transcripción inversa para integrarse en el genoma. El elemento móvil más común en el genoma humano son los Elementos Nucleares Intercalados Largos (LINES) y los Elementos Nucleares Intercalados Cortos (SINE). Estos retrotransposones están representados de manera más abundante por los elementos autónomos LINE1 (L1) y Alu no autónomos, respectivamente. Los elementos de Alu se basan en la expresión de la L1 para ser transcritos inversos e integrados en el genoma. Estos retrotransposones funcionan en un mecanismo de “copiar y pegar” y son responsables de la expansión genómica. Como significan sus clasificaciones, las LÍNEAS son más largas que los SINE. Esto se debe a la presencia de un segundo marco de lectura que codifica la transposasa.
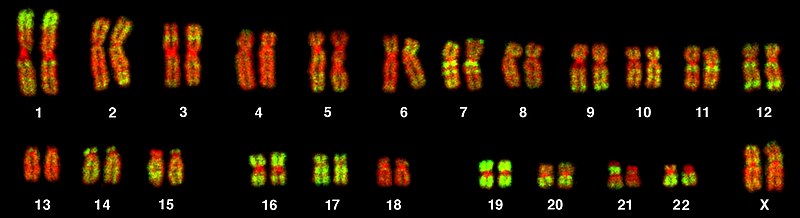
El Genoma Humano está plagado de ADN “basura”. La coloración verde corresponde a las secuencias de Alu en este cariograma del genoma humano.
Crédito: Andreas Bolzer, Gregor Kreth, Irina Solovei, Daniela Koehler, Kaan Saracoglu, Christine Fauth, Stefan Müller, Roland Eils, Christoph Cremer, Michael R. Speicher, Thomas Cremer (CC-BY 2.5)



