1.15: Desviaciones de Genética Mendeliana- Vinculación (Parte 2)
- Page ID
- 56646
Al finalizar esta lección, usted será capaz de:
- Hacer predicciones sobre herencia usando distancias unitarias de mapa y marcadores genéticos.
- Ensamble mapas a partir de datos de enlace de múltiples puntos.
- Definir la relación entre mapas de ligamiento, grupos de ligamiento y mapas genómicos.
- Describir cómo se observan y utilizan ADN o marcadores moleculares en el mapeo genético.
En esta lección aprenderá a hacer predicciones sobre herencia usando distancias unitarias de mapa y marcadores genéticos, ensamblar mapas a partir de datos de enlace de múltiples puntos, definir la relación entre mapas de ligamiento, grupos de enlace y mapas genómicos, y describir cómo se observan y usan ADN o marcadores moleculares en genes cartografía.
Introducción
¿Por qué los genetistas mapean genes? Esta es una pregunta legítima después de toda esta discusión. El mapeo genético ayuda a los genetistas a hacer dos cosas, hacer mejores predicciones y aislar genes. El siguiente es un ejemplo de cómo el mapeo genético puede ayudar a los criadores a hacer mejores selecciones.
Mapas de genes y selección
Este ejemplo muestra cómo el mapeo genético tiene una aplicación práctica. La soja tiene flores perfectas que se autopolinizan. Se requiere una tediosa manipulación de flores para hacer una semilla híbrida. A los criadores de soya les gustaría encontrar formas de hacer semillas híbridas en cantidades comerciales. Han descubierto un gen que provoca la falta de formación de polen y hace que las plantas sean masculinas estériles. Por lo tanto, cuando las abejas visiten estas plantas, realizarán una polinización cruzada y producirán semillas híbridas si transportan polen de una planta cercana con fertilidad masculina. Se podría establecer un campo de producción de semillas híbridas si pudieran tener todas las plantas estériles masculinas en una fila y fértiles masculinas en la siguiente. El control genético de este rasgo se muestra a continuación:
Plantas masculinas fértiles: MM o Mm
Estéril macho: mm
¿Ves el problema que uno tendría en la generación de semillas que son todas estériles masculinas (genotipo mm)? Estas plantas nunca serán verdaderas reproductoras porque no pueden autopolinizarse (tienen partes femeninas de trabajo pero no polen). Las únicas plantas que pueden autopolinizarse para producir mm de descendencia son las plantas Mm. La progenie de plantas Mm autofecundadas segregará 3 fértiles a 1 estéril, haciendo imposible obtener una colección pura de semillas mm para plantar en una fila 'hembra' para la producción de semillas híbridas. Si el genotipo mm pudiera recogerse en semillas antes de plantar, esto resolvería el problema. Desafortunadamente, el rasgo estéril masculino es imposible de seleccionar en la semilla pero los criadores descubrieron un truco que podían usar que aprovechaba su conocimiento de los mapas genéticos. Un segundo rasgo en la soya que es fácil de seleccionar en semillas está controlado por un gen que estaba estrechamente vinculado al gen M, m macho estéril. Este rasgo es la capa de semilla verde vs amarilla. El rasgo de recubrimiento de semilla se controla de la siguiente manera:
GG o Gg: verde
gg: amarillo
El locus G, g y el locus M, m han sido mapeados y están separados por dos unidades de mapa. Por lo tanto, podemos aprovechar esta vinculación para seleccionar semillas de un cruce que tenderá a ser también macho estéril. Así funcionaría el proceso. Se realiza la siguiente cruz:
GGMM (verde, macho-fértil) X ggmm (amarillo, macho estéril)
El dihíbrido cis (GM/gm) será macho-fértil y puede ser autopolinizado. Debido a la vinculación, los gametos GM y gm parentales se hacen 98% del tiempo. Por lo tanto, las semillas que son gg y amarillas casi siempre van a ser mm y estériles masculinas. Si el criador ordena las semillas F 2 en función del color, aproximadamente una cuarta parte de las semillas serán amarillas. Con base en la distancia unitaria de 2 mapas, aproximadamente 0.24 sería la frecuencia esperada de ggmm de todos los F 2 pero 96% del F 2 amarillo será estéril macho (0.24 de 0.25). Por lo tanto, 96 de cada 100 semillas sembradas en la fila 'hembra' de semillas amarillas seleccionadas serán estériles masculinas (mm). La recolección de semillas de estas hileras proporcionará un alto porcentaje de semillas híbridas. Así, el criador aprovechó la vinculación para seleccionar por un rasgo que era fácil de detectar (semillas amarillas) y obtener individuos con un rasgo que era imposible de seleccionar (masculino-estéril). El poder de este potencial de predicción ha impulsado gran parte de los esfuerzos recientes en el mapeo genético en plantas de cultivo, ganado y humanos.
0.49 grande G, grande M
0.01 grande G, pequeño m
0.01 poco g, grande M
0.49 poco g, poco m
0.49 grande G, grande M
0.01 grande G, pequeña M
0.01 grande G, pequeño m
0.01 poco g, grande M
0.0001
0.0049
0.49 poco g, poco m
0.0049
0.2401 (seleccionado)
Cruce de prueba de tres puntos: mapeo génico de múltiples puntos
Los mapeadores de genes están motivados para mapear todas las decenas de miles de genes que se encuentran en los cromosomas de plantas o animales. Analizar datos de cruces para determinar distancias de mapa para dos genes a la vez hace que el proceso sea lento y tedioso. Por lo tanto, los genetistas a menudo intentarán mapear tantos genes como sea posible a partir de un solo conjunto de progenie. Pasaremos por el ejemplo más simple de mapeo de puntos múltiples, un cruce de prueba de tres puntos, para demostrar este proceso.
Volveremos a usar nuestros tres loci de rasgos de semilla de maíz y usaremos datos de un cruce para mapear estos tres loci. El primer paso sería obtener un individuo trihíbrido que sea heterocigoto en los tres loci y luego realizar un cruce de prueba con este trihíbrido.
Padres: CCSswW (CSW/CSW) X ccSSwW (CSW/CSW)
Trihíbrido: CCSSW (CSW/CSW) X ccssww (csw/csw)
|
Rasgo de semilla |
Gamete de trihíbrido
Número
Rojo, encogido, normal
CsW
2777
Blanco, regordete, ceroso
CsW
2708
Rojo, regordete, ceroso
CsW
116
Blanco, encogido, normal
CsW
123
Rojo, encogido, ceroso
Csw
643
Blanco, regordete, normal
CsW
626
Rojo, regordete, normal
CSW
4
Blanco, encogido, ceroso
csw
3
Número total de progenie:
7000
Observamos ocho fenotipos de semillas en la progenie de testcross porque el trihíbrido puede hacer ocho tipos de gametos. Como sabíamos, estos tres genes están vinculados y así la proporción desigual de fenotipos refleja las combinaciones de dos gametos parentales y seis recombinantes. Los gametos parentales siguen siendo el tipo más frecuentemente producido, y los seis gametos recombinantes se hacen cuando se produce el cruce entre los loci. Podemos dar cuenta sistemáticamente de estos crossovers si seguimos los siguientes cuatro pasos. Primero, explicaremos los pasos y luego mostraremos cómo usarlos para mapear los tres genes.
Paso 1: Identificar los gametos parentales.
Estas son CsW y CsW, esa combinación que vino de la generación parental y la combinación hecha por el trihíbrido a la frecuencia más alta.
Paso 2: Clasificar los recombinantes.
Si observamos la combinación de genes en cada uno de los seis gametos recombinantes, podemos preguntarnos si el gameto tiene una nueva combinación para cada par de genes. Por ejemplo, CsW tiene una nueva combinación para el CS y el Cw en comparación con los gametos parentales pero la misma combinación para Sw que el gameto parental CsW. Por lo tanto, estos 116 gametos representan cruces entre C, c y S, s así como C, c y W, s. Esta información se registra (ver abajo).
Paso 3: Determinar la frecuencia de gametos recombinantes.
Una vez clasificados los seis gametos recombinantes, se puede sumar el número total de cruces entre los tres loci y determinar el porcentaje de cruce entre cada par de loci. Este número reflejará la distancia génica pero se necesita un paso más para completar el proceso.
Paso 4: Añadir en los gametos de doble cruce.
Dos de los seis gametos recombinantes se realizaron como resultado de cruzamientos dobles entre los dos loci que están más alejados. Estos cruces se han agregado al mapa distancias entre el locus medio y los dos loci externos. Ahora necesitamos agregar estos cruces dobles a la distancia loci exterior.
Aplicar estos cuatro pasos a nuestros datos cruzados de prueba de tres puntos funcionaría de la siguiente manera:
- CsW y CsW
- C, c a S, s: 116 (CsW) + 123 (CsW) + 4 (CSW) + 3 (csw) = 246
C, c a W, w: 116 (CsW) + 123 (CsW) + 643 (Csw) + 626 (CsW) = 1508
S, s a W, w: 643 (Csw) + 626 (CsW) + 4 (CSW) + 3 (csw) = 1276 - 246/7000 = 3.5% gametos recombinantes C, c a S, s 1508/7000 = 21.5% gametos recombinantes.
C, c a W, w 1276/7000 = 18.2% gametos recombinantes S, s a W, w. - De la información del paso 3 podemos ver que los loci C, c y W, w son los más alejados por lo que el locus S, s debe estar entre ellos. También podemos ver que hemos subestimado la distancia entre los loci exteriores (3.5 C, c a S, s + 18.2 S, s a W, w = 21.7 unidades de mapa no 21.5). Si bien esta diferencia es pequeña, podemos rectificar esto y volver a verificar nuestro trabajo agregando en los dobles crossovers. Los gametos CSW y csw se hacen muy raramente. Eso se debe a que se necesitan dos cruces en el cromosoma del trihíbrido para hacerlos, no solo uno.
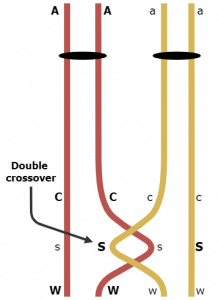
Cuando se producen dos cruces entre los loci C, c y W, se restauran las combinaciones parentales de CW y cw. Por lo tanto, no se contabilizaron los gametos CSW y CSW como representativos de crossovers cuando en realidad representan dos crossovers cada uno (Figura 1). Así, debemos sumar los cruces dobles (3 + 4 = 7), multiplicar por dos y sumar estos 14 cruces a la distancia C, c a W, w (1508 + 14 = 1522/7000 = 21.7 unidades del mapa). Ahora hemos revisado dos veces las distancias de nuestro mapa y tenemos nuestro mapa de tres puntos completo.
Puede resultar intimidante ver todos los datos generados a partir de un cruce de prueba de tres puntos. Esta información, sin embargo, puede ser utilizada sistemáticamente para ahorrar tiempo a los genetistas y mapear tres genes en un solo experimento. Los datos de tres puntos también proporcionan una medida más precisa de las distancias del mapa en comparación con los datos de dos puntos cuando los genes están más separados en un cromosoma. Esto se debe a que un tercer gen entre los loci más distantes puede dar cuenta de cruces dobles que no serían detectables en un análisis de dos puntos. Por esta razón, las distancias del mapa tienden a subestimarse cuando los genes están más separados como vimos en un grado modesto en este ejemplo (21.5 de dos puntos vs 21.7 de tres puntos).

¿Cómo trabajaría un genetista con un cruce de prueba de cuatro puntos? Habría dieciséis fenotipos en la progenie como resultado de que el progenitor híbrido hiciera dos gametos parentales y catorce gametos recombinantes. Estos gametos recombinantes serían el resultado de cruces simples, dobles e incluso triples ocurridos en la profase I. Si bien sería tedioso trabajar con los datos, se podría analizar un conjunto de datos para revelar el mapa de cuatro genes. No es de extrañar que los genetistas ahora hayan escrito programas de computadora que realizarán este tipo de cálculos, ahorrarán tiempo y reducirán la posibilidad de error en la calculadora. Los genes de mapeo del genetista en plantas y animales económicamente importantes también generarán poblaciones cartográficas que son resultado del cruce de padres que tienen diferentes alelos en cientos o miles de loci. A pesar de que el desarrollo de computadoras y programas se ha convertido en parte del mapeo genético moderno, el proceso de medir indirectamente la frecuencia de cruce mediante la observación de la herencia de combinaciones de rasgos es la base para generar estos mapas genéticos.
Mapas de enlace, grupos de enlace
El organismo en el que se realizó el mapeo genético por primera vez fueron las moscas de la fruta Trabajar con moscas de la fruta dio muchas ventajas a los primeros mapeadores de genes. Primero, los genetistas de la mosca de la fruta observaron una gran variación genética entre las moscas de la fruta por rasgos de parte del cuerpo (tamaño del ala, forma y color de los ojos, tipos de cerdas de las piernas, etc.) que eran fáciles de ver si tenían un microscopio de baja potencia. Las variantes de fenotipo surgieron de forma natural o podrían ser inducidas por mutagénesis química o por radiación. Adicionalmente, el corto tiempo de generación, el gran número de crías y los bajos costos de crianza les permitieron completar experimentos de vinculación informativa en poco tiempo. Finalmente, los citogenetistas sabían que las moscas de la fruta tenían cuatro pares de cromosomas por lo que todos los genes se mapearían en cuatro grupos de enlace. Un cromosoma era muy pequeño, y pocos genes mapeados a este cromosoma. El cromosoma X y el autosoma más grande tienen muchos genes y más de 100 unidades de mapa separan genes en extremos opuestos del cromosoma.
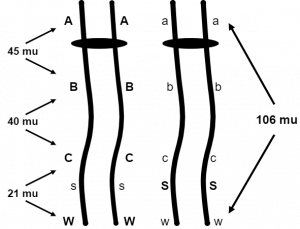
Detengámonos y pensemos en cómo determinaron esta distancia de unidad de mapa grande. Si los loci A, a y W, w están separados por 106 unidades de mapa, ¿con qué frecuencia un dihíbrido AW/aw elaborará los gametos Aw y aW? Dado que estos son los gametos recombinantes y la distancia del mapa es la frecuencia de los gametos recombinantes, estamos tentados a decir 106% pero eso es imposible. Los gametos parentales para cualquiera de dos loci nunca se harán menos del 50% del tiempo, el porcentaje que observamos cuando dos loci están surtiendo independientemente. De hecho, el 50% es la respuesta correcta aquí. Cada vez que los genes que están en el mismo grupo de ligamiento estén separados por 50 unidades de mapa o más, se comportarán como si fueran independientes entre sí. Eso se debe a que los genes están lo suficientemente separados como para permitir siempre que se produzca al menos un cruce entre ellos durante la profase I. Entonces, ¿cómo podemos determinar que A, a y W, w están separadas 106 unidades de mapa? Simplemente determinando las distancias entre estos genes y otros genes entre ellos que están separados por menos de 50 unidades cartográficas (Figura 3). La Figura 3 muestra que se pueden encontrar distancias entre A, a y B, b, B, b y C, c, luego C, c y W, w. Cada una de estas distancias se suma para llegar a las 106 unidades de mapa entre A, a y W, w. Por lo tanto, si el genetista combina la información de mapeo de muchos experimentos de ligamiento o realiza un análisis de ligamiento de múltiples puntos en un grupo de progenie que segrega para muchos genes enlazados, puede deducir las distancias de unidades de mapa más grandes.
Rasgos limitados, mapas de vinculación limitada
Muchos mapeadores de genes no disfrutan de las ventajas que tienen los genetistas de la mosca de la fruta en el mapeo de genes. El ganado, por ejemplo, tiene treinta grupos de ligamiento (2n = 60), a menudo tienen una descendencia por cruce, y es difícil observar cientos de diferencias de fenotipo controladas por genes individuales entre el ganado bovino. La soya tiene 20 grupos de ligamiento pero durante muchas décadas, los genetistas de soya habían compilado alrededor de 30 mapas de ligamiento diferentes y no tenían idea de qué mapas formaban parte realmente del mismo grupo de enlace. Los genetistas de soya necesitaron descubrir más genes que tuvieran claros efectos de fenotipo y luego realizar cruces que compararan la herencia de estos nuevos genes con los genes que ya habían sido mapeados. En los años 1970 y 1980, se descubrió un nuevo tipo de rasgo genético, los marcadores moleculares, que aceleraron enormemente el proceso de mapeo genético en todos los organismos.


