18: Regulación transcripcional después del inicio
( \newcommand{\kernel}{\mathrm{null}\,}\)
Aunque la regulación del inicio de la transcripción parece ser un factor dominante en el control de la expresión de muchos genes, la importancia de la regulación después de la iniciación es cada vez más apreciada en un número y variedad crecientes de sistemas. Los sistemas clásicos en los que se han explorado estos temas son la antiterminación en el bacteriófago l y en la atenuación de la transcripción en operones biosintéticos bacterianos, en particular el operón trp en E. coli. Aunque algunos de los detalles mecanicistas pueden ser peculiares de las bacterias, especialmente la necesidad de una transcripción y traducción acopladas en el sistema de atenuación trp, el fenómeno de regulación después de la iniciación se observa en una amplia variedad de organismos, que van desde bacterias hasta humanos. Parte de este trabajo se discutió en las secciones sobre elongación de la transcripción del Capítulo II de la Tercera Parte.
Introducción
Ambos sistemas discutidos en este capítulo controlan la frecuencia de terminación de la transcripción. La antiterminación en el bacteriófago l puede evitar que la ARN polimerasa se detenga en terminadores dependientes de r, conduciendo así a la transcripción de genes aguas abajo. La atenuación en el operón trp también controla la frecuencia a la que la ARN polimerasa se detiene en un terminador temprano en el operón, regulando así la transcripción de genes aguas abajo. A diferencia del sistema en l, la atenuación en trp regula la terminación en un terminador r-independiente.
Antiterminación en Bacteriófago l
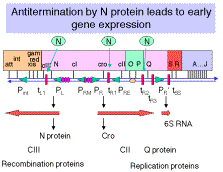
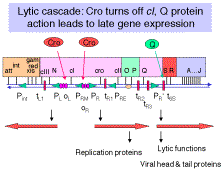
Sólo para revisar rápidamente uno de los puntos del Capítulo III, la antiterminación ocurre en dos momentos diferentes del ciclo vital. La proteína N permite la transcripción de lectura en el cambio de la transcripción inmediata temprana a la temprana, y la proteína Q permite la transcripción de lectura completa de los genes tardíos.
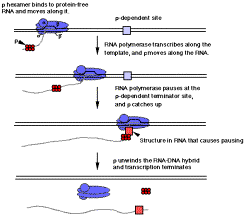
Recordemos de la tercera parte del texto que los terminadores r-dependientes no tienen una secuencia bien conservada o estructura secundaria. Además, la proteína r rastrea a lo largo de regiones libres de proteínas del ARN hasta que golpea un complejo transcripcional pausado en un sitio dependiente de r, momento en el que su actividad de ARN helicasa puede causar la terminación y disociación de la polimerasa y transcripción del ADN molde.

Los sitios en el ADN necesarios para la antiterminación en bacteriófago l. Los sitios de nuez (sitios de N ut ilización) para pN, sitio qut para pQ y los sitios de nuez están dentro de la unidad de transcripción, no en el promotor y no en el terminador.
- NutL está en la región 5' no traducida del gen N, y nutR está en la región 3' no traducida del gen cro.
- En ambos casos, el sitio de la tuerca precede al terminador en el que actuará pN.

Tanto NutL como NutR son secuencias de 17 pb con simetría díada.
5' A GCCCT GAARAAGGGCA
TCGGGACTTYT TCCCG T 5'
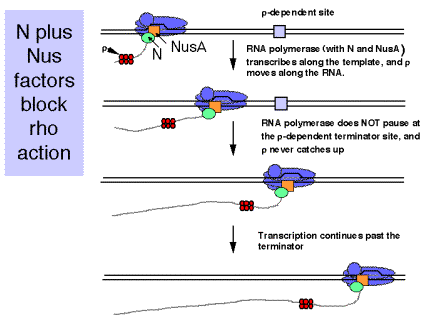
La proteína pN reconoce el sitio de la nuez y se une a la ARN polimerasa a medida que se transcribe a través del sitio. El complejo de pN con las ARN polimerasas es altamente procesivo y anula los esfuerzos de r en el terminador.
Proteínas de E. coli (huésped) necesarias para la acción de pN. Éstas se aislaron como funciones hospedadoras que al mutarse impidieron la acción de pN. La nuSA (codificada por NuSA, para la ubstancia de la utilización de N u, el grupo de complementación A) es la mejor caracterizada.
- Puede formar parte del complejo de transcripción
- Se ha propuesto unirse al núcleo de la ARN polimerasa después de la disociación de s.
- También puede unir pN.
- Modelo:
NuSA se une a la polimerasa central después de que s se disocia. Como este complejo se transcribe a través de un sitio de nuez, pN se une también. El complejo a2bB'-NUSA-PN evita la terminación dependiente de r en tR1, tR2 y TL1.
Se han identificado varios otros genes nus. NuSG es el homólogo bacteriano de una familia de proteínas conservadas involucradas en la elongación. Es homóloga a la subunidad grande del DSIF, que es un factor de elongación en mamíferos. El DSIF es el factor inductor de sensibilidad al DRB. Los estudios actuales lo implican tanto en efectos negativos como positivos sobre la elongación. Tiene dos subunidades, una de 160 kDa que es homóloga a la proteína reguladora transcripcional de levadura Spt5, y una de 14 kDa que es homóloga a la proteína Spt4 de levadura. Otro gen nus codifica una proteína ribosómica. Se necesita aprender mucho más tanto sobre la terminación como sobre la antiterminación. El fenotipo nus de mutaciones en un gen que codifica una proteína ribosómica sugiere que la traducción también se acopla a este proceso.
Componentes del operón trp de E. coli
El operón trp codifica las enzimas necesarias para la biosíntesis de triptófano. Más específicamente, sus cinco genes (trpedCBA) codifican cinco subunidades de proteínas que en total catalizan cinco etapas enzimáticas, convirtiendo el ácido corismico en triptófano. Sin embargo, no existe una correspondencia 1:1 entre un cistrón y una enzima. Por ejemplo, TrpB y TrpA codifican, respectivamente, las subunidades b y a de la triptófano sintasa, que cataliza la sustitución de glicerol-3-fosfato de indol-3-glicerol-fosfato con serina para formar triptófano, con gliceraldehído-3-fosfato como el otro producto
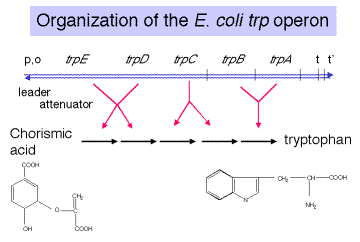
Una secuencia líder separa el promotor y el operador del primer gen estructural del operón, trpE. Un atenuador de transcripción sigue al líder. Como veremos con más detalle a continuación, la eficiencia de la terminación “prematura” en este atenuador está determinada por el grado de traducción del líder, que a su vez está determinado por la disponibilidad de Trp-TrnaTP. Esta es una parte importante de la regulación del operón. Dos terminadores de la transcripción siguen los genes estructurales, uno dependiente de r y otro independiente de r.
Modos de regulación: apagar el operón en presencia de Trp
Represor-operador: requiere una unión proteica a un sitio específico en presencia de Trp para disminuir la eficiencia de inicio de la transcripción. Atenuación: la elongación (y terminación) de la transcripción por la ARN polimerasa está ligada al progreso de la traducción por un ribosoma. En presencia de Trp, la traducción por el ribosoma provoca la terminación de la transcripción de los genes posteriores en el operón.
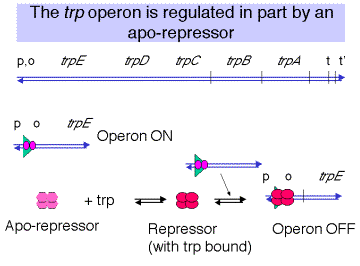
Represor: apo-represor y co-represor (Trp)
El apo-represor es codificado por TrpR en un locus distante. El apo-represor es un homo-tetrámero. Tiene una alta afinidad por el operador solo cuando está unido por el aminoácido Trp, que sirve como co-represor. Así, el represor activo es un tetrámero de (anteriormente apo-) represor en complejo con Trp. El represor activo se une al operador para evitar el inicio de la transcripción. El operador se solapa con el promotor, incluyendo la región -10 del promotor. Tiene un eje de simetría díada.
Atenuación
El atenuador es un terminador transcripcional condicional utilizado para regular la expresión de operones biosintéticos en bacterias. Está aguas arriba de los genes estructurales trpedCBA y es un sitio de terminación independiente de r. Su capacidad para terminar la transcripción depende de su capacidad para formar el tallo del ARN dúplex que es característico de los sitios de terminación independientes de r.
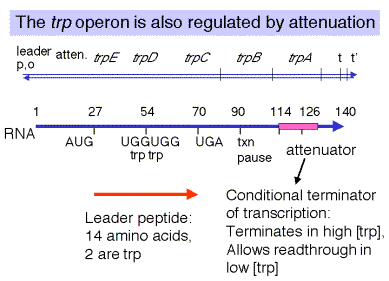
La fracción de transcritos que leen a través del atenuador está determinada por el [Trp-trNATP]. La concentración de ARNt cargados es una medida de la cantidad de Trp disponible para la síntesis de proteínas. Si se cobra la mayor parte de TrnaTP, hay abundancia de Trp, y la célula no necesita hacer más. Bajo [Trp-TrNATRP] permite la transcripción de lectura a través del atenuador, de modo que TrpedCBA se expresa y [Trp-TrnaTP] alto provoca la terminación de la transcripción en el atenuador.
El [Trp-TrNATRP] determina el progreso de los ribosomas a medida que traducen un péptido líder corto. El péptido líder es un polipéptido corto de 14 aminoácidos codificado por TrPL. Dos codones para Trp están en el líder, y el progreso de los ribosomas más allá de estos codones Trp estará determinado por la disponibilidad de Trp-TrnaTP. Cuando la concentración de triptofanil-ARNt sea alta, se completará la traducción del líder trp, pero cuando sea baja, la traducción se detendrá en los codones de triptófano.
El grado de progreso de los ribosomas determina las estructuras secundarias formadas en el ARN líder. Cuando el [Trp-TrnaTP] es alto, los ribosomas se traducen más allá de los codones Trp para completar el líder de síntesis del péptido. Esto permite que el ARN naciente forme la estructura para el terminador r-independiente. Así, la transcripción termina antes de que la ARN polimerasa alcance trpedCBA. Cuando el [Trp-trNATP] es bajo, los ribosomas se detienen en los codones Trp, lo que impide la formación de las estructuras secundarias en el ARN necesarias para la terminación en el atenuador. Así, la transcripción de lectura continua a través de TrpedCBA y se expresa el operón, de manera que se hace más Trp.
| [TRP-ARNt] | traducción de TrPL | estructuras secundarias formadas en ARN | Atenuador | Operón |
|---|---|---|---|---|
| Alto | completar | 3-4 tallo | terminar la transcripción | OFF |
| Bajo | puestos en codones Trp | 2-3 tallo | permitir la transcripción de lectura | EN |
Estructuras alternativas de pares de bases en ARN líder. Cuatro regiones del ARN líder pueden estar involucradas en la formación de estructuras secundarias, en particular los tallos con pares de bases; estos se denominan simplemente regiones 1, 2, 3 y 4. Potencialmente, 1 puede emparejarse con 2, 2 pueden emparejarse con 3 y 3 pueden emparejarse con 4.
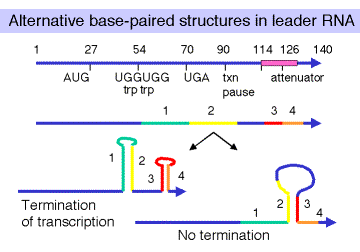
Un tallo formado por emparejamiento entre 3 y 4 hace un tallo rico en G+C seguido de U, lo que es suficiente para la terminación de la transcripción independiente de r. Cuando el [Trp-trNATRP] es alto, se forma la estructura pareada de 3-4 bases y la transcripción termina en el atenuador. Esto apaga el operón. La formación de un tallo con pares de bases entre las regiones 2 y 3 impide la formación del terminador 3-4, y la transcripción continuará en los genes estructurales trpedCBA. Esto enciende el operón.
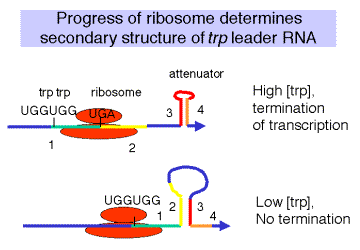
La elección entre un tallo 2-3 o un tallo 3-4 viene dictada por el progreso del ribosoma. Si el ribosoma puede traducirse más allá de los codones Trp (cuando el [Trp-TrnaTP] es alto), entonces alcanzará un codón natural de terminación de la traducción. Cuando el ribosoma está en esa posición, la región 2 del ARN líder está cubierta por el ribosoma, por lo que el tallo 2-3 no puede formarse pero el tallo 3-4 sí. Esto genera la estructura secundaria necesaria para la terminación de la transcripción en el atenuador. En contraste, si el ribosoma se detiene en los codones Trp en el líder, debido a que el [Trp-TrnaTP] es bajo, entonces la región 2 del ARN líder no está cubierta por el ribosoma. Luego puede emparejar bases con la región 3. Esto evita la formación del terminador 3-4, y la ARN polimerasa puede continuar la elongación a través de TrpedCBA.
Análisis mutacional (ejemplos seleccionados)
- La traducción de TrPL es necesaria para la regulación por atenuación. La mutación del AUG para el inicio de la traducción del ARN líder impide la transcripción más allá del atenuador. En ausencia de traslación, se pueden formar tanto los tallos 1-2 como los 3-4. Este último 3-4 tallo es el terminador.
- Se requiere TrnaTP cargado para la regulación. La mutación de los genes para trnaTRP o Trp-trNATRP sintetasa conduce a la expresión constitutiva de trpedCBA. En estos mutantes, la traducción se detendrá en los codones Trp independientemente del [Trp] intracelular, y no se formará ningún terminador en el atenuador.
- Se necesitan estructuras secundarias específicas en el ARN líder trp para la regulación. Por ejemplo, las mutaciones que disminuyen el número de pares de bases entre las regiones 3 y 4 disminuirán la cantidad de terminación transcripcional (es decir, aumentarán la expresión del operón). Las mutaciones compensatorias que incrementen el número de pares de bases entre 3 y 4 suprimirán las mutaciones originales.
La atenuación requiere transcripción y traducción acopladas
No requiere proteínas reguladoras: la carga del ARNt cognado es la señal reguladora. Se necesita un sitio de pausa transcripcional en +90 para permitir que los ribosomas alcancen la ARN polimerasa y así afectar las estructuras secundarias en el ARN naciente.
La atenuación es un mecanismo común para regular operones biosintéticos
Muchos operones que codifican las enzimas que catalizan la biosíntesis de aminoácidos están regulados por la atenuación. En cada caso, el polipéptido líder es rico en el aminoácido que es el producto de la vía, por ejemplo his, phe, leu, thr, ilv.
Lecturas adicionales
- Friedman, D.I. y Count, D.L. (1995) Antiterminación transcripcional: El paradigma lambda actualizado. Microbiología Molecular 18:191-200.
- Henkin, T. (2000) Terminación transcripcional en bacterias. Opiniones Actuales en Microbiología 3:149-153.
- Gusarov, I. y Nudler, E. (2001) Control de la terminación transcripcional intrínseca por N y NuSA: El mecanismo básico. Celda 107:437-449.


