19: Genómica
- Page ID
- 56138
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)La genómica es un campo que estudia toda la colección del ADN o genoma de un organismo. Implica secuenciar, analizar y comparar la información contenida en los genomas. Dado que la secuenciación se ha vuelto mucho menos costosa y más eficiente, ahora se dispone de grandes cantidades de información genómica sobre una amplia variedad de organismos, pero particularmente microbios, con su tamaño de genoma más pequeño. De hecho, el mayor cuello de botella actualmente no es la falta de información sino la falta de potencia informática para procesar la información.
Secuenciación
La secuenciación, o la determinación del orden de base del ADN o ARN de un organismo, suele ser uno de los primeros pasos para encontrar información detallada sobre un organismo. Un genoma bacteriano puede variar de 130 pares de kilobases (kbp) a más de 14 pares de megabases (Mbp), mientras que un genoma viral varía de 0.859 a 2473 kbp. A modo de comparación, el genoma humano contiene alrededor de 3 mil millones de pares de bases.
Secuenciación escopeta
La secuenciación de escopeta implica inicialmente la construcción de una biblioteca genómica, donde el genoma se divide en fragmentos de tamaño aleatorio que se insertan en vectores para producir una biblioteca de clones. Los fragmentos son secuenciados y luego analizados por una computadora, que busca regiones superpuestas para formar un tramo más largo de secuencia. Finalmente, todas las secuencias se alinean para dar la secuencia completa del genoma. Los errores se reducen porque muchos de los clones contienen secuencias idénticas o casi idénticas, lo que resulta en una buena “cobertura” del genoma.
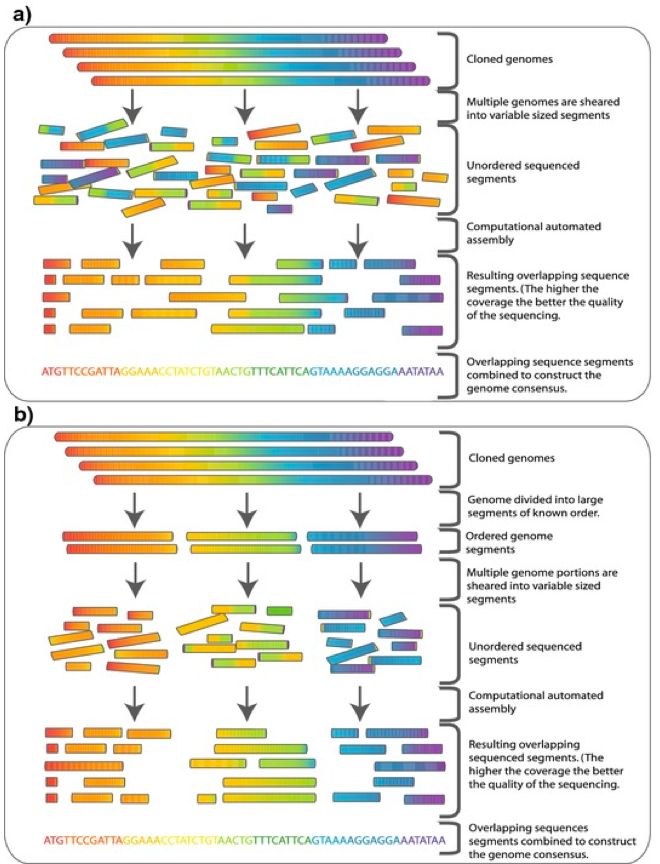
Secuenciación Escopeta. Por Commins, J., Taft, C., Fares, M. A. [CC BY-SA 2.5], vía Wikimedia Commons
Secuenciación ADN de segunda generación
La secuenciación de ADN de segunda generación utiliza métodos masivamente paralelos, donde múltiples muestras se secuencian lado a lado. Los fragmentos de ADN de unos pocos cientos de bases cada uno se amplifican por PCR y luego se unen a una pequeña perla, de manera que cada perla lleva varias copias de la misma sección de ADN. Las perlas se colocan en una placa que contiene más de un millón de pocillos, cada uno con una perla, y se secuencian los fragmentos de ADN.
Secuenciación de ADN de tercera y cuarta generación
La secuenciación de ADN de tercera generación implica la secuenciación de moléculas individuales de ADN. La secuenciación de ADN de cuarta generación, también conocida como “secuenciación post luz”, utiliza métodos distintos de la detección óptica para la secuenciación.
Bioinformática
Después de la secuenciación, es el momento de darle sentido a la información. El campo de la bioinformática combina muchos campos juntos (es decir, biología, informática, estadística) para utilizar el poder de las computadoras para analizar la información contenida en la secuencia genómica. La localización de genes específicos dentro de un genoma se conoce como anotación del genoma.
Marcos de Lectura Abiertos (ORFS)
Un marco de lectura abierto u ORF denota un posible gen codificante de proteínas. Para el ADN bicatenario, hay seis marcos de lectura a analizar, ya que el ADN se lee en conjuntos de tres bases a la vez y hay dos cadenas de ADN. Un ORF típicamente tiene al menos 100 codones antes de un codón de terminación, con secuencias terminadoras 3'. Un ORF funcional es aquel que en realidad es utilizado por el organismo para codificar una proteína. Las computadoras se utilizan para buscar la secuencia de ADN buscando ORF, con aquellos que se presume codifican proteínas analizadas posteriormente por un bioinformático.
A menudo es útil que la secuencia sea comparada con una base de datos de secuencias que codifican proteínas conocidas. GenBank es una base de datos de más de 200 mil millones de pares de bases de secuencias a las que los científicos pueden acceder, para tratar de encontrar coincidencias con la secuencia de interés. La herramienta de búsqueda de bases de datos BLAST (herramienta básica de búsqueda de alineación local) cuenta con programas para comparar tanto secuencias de nucleótidos como secuencias de aminoácidos, proporcionando una clasificación de resultados en orden decreciente de similitud.
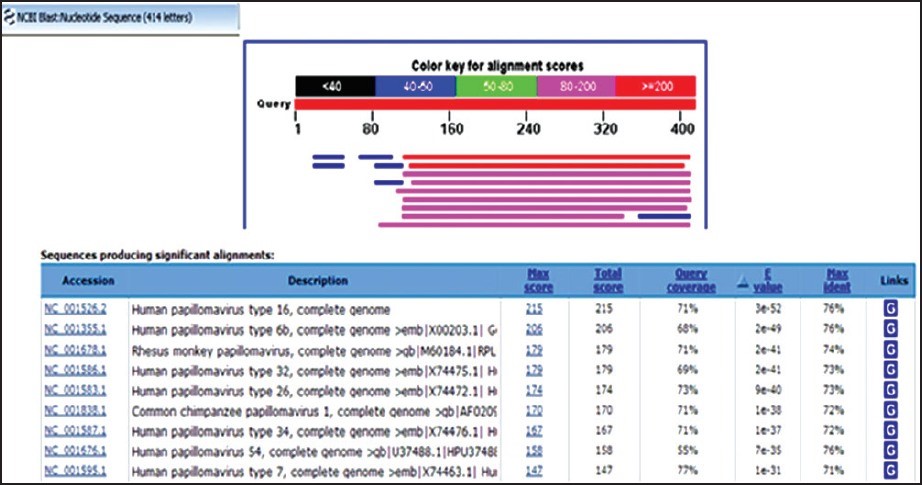
Resultados BLAST.
Genómica Comparada
Una vez obtenidas las secuencias de los organismos, se puede recabar información significativa utilizando genómica comparativa. Para ello se evalúan los genomas para obtener información sobre el tamaño, la organización y el contenido de genes.
La comparación del genoma de las cepas microbianas ha dado a los científicos una mejor imagen con respecto a los genes que captan los organismos. Un grupo de múltiples cepas comparten un genoma central, genes que codifican funciones celulares esenciales que todas tienen en común. El genoma pan representa todos los genes que se encuentran en todos los miembros de las especies, por lo que proporciona una buena idea de la diversidad de un grupo. La mayoría de estos genes “extra” probablemente son captados por transferencia génica horizontal.
La genómica comparativa también muestra que muchos genes se derivan como resultado de la duplicación génica. Los genes dentro de un solo organismo que probablemente surgieron debido a la duplicación génica se denominan parálogos. En muchos casos uno de los genes podría ser alterado para asumir una nueva función. También es posible que la duplicación de genes se encuentre en diferentes organismos, como resultado de adquirir el gen original de un ancestro común. Estos genes se llaman ortólogos.
Genómica Funcional
La secuencia de un genoma y la ubicación de los genes proporcionan parte de la imagen, pero para entender completamente un organismo necesitamos una idea de lo que la célula está haciendo con sus genes. En otras palabras, ¿qué sucede cuando se expresan los genes? Aquí es donde entra en juego la genómica funcional, colocando la información genómica en contexto.
El primer paso en la expresión génica es la transcripción o la fabricación de ARN. El transcriptoma se refiere a todo el complemento de ARN que una célula puede producir a partir de su genoma, mientras que el proteoma se refiere a todas las proteínas codificadas por el genoma de un organismo, en el paso final de la expresión génica.
Microarrays
Las micromatrices o chips génicos son soportes sólidos sobre los que se colocan múltiples manchas de ADN, en forma de cuadrícula. Cada mancha de ADN representa un solo gen u ORF. Los fragmentos conocidos de ácido nucleico se marcan y se utilizan como sondas, con una señal producida si se produce la unión. Las micromatrices se pueden usar para determinar qué genes podrían encenderse o apagarse en condiciones particulares, como comparar el crecimiento de un patógeno bacteriano dentro del hospedador frente al exterior del hospedador.
Proteómica
El estudio de las proteínas de un organismo (o el proteoma) se conoce como proteómica. Gran parte del interés se centra en la proteómica funcional, que examina las funciones de las proteínas celulares y las formas en que interactúan entre sí.
Una técnica común utilizada en el estudio de las proteínas es la electroforesis en gel bidimensional, que primero separa las proteínas en función de sus puntos isoeléctricos. Esto se logra mediante el uso de un gradiente de pH, que separa las proteínas en función de su contenido de aminoácidos. Las proteínas separadas se pasan luego a través de un gel de poliacrilamida, proporcionando la segunda dimensión a medida que las proteínas se separan por tamaño.
La proteómica estructural se centra en la estructura tridimensional de las proteínas, que a menudo se determina mediante el modelado de proteínas, utilizando algoritmos informáticos para predecir el plegamiento más probable de la proteína basándose en la información de aminoácidos y patrones de proteínas conocidos.
Metabolómica
Metabolómica se esfuerza por identificar el conjunto completo de intermedios metabólicos producidos por un organismo. Esto puede ser extremadamente complicado, ya que muchos metabolitos son utilizados por las células en múltiples vías.
Metagenómica
Metagenómica o genómica ambiental se refiere a la extracción de ADN agrupado directamente de un ambiente específico, sin el aislamiento e identificación inicial de organismos dentro de ese ambiente. Dado que muchas especies microbianas son difíciles de cultivar en el laboratorio, estudiar el metagenoma de un ambiente permite a los científicos considerar todos los organismos que puedan estar presentes. Los taxones pueden incluso identificarse en ausencia de aislamiento de organismos usando secuencias de ácido nucleico solas, donde el taxón se conoce como filotipo.
Palabras clave
genómica, secuenciación, secuenciación escopeta, biblioteca genómica, secuenciación de ADN de segunda generación, métodos masivamente paralelos, secuenciación de ADN de tercera y cuarta generación, bioinformática, anotación genómica, marco de lectura abierto/ORF, ORF funcional, GenBank, herramienta de búsqueda de alineación local Blast/básica, genómica comparada, genoma central, genoma pan, parálogo, ortólogo, genómica funcional, transcriptoma, proteoma, micromatriz/chips génicos, sonda, proteómica, proteómica funcional, electroforesis en gel bidimensional, proteómica estructural, metabolómica, metagenómica/genómica ambiental, metagenoma, filotipo.
Preguntas de Estudio
- ¿Qué engloba el campo de la genómica?
- ¿Qué es la secuenciación de escopeta y cómo permite esto la secuenciación completa del genoma de un organismo?
- ¿Cuáles son las diferencias básicas entre la secuenciación de segunda, tercera y cuarta generación?
- ¿Qué es un marco de lectura abierto y cómo pueden usarlo los científicos para determinar información sobre un genoma y sus productos?
- ¿En qué se diferencia la genómica funcional de la genómica comparada? ¿Cuáles son las herramientas utilizadas en la genómica funcional y qué información se puede obtener de cada una?

