22: Los virus
- Page ID
- 56053
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Clasificación Viral
Dado que los virus carecen de ribosomas (y por lo tanto ARNr), no pueden clasificarse dentro del esquema de Clasificación de Tres Dominios con organismos celulares. Alternativamente, el Dr. David Baltimore derivó un esquema de clasificación viral, uno que se centra en la relación entre un genoma viral y cómo produce su ARNm. El Esquema de Baltimore reconoce siete clases de virus.
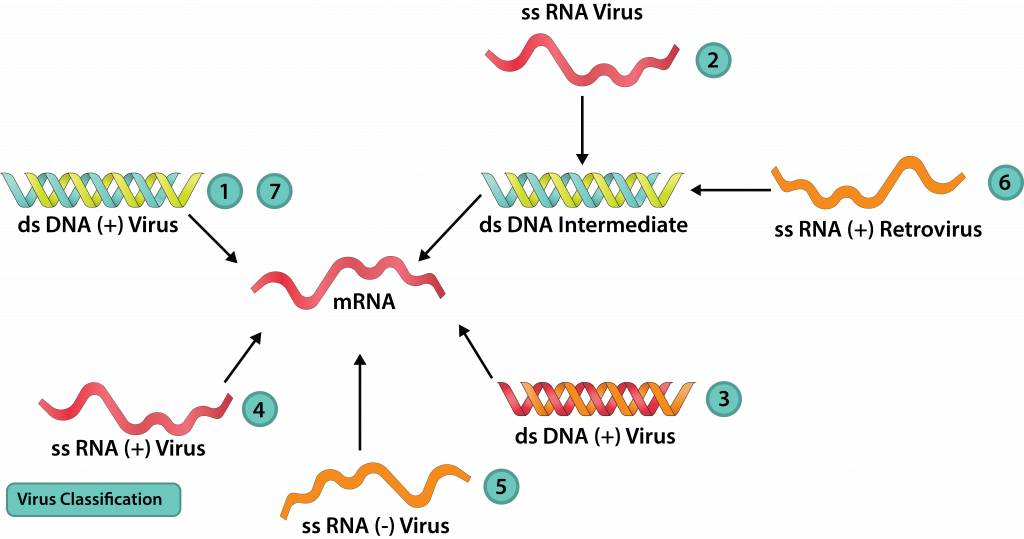
Virus de ADN
Clase I: dsDNA
Los virus de ADN con un genoma de ADNds, como los bacteriófagos T4 y lambda, tienen un genoma exactamente igual que la célula huésped que están infectando. Por esta razón, muchas enzimas hospedadoras pueden ser utilizadas para la replicación y/o producción de proteínas. El flujo de información sigue una vía convencional: ADNbc → ARNm → proteína, con una ARN-polimerasa dependiente de ADN que produce el ARNm y el ribosoma huésped que produce la proteína. La replicación del genoma, ADNds → ADNds, requiere una ADN-polimerasa dependiente de ADN del virus o de la célula hospedadora.
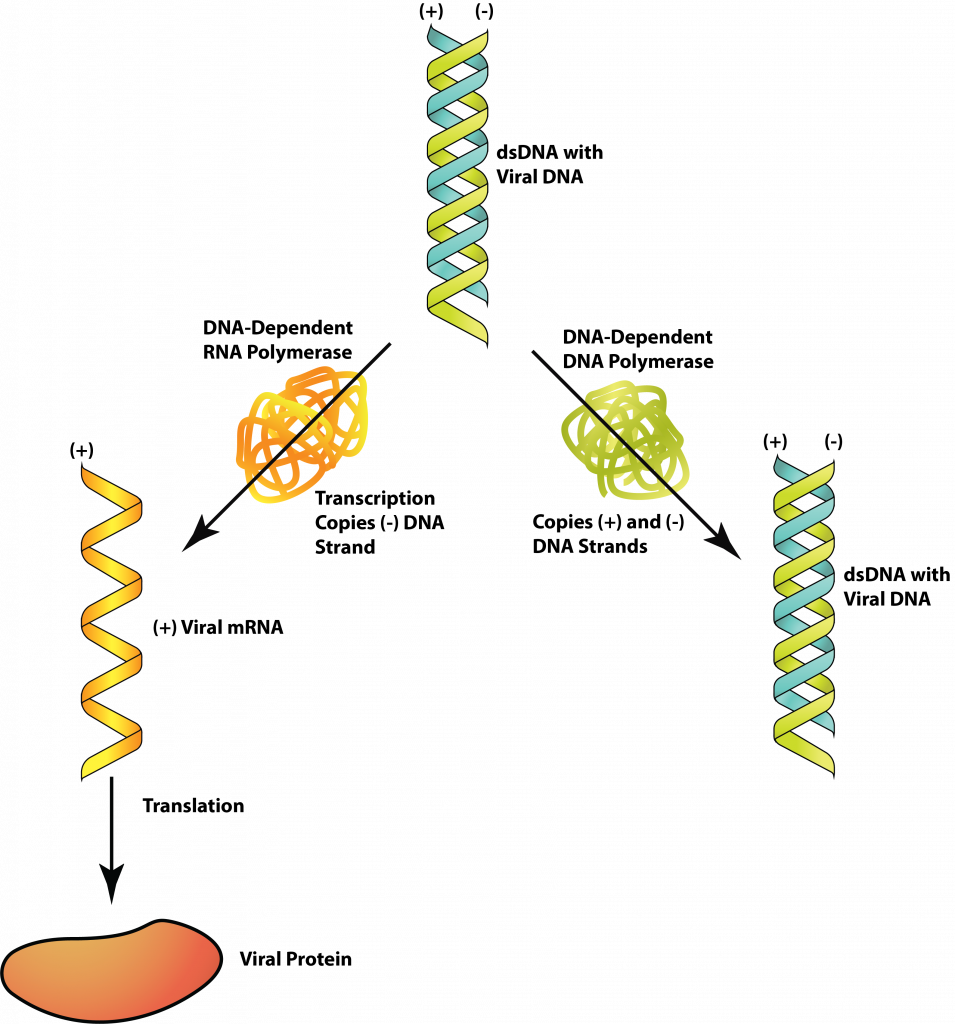
ADNds.
El virus a menudo emplea estrategias para el control de la expresión génica, para asegurar que los productos virales particulares se elaboran en momentos específicos de la replicación del virus. En el caso de T4, la ARN polimerasa huésped se une al ADN viral y comienza a transcribir genes tempranos inmediatamente después de inyectar el ADN en la célula. Una de las proteínas virales tempranas modifica la ARN polimerasa huésped para que ya no reconozca a los promotores del hospedador en absoluto, además de pasar a transcribir genes para proteínas virales de etapa media. Una modificación adicional (catalizada por proteínas virales de etapa media) modificó aún más la ARN polimerasa para reconocer genes virales que codifican proteínas de etapa tardía. Esto asegura una producción ordenada de proteínas virales.
La replicación de varios virus dsDNA da como resultado la producción de concatémeros, donde varios genomas virales se unen entre sí debido a regiones cortas monocatenarias con repeticiones terminales. A medida que el genoma se empaqueta en la cápside, una endonucleasa viral corta el concatémero a una longitud apropiada.
Existen varios virus animales con genomas de ADNds, como los virus de la viruela y los adenovirus. Los herpesvirus tienen varias características notables, como el vínculo de varios miembros con el cáncer y la capacidad de los virus para permanecer en forma latente dentro de su huésped. Una infección productiva da como resultado una población viral explosiva, muerte celular y desarrollo de signos de enfermedad, durante los cuales se infectan las neuronas. Una infección latente se desarrolla en las neuronas, permitiendo que el virus permanezca sin ser detectado en el huésped. Si se reactiva el genoma viral, se produce una infección productiva, lo que lleva a la replicación viral y a los signos de la enfermedad nuevamente.
Clase II: ADNss
El flujo de información para los virus ssDNA, como los parvovirus, seguirá la vía convencional, hasta cierto punto: ADN → ARNm → proteína. Pero el genoma viral puede tener la misma secuencia de bases que el ARNm (ADN de cadena positiva) o ser complementario al ARNm (ADN de cadena menor). En el primer caso, primero se debe fabricar una cadena de ADN que sea complementaria al genoma viral, formando una forma replicativa bicatenaria (RF). Esto se puede usar tanto para fabricar proteínas virales como como molde para copias del genoma viral. Para los virus de ADN de cadena menor, el genoma puede usarse directamente para producir ARNm pero aún será necesario hacer una copia complementaria, que sirva como molde para las copias del genoma viral.
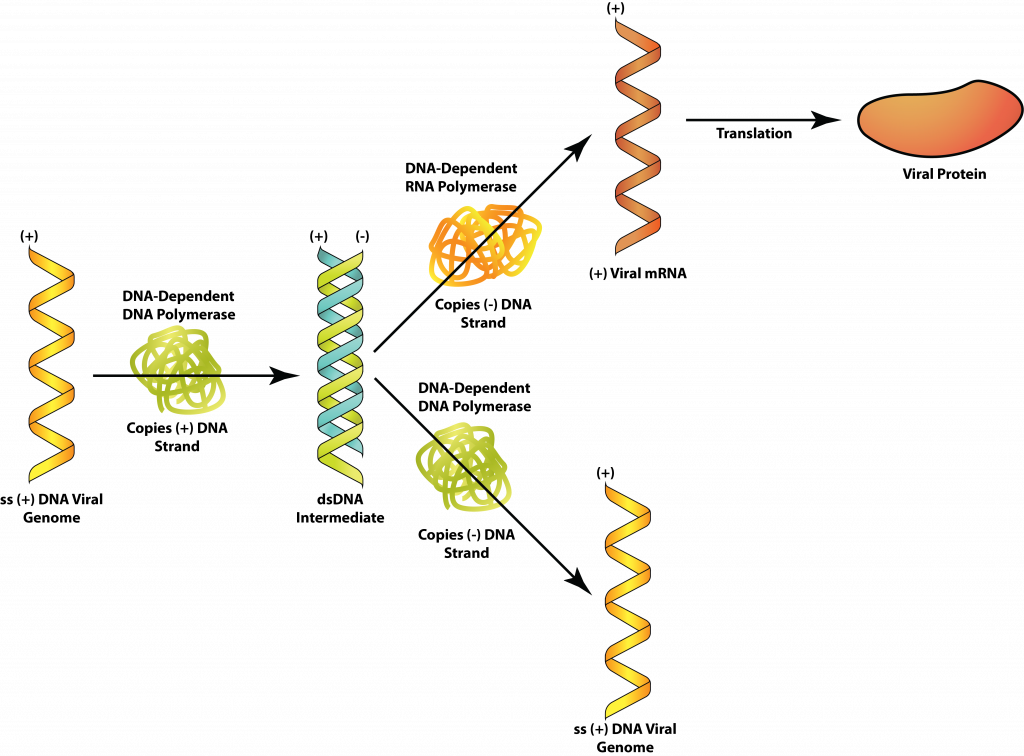
ADNss.
La forma replicativa se puede usar para la replicación de círculo rodante, donde se corta una cadena y se usan enzimas de replicación para extender el extremo 3' libre. A medida que se sintetiza una cadena complementaria alrededor del ADN circular, el extremo 5' se despega, lo que lleva a una hebra desplazada que continúa creciendo en longitud.
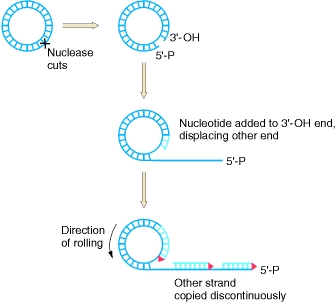
Replicación de Círculo Rodante.
Clase VII: Virus de ADN que usan transcriptasa inversa
Los hepadnavirus contienen un genoma de ADN que es parcialmente bicatenario, pero contiene una región monocatenaria. Después de entrar en el núcleo de la célula, las enzimas de la célula hospedadora se utilizan para llenar el hueco con bases complementarias para formar un bucle cerrado de ADNds. La transcripción génica produce un ARN de cadena positiva conocido como pregenoma, así como la enzima viral transcriptasa inversa, una ADN polimerasa dependiente de ARN. El pregenoma se utiliza como molde para que la transcriptasa inversa produzca genomas de ADN de cadena menor, con un pequeño trozo de pregenoma utilizado como cebador para producir la región bicatenaria de los genomas.
Virus de ARN
Clase III: ARNds
Los virus de ARN bicatenario infectan bacterias, hongos, plantas y animales, como el rotavirus que causa enfermedades diarreicas en humanos. Pero las células no utilizan ARNds en ninguno de sus procesos y tienen sistemas para destruir cualquier ARNds que se encuentre en la célula. Así, el genoma viral, en su forma de ARNds, debe estar oculto o protegido de las enzimas celulares. Las células también carecen de RNA-polimerasas dependientes de ARN, necesarias para la replicación del genoma viral por lo que el virus debe proporcionar esta enzima por sí misma. La ARN polimerasa dependiente de ARN viral actúa tanto como transcriptasa para transcribir ARNm, como replicasa para replicar el genoma del ARN.
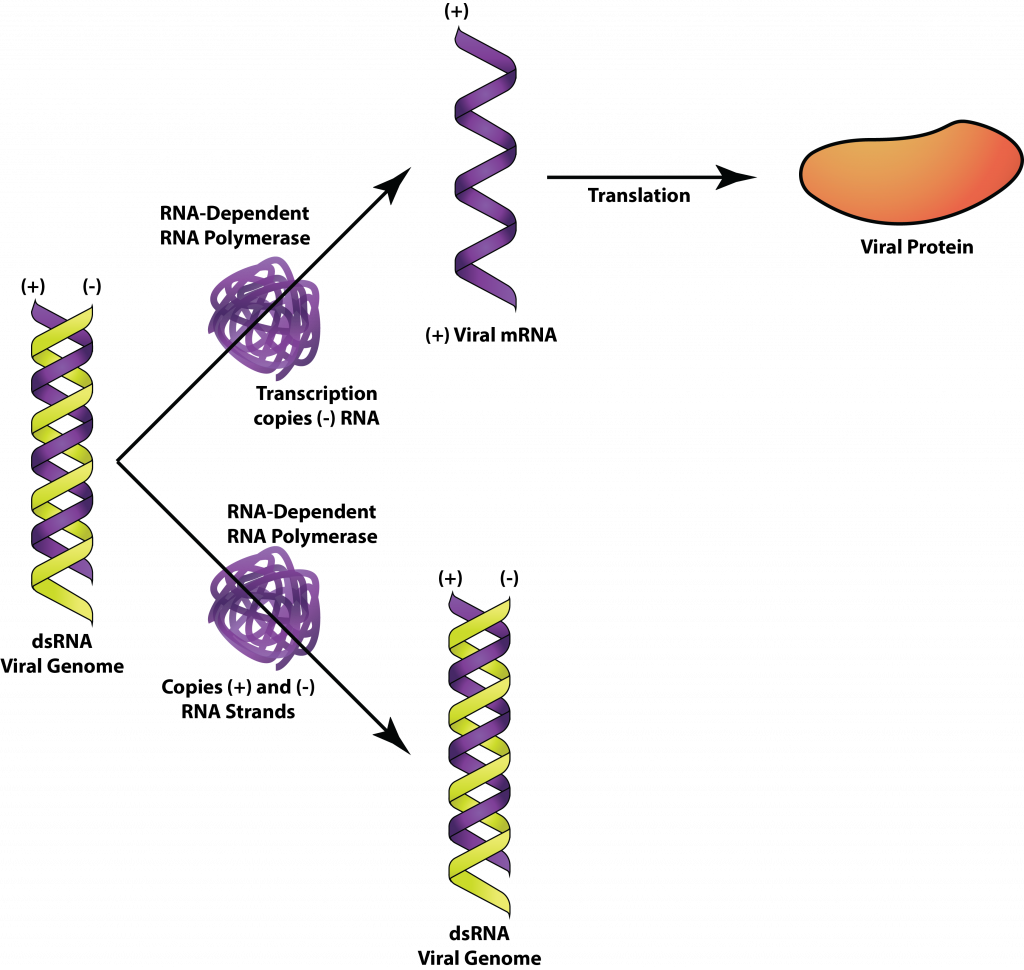
ARNds.
Para el rotavirus, la nucleocápsida viral permanece intacta en el citoplasma con eventos de replicación ocurridos en su interior, permitiendo que el ARNds permanezca protegido. El ARN mensajero se transcribe a partir de la cadena menos del genoma del ARN y luego se traduce por el ribosoma huésped en el citoplasma. Las proteínas virales se agregan para formar nuevas nucleocápsidas alrededor de ARN replicasa y ARN de cadena positiva. El ARN de cadena menor es sintetizado luego por la ARN replicasa dentro de la nucleocápsida, asegurando una vez más la protección del genoma del ARNds.
Clase IV: +ARNss
Los virus con ARN de cadena positiva, como el poliovirus, pueden usar su genoma directamente como ARNm con la traducción por parte del ribosoma huésped que ocurre tan pronto como el genoma viral no segmentado gana entrada en la célula. Uno de los genes virales expresados produce una ARN polimerasa dependiente de ARN (o ARN replicasa), que crea ARN de cadena menor a partir del genoma de cadena positiva. El ARN de cadena menor se puede usar como molde para más ARN de cadena positiva, que se puede usar como ARNm o como genomas para los virus recién formados.
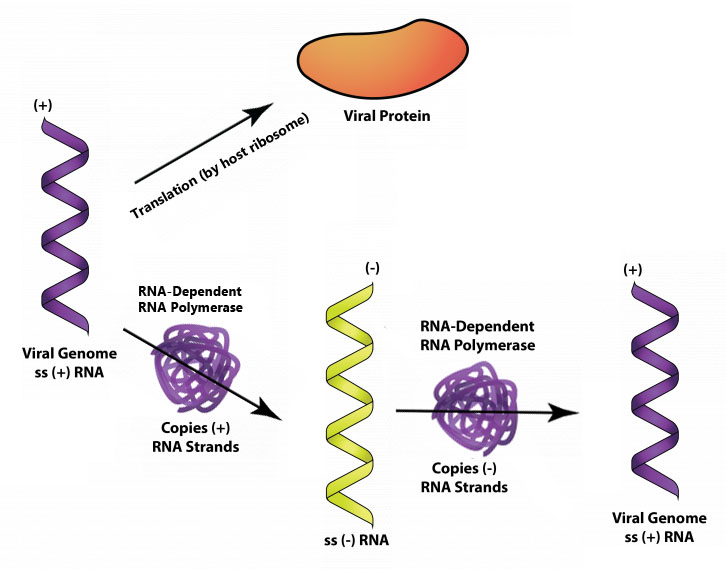
+ARN monocatenario.
La traducción del genoma del poliovirus produce una poliproteína, una proteína grande con actividad proteasa que se escinde en tres proteínas más pequeñas. La actividad de escisión adicional finalmente produce todas las proteínas necesarias para la formación de la cápside, así como una ARN-polimerasa dependiente de ARN.
La formación de una poliproteína que se corta en varias proteínas más pequeñas ilustra una posible estrategia para un problema que enfrentan muchos virus +ARNmc: ¿cómo generar múltiples proteínas a partir de un genoma de +ARNmc no segmentado? Otras posibilidades incluyen:
- ARNm subgenómico: durante la traducción, se pueden omitir porciones del ARN viral, lo que resulta en proteínas diferentes a las que se elabora a partir del ARN viral en su totalidad.
- cambio de marco ribosómico: el ribosoma “lee” el ARNm en grupos de tres nucleótidos o codón, que se traducen en un aminoácido. Si el ribosoma comienza con el nucleótido #1, ese es un marco de lectura abierto (ORF), resultando en un conjunto de aminoácidos. Si el ribosoma se moviera hacia adelante donde el nucleótido 2 es el nucleótido de partida ese sería ORF #2, resultando en un conjunto completamente diferente de aminoácidos. Si el ribosoma volviera a avanzar donde el nucleótido 3 es el nucleótido de partida ese sería el ORF #3, resultando en un conjunto completamente diferente de aminoácidos. Algunos virus tienen genes virales que se superponen deliberadamente dentro de diferentes ORF, lo que lleva a la producción de diferentes proteínas a partir de un solo ARNm.
- mecanismo de lectura: un genoma viral puede tener codones de parada incrustados a lo largo de la secuencia. Cuando el ribosoma llega a un codón de parada puede detenerse, terminar la secuencia de aminoácidos, o puede ignorar el codón de parada, continuando para formar una cadena más larga de aminoácidos. Para los virus con el mecanismo de lectura, adquieren una variedad de proteínas al tener codones de parada que periódicamente se ignoran. A veces esta función se combina con el cambio de marco ribosómico para producir una variedad aún mayor de proteínas virales.
Clase V: -ARNss
Los virus de ARN de cadena menor incluyen muchos miembros notables para los humanos, como el virus de la influenza, el virus de la rabia y el virus del Ébola. Dado que el genoma de los virus de ARN de cadena menor no puede usarse directamente como ARNm, el virus debe portar una ARN-polimerasa dependiente de ARN dentro de su cápside. Al entrar en la célula hospedadora, los ARN de cadena positiva generados por la polimerasa se utilizan como ARNm para la producción de proteínas. Cuando se necesitan genomas virales, los ARN de cadena positiva se usan como moldes para hacer ARN de cadena menor.
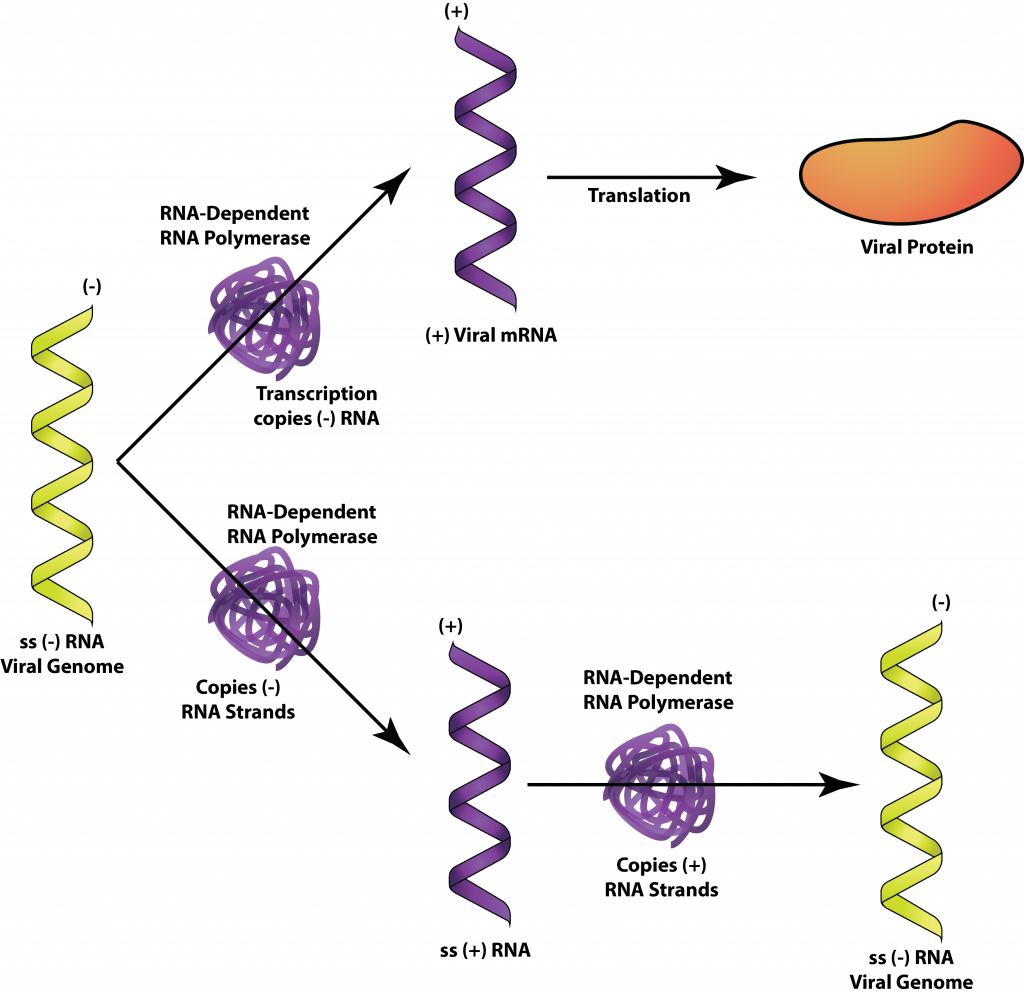
-ARN monocatenario.
Clase VI: +ARNss, retrovirus
A pesar de que el genoma retroviral está compuesto por +ARNss, no se utiliza como ARNm. En cambio, el virus utiliza su transcriptasa inversa para sintetizar una pieza de ADNss complementaria al genoma viral. La transcriptasa inversa también posee actividad ribonucleasa, que se utiliza para degradar la cadena de ARN del híbrido ARN-ADN. Por último, la transcriptasa inversa se utiliza como ADN polimerasa para hacer una copia complementaria al ssADN, produciendo una molécula de ADNds. Esto permite que el virus inserte su genoma, en forma de ADNds, en el cromosoma huésped, formando un provirus. A diferencia de un profago, un provirus puede permanecer latente indefinidamente o provocar la expresión de genes virales, lo que lleva a la producción de nuevos virus. La escisión del provirus no ocurre para la expresión génica.
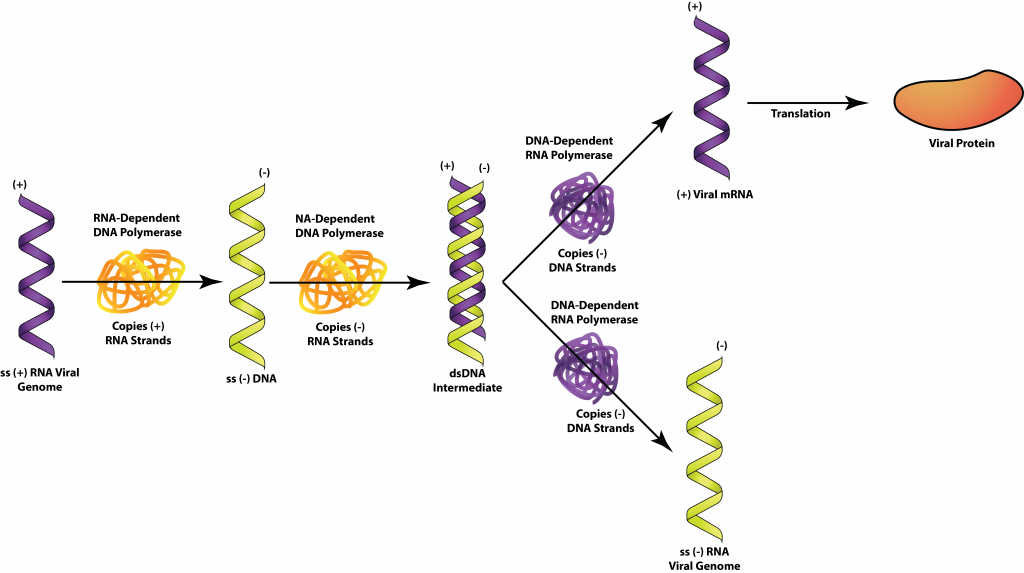
+ARNss, retrovirus.
Otros Agentes Infecciosos
Viroides
Los viroides son pequeñas moléculas circulares de ARN monocatenario que carecen de proteínas. Estas moléculas infecciosas están asociadas con una serie de enfermedades de las plantas. Dado que el ARN monocatenario es altamente susceptible a la degradación enzimática, el ARN viroide tiene un amplio apareamiento de bases complementarias, lo que hace que el viroide adquiera una configuración de horquilla que es resistente a las enzimas. Para la replicación, los viroides se basan en una ARN polimerasa vegetal con actividad de ARN replicasa.
Priones
Los priones son agentes infecciosos que carecen completamente de ácido nucleico de cualquier tipo, estando hechos enteramente de proteína. Se asocian a una variedad de enfermedades, principalmente en animales, aunque se ha encontrado un prión que infecta la levadura (!). Las enfermedades incluyen la encefalopatía espongiforme bovina (EEB o “enfermedad de las vacas locas”), la enfermedad de Creutzfeld-Jakob en humanos y la tembladera en ovejas.
La proteína priónica se encuentra en las neuronas de animales sanos (PrPc o Prion Protein Cellular), con una estructura secundaria particular. La forma patógena (PrPSc o Prion Protein Scrapie) tiene una estructura secundaria diferente y es capaz de convertir la PrPc en la forma patógena. La acumulación de la forma patógena provoca la destrucción del cerebro y el tejido nervioso, lo que lleva a síntomas de la enfermedad como pérdida de memoria, falta de coordinación y, finalmente, la muerte.
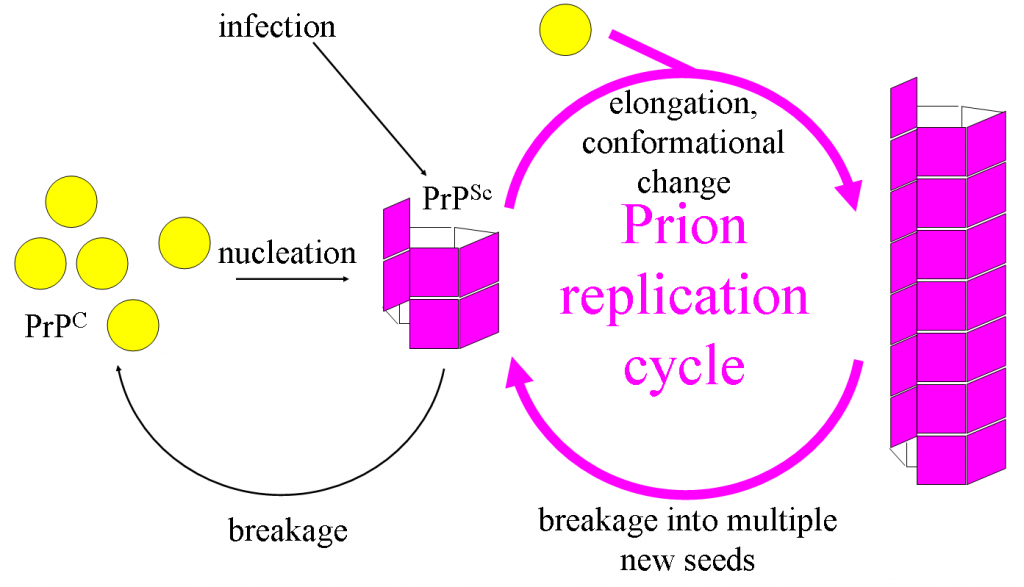
Priones. Joannamasel en Wikipedia en inglés [CC BY-SA 3.0], vía Wikimedia Commons
Palabras clave
Esquema Baltimore, Clase I, Clase II, Clase III, Clase IV, Clase V, Clase VI, Clase VII, ARN polimerasa dependiente de ADN, ADN-polimerasa dependiente de ADN, concatémero, infección productiva, infección latente, ADN/+ADN de cadena positiva, ADN/ADN de cadena menor, ADNbc, ADNss, forma replicativa (RF), replicación en círculo rodante, pregenoma, transcriptasa inversa, ADN-polimerasa dependiente de ARN, ARNbc, ARN polimerasa dependiente de ARN, transcriptasa, replicasa, ARN de cadena más/+ARN monocatenario, ARN/ARNss de cadena menor, poliproteína, ARNm subgenómico, cambio de marco ribosómico, marco de lectura abierto (ORF), mecanismo de lectura, codón de parada, retrovirus, ribonucleasa, provirus, viroides, prión, PrPC/Prión Proteína Celular, PrPSC/Prión Proteína Scrapie.
Preguntas/Objetivos Esenciales
- ¿Cuál es el sistema de clasificación de Baltimore? ¿Qué características virales utiliza? ¿Cómo produce cada grupo viral proteínas y replica su genoma? ¿De dónde provienen los componentes necesarios? (virus o célula hospedadora) ¿Qué modificaciones son necesarias, para los virus con un genoma diferente al de la célula hospedadora?
- ¿Qué estrategia utilizan los virus dsDNA para el control de la expresión génica? ¿Qué son los concatémeros? ¿Qué son las infecciones productivas y latentes?
- ¿Qué es una forma replicativa? ¿Qué es la replicación de círculo rodante? ¿Cuál es la ventaja de estos mecanismos virales?
- ¿Qué es un pregenoma? ¿Qué es la transcriptasa inversa? ¿Qué papel juega para los virus Clase VII?
- ¿A qué problemas se enfrentan los virus de ARNds? ¿Cómo superan estos problemas? ¿Qué es una transcriptasa? ¿Qué es una replicasa?
- ¿Cómo utilizan el genoma los virus Clase IV +ARNss? ¿Cuáles son las estrategias utilizadas por estos virus para generar múltiples proteínas a partir de un genoma no segmentado?
- ¿Qué pasos son necesarios para los virus —ARNss?
- ¿En qué se diferencian los retrovirus, como virus +ARNss, de los virus de Clase IV? ¿Qué es una ribonucleasa? ¿Qué es un provirus?
- ¿Qué es un viroide? ¿Qué es un prión? ¿Cómo causan enfermedades estos agentes? ¿Cómo se replican?
Preguntas Exploratorias (OPCIONAL)
- ¿Por qué los científicos fueron inicialmente tan resistentes a la idea de que los priones carecían de algún tipo de ácido nucleico?

