11.3: Transcripción de ARN
- Page ID
- 54604
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Objetivos de aprendizaje
- Explicar cómo se sintetiza el ARN usando ADN como plantilla
- Distinguir entre transcripción en procariotas y eucariotas
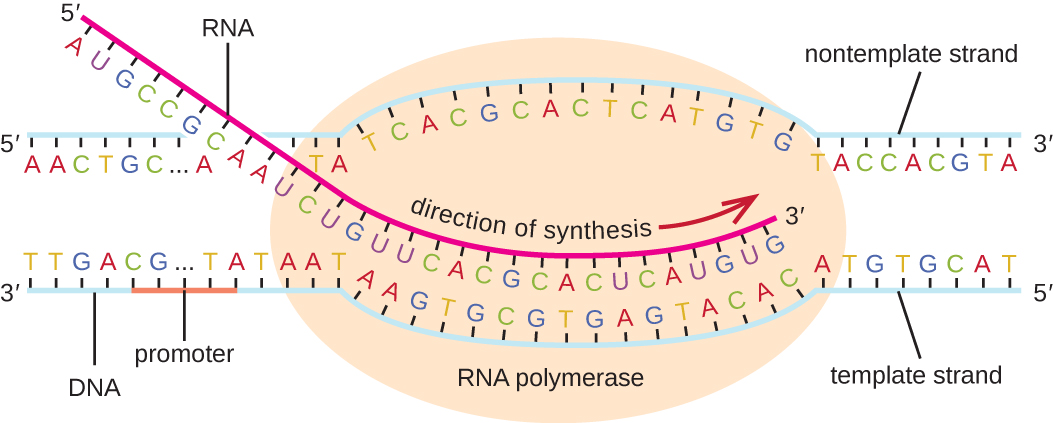
Durante el proceso de transcripción, la información codificada dentro de la secuencia de ADN de uno o más genes se transcribe en una cadena de ARN, también llamada transcripción de ARN. La molécula de ARN monocatenario resultante, compuesta por ribonucleótidos que contienen las bases adenina (A), citosina (C), guanina (G) y uracilo (U), actúa como una copia molecular móvil de la secuencia de ADN original. La transcripción en procariotas y en eucariotas requiere que la doble hélice de ADN se desenrolle parcialmente en la región de la síntesis de ARN. La región desenrollada se llama burbuja de transcripción. La transcripción de un gen particular siempre procede de una de las dos cadenas de ADN que actúa como molde, la llamada cadena antisentido. El producto de ARN es complementario a la cadena molde de ADN y es casi idéntico a la cadena de ADN no molde, o la cadena sentido. La única diferencia es que en el ARN, todos los nucleótidos T son reemplazados por nucleótidos U; durante la síntesis de ARN, U se incorpora cuando hay una A en la cadena antisentido complementaria.
Transcripción en Bacterias
Las bacterias utilizan la misma ARN polimerasa para transcribir todos sus genes. Al igual que la ADN polimerasa, la ARN polimerasa agrega nucleótidos uno por uno al grupo 3'-OH de la cadena de nucleótidos en crecimiento. Una diferencia crítica en la actividad entre la ADN polimerasa y la ARN polimerasa es el requisito de un 3'-OH sobre el que agregar nucleótidos: la ADN polimerasa requiere tal grupo 3'-OH, por lo que necesita un cebador, mientras que la ARN polimerasa no. Durante la transcripción, se agrega un ribonucleótido complementario a la cadena molde de ADN a la cadena de ARN en crecimiento y se forma un enlace fosfodiéster covalente por síntesis por deshidratación entre el nuevo nucleótido y el último agregado. En E. coli, la ARN polimerasa comprende seis subunidades polipeptídicas, cinco de las cuales componen la enzima central de la polimerasa responsable de agregar nucleótidos de ARN a una cadena en crecimiento. La sexta subunidad se conoce como sigma (σ). El factor σ permite que la ARN polimerasa se una a un promotor específico, permitiendo así la transcripción de varios genes. Existen diversos factores σ que permiten la transcripción de diversos genes.
Iniciación
El inicio de la transcripción comienza en un promotor, una secuencia de ADN a la que se une la maquinaria de transcripción e inicia la transcripción. El par de nucleótidos en la doble hélice de ADN que corresponde al sitio desde el que se transcribe el primer nucleótido de ARN 5' es el sitio de inicio. “Los nucleótidos que preceden al sitio de iniciación se designan “" aguas arriba "”, mientras que los nucleótidos que siguen al sitio de iniciación se denominan nucleótidos “" En la mayoría de los casos, los promotores se localizan justo aguas arriba de los genes que regulan. Aunque las secuencias promotoras varían entre los genomas bacterianos, se conservan algunos elementos. En las posiciones —10 y —35 dentro del ADN antes del sitio de inicio (designadas +1), hay dos secuencias consenso promotoras, o regiones que son similares en todos los promotores y en diversas especies bacterianas. La secuencia consenso —10, llamada caja TATA, es TATAAT. La secuencia —35 es reconocida y unida por σ.
Alargamiento
El alargamiento en la fase de transcripción comienza cuando la subunidad σ se disocia de la polimerasa, permitiendo que la enzima central sintetice ARN complementario al molde de ADN en una dirección 5' a 3' a una velocidad de aproximadamente 40 nucleótidos por segundo. A medida que avanza la elongación, el ADN se desenrolla continuamente por delante de la enzima central y se rebobina detrás de ella (Figura\(\PageIndex{1}\)).
Terminación
Una vez que se transcribe un gen, la polimerasa bacteriana debe disociarse del molde de ADN y liberar el ARN recién hecho. Esto se conoce como terminación de la transcripción. El molde de ADN incluye secuencias de nucleótidos repetidas que actúan como señales de terminación, haciendo que la ARN polimerasa se detenga y se libere del molde de ADN, liberando el transcrito de ARN.
Ejercicio\(\PageIndex{1}\)
- ¿Dónde se une el factor σ de la ARN polimerasa al ADN para iniciar la transcripción?
- ¿Qué ocurre para iniciar la actividad de polimerización de la ARN polimerasa?
- ¿De dónde viene la señal para terminar la transcripción?
Transcripción en Eucariotas
Los procariotas y eucariotas realizan fundamentalmente el mismo proceso de transcripción, con algunas diferencias significativas (ver Tabla\(\PageIndex{1}\)). Los eucariotas utilizan tres polimerasas diferentes, las ARN polimerasas I, II y III, todas estructuralmente distintas de la ARN polimerasa bacteriana. Cada uno transcribe un subconjunto diferente de genes. Curiosamente, las arqueas contienen una sola ARN polimerasa que está más estrechamente relacionada con la ARN polimerasa eucariota II que con su contraparte bacteriana. Los ARNm eucariotas también suelen ser monocistrónicos, lo que significa que cada uno codifica solo un único polipéptido, mientras que los ARNm procariotas de bacterias y arqueas son comúnmente policistrónicos, lo que significa que codifican múltiples polipéptidos.
La diferencia más importante entre procariotas y eucariotas es el núcleo unido a la membrana de este último, lo que influye en la facilidad de uso de las moléculas de ARN para la síntesis de proteínas. Con los genes unidos en un núcleo, la célula eucariota debe transportar moléculas de ARN que codifican proteínas al citoplasma para ser traducidas. Los transcritos primarios que codifican proteínas, las moléculas de ARN sintetizadas directamente por la ARN polimerasa, deben someterse a varias etapas de procesamiento para proteger a estas moléculas de ARN de la degradación durante el tiempo que se transfieren del núcleo al citoplasma y se traducen en una proteína. Por ejemplo, los ARNm eucariotas pueden durar varias horas, mientras que el ARNm procariota típico no dura más de 5 segundos.
El transcrito primario (también llamado pre-ARNm) se recubre primero con proteínas estabilizadoras de ARN para protegerlo de la degradación mientras se procesa y exporta fuera del núcleo. El primer tipo de procesamiento comienza mientras el transcrito primario aún se está sintetizando; se agrega un nucleótido especial de 7-metilguanosina, llamado capuchón 5', al extremo 5' del transcrito en crecimiento. Además de prevenir la degradación, los factores involucrados en la posterior síntesis de proteínas reconocen el cap, lo que ayuda a iniciar la traducción por los ribosomas. Una vez que se completa la elongación, otra enzima procesadora agrega una cadena de aproximadamente 200 nucleótidos adenina al extremo 3', llamada cola poli-A. Esta modificación protege además al pre-ARNm de la degradación y señala a factores celulares que el transcrito necesita ser exportado al citoplasma.
Los genes eucariotas que codifican polipéptidos están compuestos por secuencias codificantes llamadas exones (ex -on significa que son ex presionados) y secuencias intervinientes llamadas intrones (int -ron denota su papel int ervening). Las secuencias de ARN transcritas correspondientes a intrones no codifican regiones del polipéptido funcional y se eliminan del pre-ARNm durante el procesamiento. Es esencial que todas las secuencias de ARN codificadas por intrones se eliminen completa y precisamente de un pre-ARNm antes de la síntesis de proteínas para que las secuencias de ARN codificadas por exón se unan adecuadamente entre sí para codificar un polipéptido funcional. Si el proceso se equivoca incluso por un solo nucleótido, las secuencias de los exones reunidos se desplazarían, y el polipéptido resultante sería no funcional. El proceso de eliminación de secuencias de ARN codificadas por intrones y reconexión de las codificadas por exones se denomina corte y empalme de ARN y se ve facilitado por la acción de un spliceosoma que contiene pequeñas proteínas de ribonucleo nuclear (SNRNP). Las secuencias de ARN codificadas por intrones se eliminan del pre-ARNm mientras aún se encuentra en el núcleo. Aunque no se traducen, los intrones parecen tener diversas funciones, incluyendo la regulación génica y el transporte de ARNm. Al término de estas modificaciones, el transcrito maduro, el ARNm que codifica un polipéptido, es transportado fuera del núcleo, destinado al citoplasma para su traducción. Los intrones se pueden cortar y empalmar de manera diferente, dando como resultado que varios exones se incluyan o excluyan del producto final de ARNm. Este proceso se conoce como empalme alternativo. La ventaja del corte y empalme alternativo es que se pueden generar diferentes tipos de transcritos de ARNm, todos derivados de la misma secuencia de ADN. En los últimos años, se ha demostrado que algunas arqueas también tienen la capacidad de empalmar su pre-ARNm.
| Propiedad | Bacterias | Eucariotas |
|---|---|---|
| Número de polipéptidos codificados por ARNm | Monocistrónico o policistrónico | Exclusivamente monocistrónico |
| Alargamiento del filamento | núcleo + σ = holoenzima | ARN polimerasas I, II o III |
| Adición de tapa de 5' | No | Sí |
| Adición de cola poli-A 3' | No | Sí |
| Empalme de pre-ARNm | No | Sí |
Visualiza cómo ocurre el empalme de ARNm viendo el proceso en acción en este video. Vea cómo se eliminan los intrones durante el corte y empalme de ARN aquí.
Ejercicio\(\PageIndex{2}\)
- En las células eucariotas, ¿cómo se modifica la transcripción de ARN de un gen para una proteína después de ser transcrita?
- ¿Los exones o intrones contienen información para las secuencias proteicas?
Enfoque Clínico: Parte 2
En el servicio de urgencias, una enfermera le dijo a Mark que había tomado una buena decisión de acudir al hospital porque sus síntomas indicaban una infección que se había salido de control. Los síntomas de Mark habían progresado, con el área de la piel afectada y aumentando la cantidad de hinchazón. Dentro de la zona afectada había comenzado una erupción, se habían formado ampollas y se habían formado pequeñas bolsas de gas debajo de la capa más externa de la piel, y parte de la piel se estaba volviendo gris. Con base en el olor pútrido del pus que drena de una de las ampollas, la rápida progresión de la infección y la apariencia visual de la piel afectada, el médico inició de inmediato el tratamiento para la fascitis necrotizante. El médico de Mark ordenó un cultivo del líquido que drenaba de la ampolla y también ordenó análisis de sangre, incluido un recuento de glóbulos blancos.
Mark ingresó en la unidad de cuidados intensivos y comenzó la administración intravenosa de un antibiótico de amplio espectro para tratar de minimizar la propagación de la infección. A pesar de la terapia con antibióticos, la condición de Mark se deterioró rápidamente. Mark se volvió confuso y mareado. A las pocas horas de su ingreso hospitalario, su presión arterial bajó significativamente y su respiración se volvió más superficial y rápida. Adicionalmente, las ampollas aumentaron, con las ampollas intensificándose en color a negro púrpura, y la herida misma parecía estar progresando rápidamente en la pierna de Mark.
Ejercicio\(\PageIndex{3}\)
- ¿Cuáles son los posibles agentes causantes de la fascitis necrotizante de Mark?
- ¿Cuáles son algunas posibles explicaciones de por qué el tratamiento antibiótico no parece estar funcionando?
Conceptos clave y resumen
- Durante la transcripción, la información codificada en el ADN se utiliza para hacer ARN.
- La ARN polimerasa sintetiza ARN, usando la cadena antisentido del ADN como molde agregando nucleótidos de ARN complementarios al extremo 3' de la cadena en crecimiento.
- La ARN polimerasa se une al ADN en una secuencia llamada promotor durante el inicio de la transcripción.
- Los genes que codifican proteínas de funciones relacionadas se transcriben frecuentemente bajo el control de un solo promotor en procariotas, dando como resultado la formación de una molécula de ARNm policistrónica que codifica múltiples polipéptidos.
- A diferencia de la ADN polimerasa, la ARN polimerasa no requiere de un grupo 3'-OH para agregar nucleótidos, por lo que no se necesita un cebador durante el inicio.
- La terminación de la transcripción en bacterias ocurre cuando la ARN polimerasa se encuentra con secuencias de ADN específicas que conducen al bloqueo de la polimerasa. Esto da como resultado la liberación de ARN polimerasa de la cadena molde de ADN, liberando el transcrito de ARN.
- Los eucariotas tienen tres ARN polimerasas diferentes. Los eucariotas también tienen ARNm monocistrónico, cada uno codificando solo un único polipéptido.
- Los transcritos primarios eucariotas se procesan de varias maneras, incluyendo la adición de una caperuza 5' y una cola 3'-poli-A, así como el corte y empalme, para generar una molécula de ARNm madura que puede ser transportada fuera del núcleo y que está protegida de la degradación.


