11.4: Síntesis de Proteínas (Traducción)
- Page ID
- 54587
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Objetivos de aprendizaje
- Describir el código genético y explicar por qué se considera casi universal
- Explicar el proceso de traducción y las funciones de la maquinaria molecular de la traducción
- Comparar traducción en eucariotas y procariotas
La síntesis de proteínas consume más energía de una célula que cualquier otro proceso metabólico. A su vez, las proteínas representan más masa que cualquier otra macromolécula de organismos vivos. Realizan prácticamente todas las funciones de una célula, sirviendo como elementos funcionales (e.g., enzimas) y estructurales. El proceso de traducción, o síntesis de proteínas, la segunda parte de la expresión génica, implica la decodificación por un ribosoma de un mensaje de ARNm en un producto polipeptídico.
El Código Genético
La traducción de la plantilla de ARNm convierte la información genética basada en nucleótidos en el “lenguaje” de los aminoácidos para crear un producto proteico. Una secuencia proteica consiste en 20 aminoácidos que ocurren comúnmente. Cada aminoácido se define dentro del ARNm por un triplete de nucleótidos llamado codón. La relación entre un codón de ARNm y su aminoácido correspondiente se denomina código genético.
El código de tres nucleótidos significa que hay un total de 64 combinaciones posibles (4 3, con cuatro nucleótidos diferentes posibles en cada una de las tres posiciones diferentes dentro del codón). Este número es mayor que el número de aminoácidos y un aminoácido dado está codificado por más de un codón (Figura\(\PageIndex{1}\)). Esta redundancia en el código genético se llama degeneración. Típicamente, mientras que las dos primeras posiciones en un codón son importantes para determinar qué aminoácido se incorporará a un polipéptido en crecimiento, la tercera posición, llamada posición de oscilación, es menos crítica. En algunos casos, si se cambia el nucleótido en la tercera posición, todavía se incorpora el mismo aminoácido.
Mientras que 61 de los 64 posibles tripletes codifican aminoácidos, tres de los 64 codones no codifican un aminoácido; terminan la síntesis de proteínas, liberando al polipéptido de la maquinaria de traducción. Estos se denominan codones de parada o codones sin sentido s. Otro codón, AUG, también tiene una función especial. Además de especificar el aminoácido metionina, normalmente también sirve como codón de inicio para iniciar la traducción. El marco de lectura, la forma en que los nucleótidos en el ARNm se agrupan en codones, para la traducción se establece por el codón de inicio AUG cerca del extremo 5' del ARNm. Cada conjunto de tres nucleótidos después de este codón de inicio es un codón en el mensaje de ARNm.
El código genético es casi universal. Con algunas excepciones, prácticamente todas las especies utilizan el mismo código genético para la síntesis de proteínas, lo que es una poderosa evidencia de que toda la vida existente en la tierra comparte un origen común. Sin embargo, se han observado aminoácidos inusuales como selenocisteína y pirrolisina en arqueas y bacterias. En el caso de la selenocisteína, el codón utilizado es UGA (normalmente un codón de parada). Sin embargo, la UGA puede codificar para selenocisteína usando una estructura tallo-bucle (conocida como la secuencia de inserción de selenocisteína, o elemento SECIS), que se encuentra en la región 3' no traducida del ARNm. La pirrolisina utiliza un codón de terminación diferente, UAG. La incorporación de pirrolisina requiere el gen pyls y un ARN de transferencia único (ARNt) con un anticodón CUA.
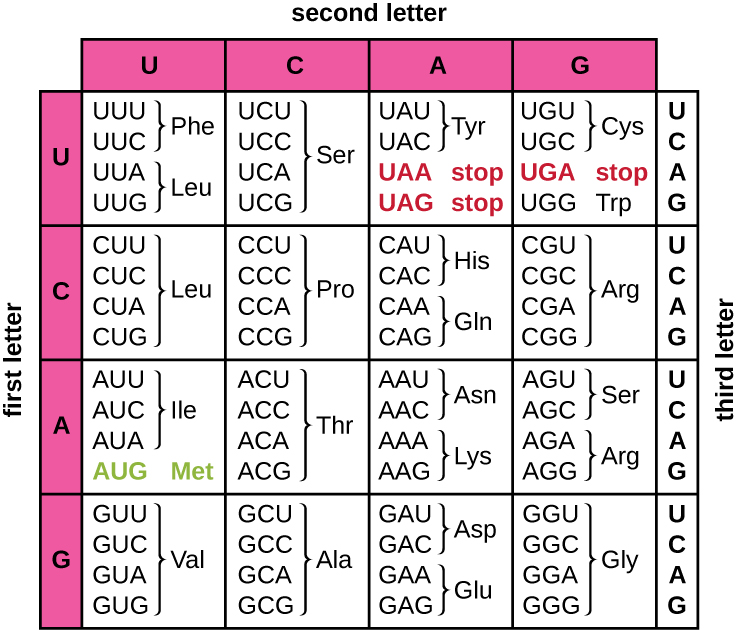
Ejercicio\(\PageIndex{1}\)
- ¿Cuántas bases hay en cada codón?
- ¿Para qué aminoácido está codificado por el codón AAU?
- ¿Qué sucede cuando se alcanza un codón de parada?
La maquinaria de síntesis de proteínas
Además del molde de ARNm, muchas moléculas y macromoléculas contribuyen al proceso de traducción. La composición de cada componente varía según los taxones; por ejemplo, los ribosomas pueden consistir en diferentes números de ARN ribosómicos (ARNr) y polipéptidos dependiendo del organismo. Sin embargo, las estructuras y funciones generales de la maquinaria de síntesis de proteínas son comparables de bacterias a células humanas. La traducción requiere la entrada de un molde de ARNm, ribosomas, ARNt y varios factores enzimáticos.
Ribosomas
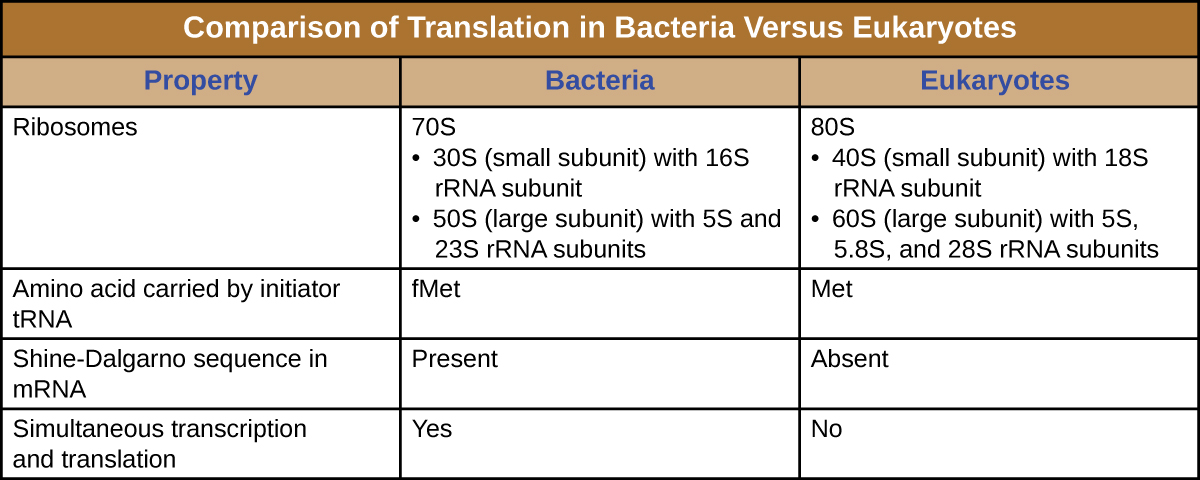
Un ribosoma es una macromolécula compleja compuesta por ARNs catalíticos (llamados ribozimas) y ARNs estructurales, así como muchos polipéptidos distintos. Los ARNr maduros constituyen aproximadamente el 50% de cada ribosoma. Los procariotas tienen ribosomas 70S, mientras que los eucariotas tienen ribosomas 80S en el citoplasma y retículo endoplásmico rugoso, y ribosomas 70S en mitocondrias y cloroplastos. Los ribosomas se disocian en subunidades grandes y pequeñas cuando no están sintetizando proteínas y se reasocian durante el inicio de la traducción. En E. coli, la subunidad pequeña se describe como 30S (que contiene la subunidad 16S rRNA), y la subunidad grande es 50S (que contiene las subunidades 5S y 23S rRNA), para un total de 70S (las unidades de Svedberg no son aditivas). Los ribosomas eucariotas tienen una pequeña subunidad 40S (que contiene la subunidad de ARNr 18S) y una subunidad 60S grande (que contiene las subunidades 5S, 5.8S y 28S rRNA), para un total de 80S. La subunidad pequeña es responsable de unirse al molde de ARNm, mientras que la subunidad grande se une a los ARNt (discutidos en la siguiente subsección).
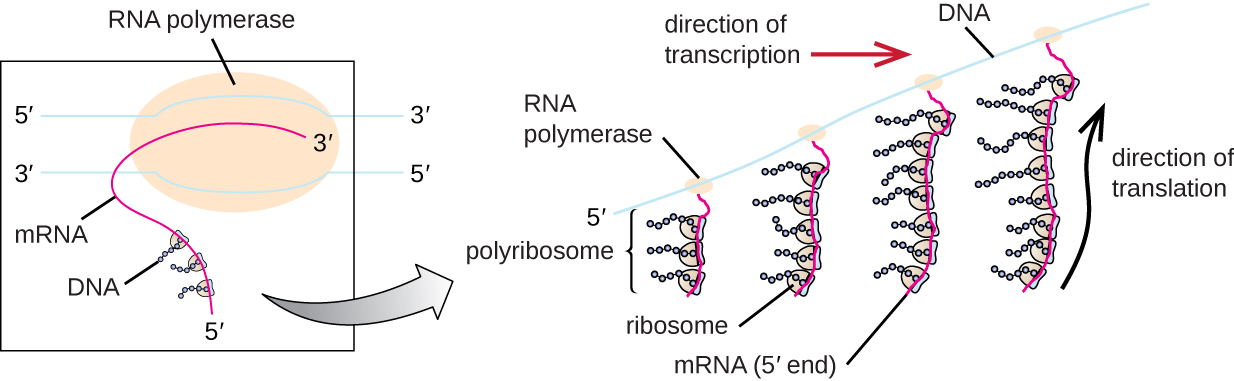
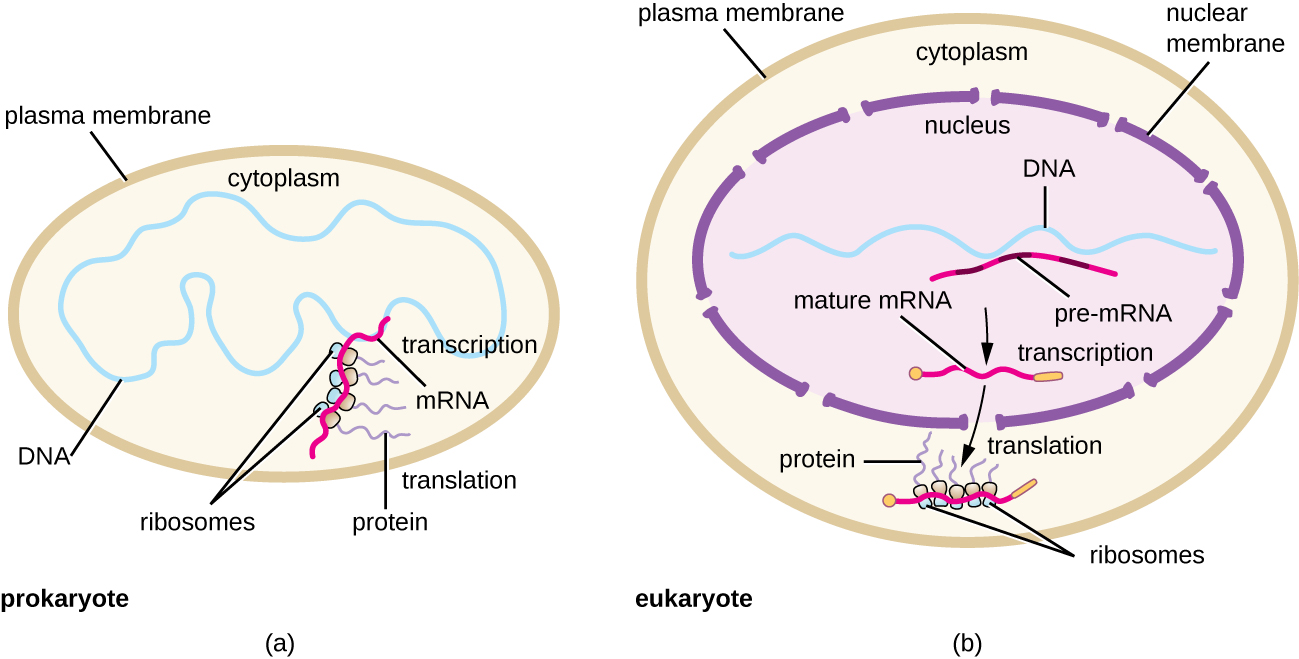
Cada molécula de ARNm es traducida simultáneamente por muchos ribosomas, todos sintetizando proteínas en la misma dirección: leyendo el ARNm de 5' a 3' y sintetizando el polipéptido desde el extremo N hasta el extremo C. La estructura completa que contiene un ARNm con múltiples ribosomas asociados se llama poliribosoma (o polisoma). Tanto en bacterias como en arqueas, antes de que ocurra la terminación de la transcripción, cada transcrito que codifica proteína ya se está utilizando para iniciar la síntesis de numerosas copias del polipéptido (s) codificado (s) porque los procesos de transcripción y traducción pueden ocurrir simultáneamente, formando poliribosomas (Figura \(\PageIndex{2}\)). La razón por la que la transcripción y la traducción pueden ocurrir simultáneamente es porque ambos procesos ocurren en la misma dirección 5' a 3', ambos ocurren en el citoplasma de la célula, y porque el transcrito de ARN no se procesa una vez que se transcribe. Esto permite que una célula procariota responda a una señal ambiental que requiere nuevas proteínas muy rápidamente. En contraste, en las células eucariotas, la transcripción y traducción simultánea no es posible. Aunque los poliribosomas también se forman en eucariotas, no pueden hacerlo hasta que la síntesis de ARN esté completa y la molécula de ARN haya sido modificada y transportada fuera del núcleo.
RNAs de transferencia
Los ARN de transferencia (ARNt) son moléculas de ARN estructurales y, dependiendo de la especie, existen muchos tipos diferentes de ARNt en el citoplasma. Las especies bacterianas suelen tener entre 60 y 90 tipos. Sirviendo como adaptadores, cada tipo de ARNt se une a un codón específico en el molde de ARNm y agrega el aminoácido correspondiente a la cadena polipeptídica. Por lo tanto, los ARNt son las moléculas que realmente “traducen” el lenguaje del ARN al lenguaje de las proteínas. Como moléculas adaptadoras de traducción, es sorprendente que los ARNt puedan encajar tanta especificidad en un paquete tan pequeño. La molécula de ARNt interactúa con tres factores: aminoacil ARNt sintetasas, ribosomas y ARNm.
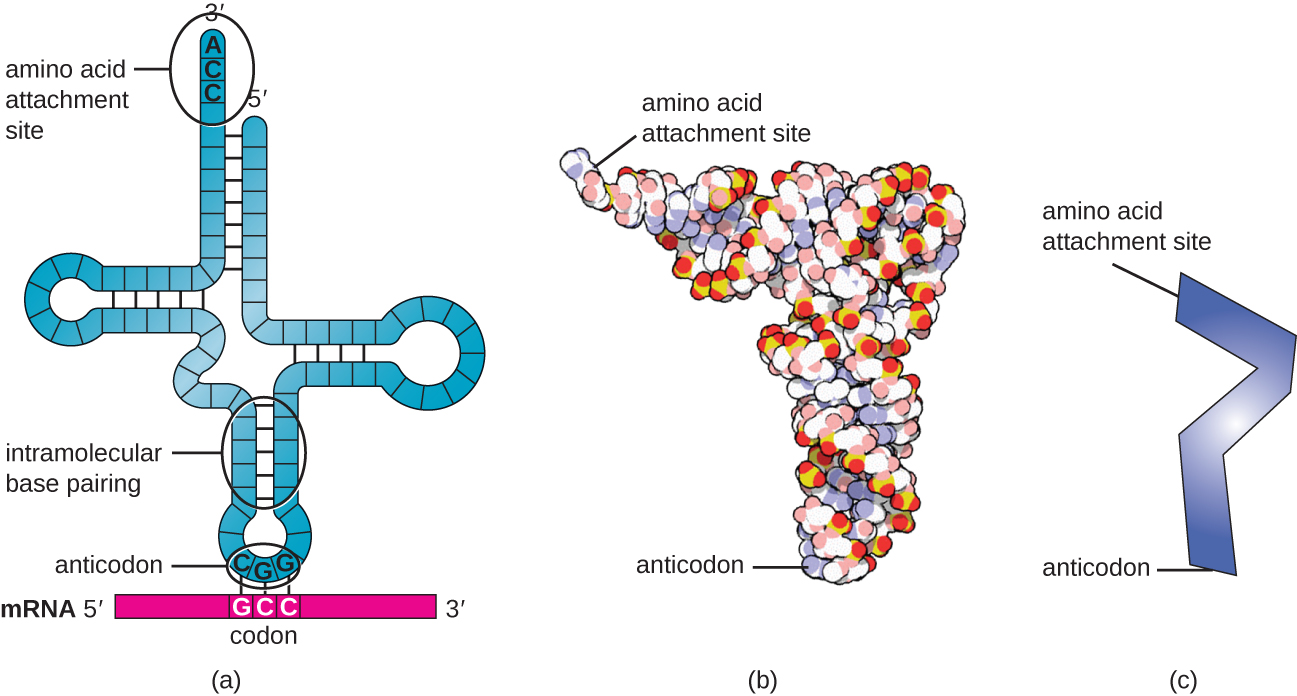
Los ARNt maduros adquieren una estructura tridimensional cuando las bases complementarias expuestas en la molécula de ARN monocatenario se unen entre sí (Figura\(\PageIndex{3}\)). Esta forma posiciona el sitio de unión de aminoácidos, llamado extremo de unión de aminoácidos CCA, que es una secuencia de citosina-citosina-adenina en el extremo 3' del ARNt, y el anticodonato en el otro extremo. El anticodón es una secuencia de tres nucleótidos que se une con un codón de ARNm a través del emparejamiento de bases complementarias.
Se agrega un aminoácido al extremo de una molécula de ARNt a través del proceso de “carga” de ARNt, durante el cual cada molécula de ARNt se une a su aminoácido correcto o afín por un grupo de enzimas llamadas aminoacil ARNt sintetasas. Al menos un tipo de aminoacil ARNt sintetasa existe para cada uno de los 20 aminoácidos. Durante este proceso, el aminoácido se activa primero mediante la adición de monofosfato de adenosina (AMP) y luego se transfiere al ARNt, convirtiéndolo en un ARNt cargado, y se libera AMP.
Ejercicio\(\PageIndex{2}\)
- Describir la estructura y composición del ribosoma procariota.
- ¿En qué dirección se lee la plantilla de ARNm?
- Describir la estructura y función de un ARNt.
El mecanismo de síntesis de proteínas
La traducción es similar en procariotas y eucariotas. Aquí exploraremos cómo se produce la traducción en E. coli, un procariota representativo, y especificaremos cualquier diferencia entre la traducción bacteriana y eucariota.
Iniciación
El inicio de la síntesis de proteínas comienza con la formación de un complejo de iniciación. En E. coli, este complejo involucra el pequeño ribosoma 30S, el molde de ARNm, tres factores de iniciación que ayudan al ribosoma a ensamblarse correctamente, trifosfato de guanosina (GTP) que actúa como fuente de energía, y un ARNt iniciador especial que porta N-formil-metionina (fMET-ARNt fMet ) (Figura\(\PageIndex{4}\)). El ARNt iniciador interactúa con el codón de inicio AUG del ARNm y porta una metionina formilada (fMET). Debido a su implicación en la iniciación, fMET se inserta al inicio (extremo N) de cada cadena polipeptídica sintetizada por E. coli. En el ARNm de E. coli, una secuencia líder aguas arriba del primer codón AUG, llamada secuencia Shine-Dalgarno (también conocida como el sitio de unión ribosómica AGGAGG), interactúa a través del apareamiento de bases complementarias con las moléculas de ARNr que componen el ribosoma. Esta interacción ancla la subunidad ribosómica 30S en la ubicación correcta en el molde de ARNm. En este punto, la subunidad ribosómica 50S luego se une al complejo de iniciación, formando un ribosoma intacto.
En eucariotas, la formación del complejo de iniciación es similar, con las siguientes diferencias:
- El ARNt iniciador es un ARNt especializado diferente que porta metionina, llamado Met-ARNt
- En lugar de unirse al ARNm en la secuencia de Shine-Dalgarno, el complejo de iniciación eucariota reconoce la tapa 5' del ARNm eucariota, luego rastrea a lo largo del ARNm en la dirección 5' a 3' hasta que se reconoce el codón de inicio AUG. En este punto, la subunidad 60S se une al complejo de Met-ARNt, ARNm y la subunidad 40S.
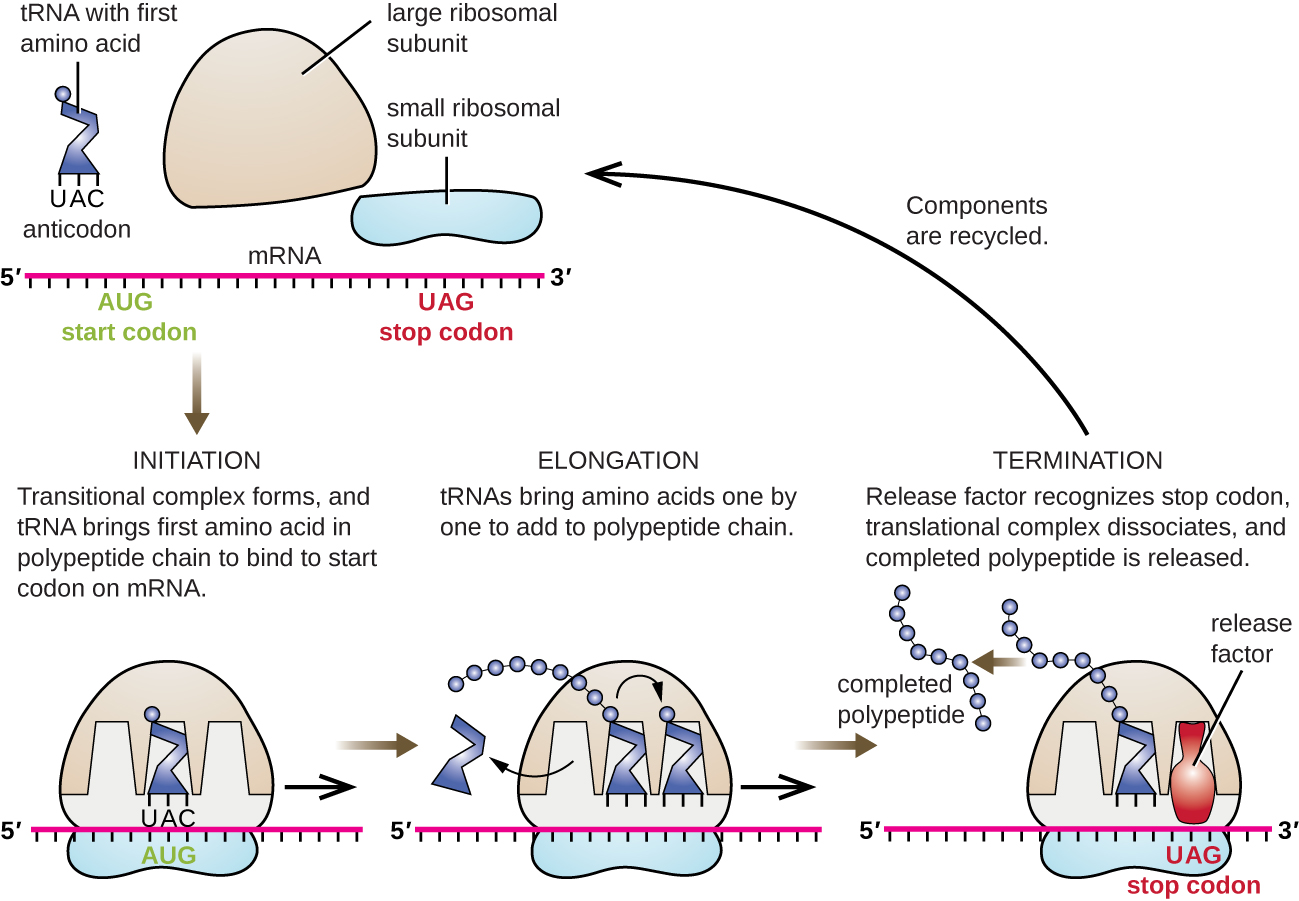
Alargamiento
En procariotas y eucariotas, los fundamentos del alargamiento de la traducción son los mismos. En E. coli, la unión de la subunidad ribosómica 50S para producir el ribosoma intacto forma tres sitios ribosómicos funcionalmente importantes: El sitio A (aminoacilo) se une a los ARNt de aminoacilo cargados entrantes. El sitio P (peptidilo) se une a ARNt cargados que portan aminoácidos que han formado enlaces peptídicos con la cadena polipeptídica en crecimiento pero que aún no se han disociado de su ARNt correspondiente. El sitio E (salida) libera ARNt disociados para que puedan recargarse con aminoácidos libres. Existe una notable excepción a esta línea de ensamblaje de ARNt: Durante la formación del complejo de iniciación, FMET- ARNt bacteriano FMET o met-ARNt eucariota ingresa al sitio P directamente sin ingresar primero al sitio A, proporcionando un sitio A libre listo para aceptar el ARNt correspondiente al primer codón después el AUG.
La elongación procede con movimientos de un solo codón del ribosoma, cada uno llamado evento de translocación. Durante cada evento de translocación, los ARNt cargados ingresan en el sitio A, luego se desplazan al sitio P y luego finalmente al sitio E para su eliminación. Los movimientos ribosómicos, o pasos, son inducidos por cambios conformacionales que hacen avanzar al ribosoma por tres bases en la dirección 3'. Se forman enlaces peptídicos entre el grupo amino del aminoácido unido al ARNt del sitio A y el grupo carboxilo del aminoácido unido al ARNt del sitio P. La formación de cada enlace peptídico es catalizada por peptidil-transferasa, una ribozima basada en ARN que se integra en la subunidad ribosómica 50S. El aminoácido unido al ARNt del sitio P también está unido a la cadena polipeptídica en crecimiento. A medida que el ribosoma atraviesa el ARNm, el antiguo ARNt del sitio P entra en el sitio E, se separa del aminoácido y se expulsa. Varias de las etapas durante la elongación, incluyendo la unión de un aminoacil ARNt cargado al sitio A y la translocación, requieren energía derivada de la hidrólisis de GTP, la cual es catalizada por factores de elongación específicos. Sorprendentemente, el aparato de traducción de E. coli tarda solo 0.05 segundos en agregar cada aminoácido, lo que significa que una proteína de 200 aminoácidos se puede traducir en solo 10 segundos.
Terminación
La terminación de la traducción ocurre cuando se encuentra un codón sin sentido (UAA, UAG o UGA) para el cual no hay ARNt complementario. Al alinearse con el sitio A, estos codones sin sentido son reconocidos por factores de liberación en procariotas y eucariotas que dan como resultado que el aminoácido del sitio P se desprenda de su ARNt, liberando el polipéptido recién hecho. Las subunidades ribosómicas pequeñas y grandes se disocian del ARNm y entre sí; son reclutadas casi inmediatamente en otro complejo de iniciación de traducción.
En resumen, hay varias características clave que distinguen la expresión génica procariota de la que se ve en eucariotas. Estos se ilustran en la Figura\(\PageIndex{5}\) y se enumeran en la Figura\(\PageIndex{6}\).
Orientación, plegamiento y modificación de proteínas
Durante y después de la traducción, los polipéptidos pueden necesitar ser modificados antes de que sean biológicamente activos. Las modificaciones posteriores a la traducción incluyen:
- eliminación de secuencias señal traducidas: colas cortas de aminoácidos que ayudan a dirigir una proteína a un compartimento celular específico
- “" "plegamiento "” apropiado del polipéptido y asociación de múltiples subunidades polipeptídicas, a menudo facilitadas por proteínas chaperonas, en una estructura tridimensional distinta
- procesamiento proteolítico de un polipéptido inactivo para liberar un componente proteico activo, y
- diversas modificaciones químicas (por ejemplo, fosforilación, metilación o glicosilación) de aminoácidos individuales.
Ejercicio\(\PageIndex{3}\)
- ¿Cuáles son los componentes del complejo de iniciación para la traducción en procariotas?
- ¿Cuáles son dos diferencias entre el inicio de la traducción procariota y la eucariota?
- ¿Qué ocurre en cada uno de los tres sitios activos del ribosoma?
- ¿Qué causa la terminación de la traducción?
Conceptos clave y resumen
- En la traducción, los polipéptidos se sintetizan usando secuencias de ARNm y maquinaria celular, incluyendo ARNt que emparejan codones de ARNm con aminoácidos específicos y ribosomas compuestos por ARN y proteínas que catalizan la reacción.
- El código genético es degenerado ya que varios codones de ARNm codifican para los mismos aminoácidos. El código genético es casi universal entre los organismos vivos.
- Los ribosomas procariotas (70S) y eucariotas citoplásmicos (80S) están compuestos cada uno por una subunidad grande y una subunidad pequeña de diferentes tamaños entre los dos grupos. Cada subunidad está compuesta por ARNr y proteína. Los ribosomas de orgánulos en células eucariotas se asemejan a los ribosomas procariotas.
- Entre 60 y 90 especies de ARNt existen en bacterias. Cada ARNt tiene un anticodón de tres nucleótidos así como un sitio de unión para un aminoácido afín. Todos los ARNt con un anticodón específico portarán el mismo aminoácido.
- El inicio de la traducción ocurre cuando la subunidad ribosómica pequeña se une con factores de iniciación y un ARNt iniciador en el codón de inicio de un ARNm, seguido de la unión al complejo de iniciación de la subunidad ribosómica grande.
- En las células procariotas, el codón de inicio codifica la N-formil-metionina transportada por un ARNt iniciador especial. En las células eucariotas, el codón de inicio codifica la metionina transportada por un ARNt iniciador especial. Además, mientras que la unión ribosómica del ARNm en procariotas se ve facilitada por la secuencia de Shine-Dalgarno dentro del ARNm, los ribosomas eucariotas se unen a la tapa 5' del ARNm.
- Durante la etapa de elongación de la traducción, un ARNt cargado se une al ARNm en el sitio A del ribosoma; se cataliza un enlace peptídico entre los dos aminoácidos adyacentes, rompiendo el enlace entre el primer aminoácido y su ARNt; el ribosoma mueve un codón a lo largo del ARNm; y el primer ARNt se mueve del sitio P del ribosoma al sitio E y abandona el complejo ribosómico.
- La terminación de la traducción ocurre cuando el ribosoma encuentra un codón de parada, que no codifica un ARNt. Los factores de liberación provocan la liberación del polipéptido y el complejo ribosómico se disocia.
- En procariotas, la transcripción y la traducción pueden acoplarse, con la traducción de una molécula de ARNm comenzando tan pronto como la transcripción permita suficiente exposición al ARNm para la unión de un ribosoma, antes de la terminación de la transcripción. La transcripción y la traducción no se acoplan en eucariotas porque la transcripción ocurre en el núcleo, mientras que la traducción ocurre en el citoplasma o en asociación con el retículo endoplásmico rugoso.
- Los polipéptidos a menudo requieren una o más modificaciones postraduccionales para llegar a ser biológicamente activos.


