11.2: Replicación de ADN
- Page ID
- 54608
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Objetivos de aprendizaje
- Explicar el significado de la replicación semiconservativa del ADN
- Explicar por qué la replicación del ADN es bidireccional e incluye tanto una hebra líder como una retrasada
- Explicar por qué se forman los fragmentos de Okazaki
- Describir el proceso de replicación del ADN y las funciones de las enzimas involucradas
- Identificar las diferencias entre la replicación del ADN en bacterias y eucariotas
- Explicar el proceso de replicación de círculo rodante
La elucidación de la estructura de la doble hélice por James Watson y Francis Crick en 1953 proporcionó una pista sobre cómo se copia el ADN durante el proceso de replicación. La separación de las hebras de la doble hélice proporcionaría dos plantillas para la síntesis de nuevas cadenas complementarias, pero aún no estaba claro exactamente cómo se construyeron nuevas moléculas de ADN. En un modelo, la replicación semiconservativa, las dos cadenas de la doble hélice se separan durante la replicación del ADN, y cada hebra sirve como molde a partir del cual se copia la nueva cadena complementaria; después de la replicación, cada ADN bicatenario incluye una hebra parental o “vieja” y una hebra “nueva”. También se sugirieron dos modelos competidores: conservador y dispersivo, los cuales se muestran en la Figura\(\PageIndex{1}\).

Matthew Meselson (1930—) y Franklin Stahl (1929—) idearon un experimento en 1958 para probar cuál de estos modelos representa correctamente la replicación del ADN (Figura\(\PageIndex{2}\)). Cultivaron E. coli durante varias generaciones en un medio que contenía un isótopo “pesado” de nitrógeno (15 N) que se incorporó a bases nitrogenadas y, finalmente, al ADN. Esto etiquetó el ADN parental. El cultivo de E. coli se desplazó a un medio que contenía 14 N y se dejó crecer por una generación. Se cosecharon las células y se aisló el ADN. El ADN se separó por ultracentrifugación, durante la cual el ADN formó bandas de acuerdo a su densidad. Se esperaría que el ADN cultivado en 15 N forme una banda en una posición de mayor densidad que la cultivada en 14 N. Meselson y Stahl señalaron que después de una generación de crecimiento en 14 N, la banda única observada fue intermedia en posición entre ADN de células cultivadas exclusivamente en 15 N o 14 N. Esto sugirió un modo de replicación semiconservativo o dispersivo. Algunas células se dejaron crecer por una generación más en 14 N y se volvieron a girar. El ADN recolectado de células cultivadas durante dos generaciones en 14 N formó dos bandas: una banda de ADN estaba en la posición intermedia entre 15 N y 14 N, y la otra correspondió a la banda de ADN de 14 N. Estos resultados sólo podrían explicarse si el ADN se replica de manera semiconservativa. Por lo tanto, se descartaron los otros dos modelos. Como resultado de este experimento, ahora sabemos que durante la replicación del ADN, cada una de las dos hebras que componen la doble hélice sirve como plantilla a partir de la cual se copian nuevas hebras. La nueva hebra será complementaria a la hebra parental o “vieja”. Las moléculas de ADN resultantes tienen la misma secuencia y se dividen en partes iguales en las dos células hijas.
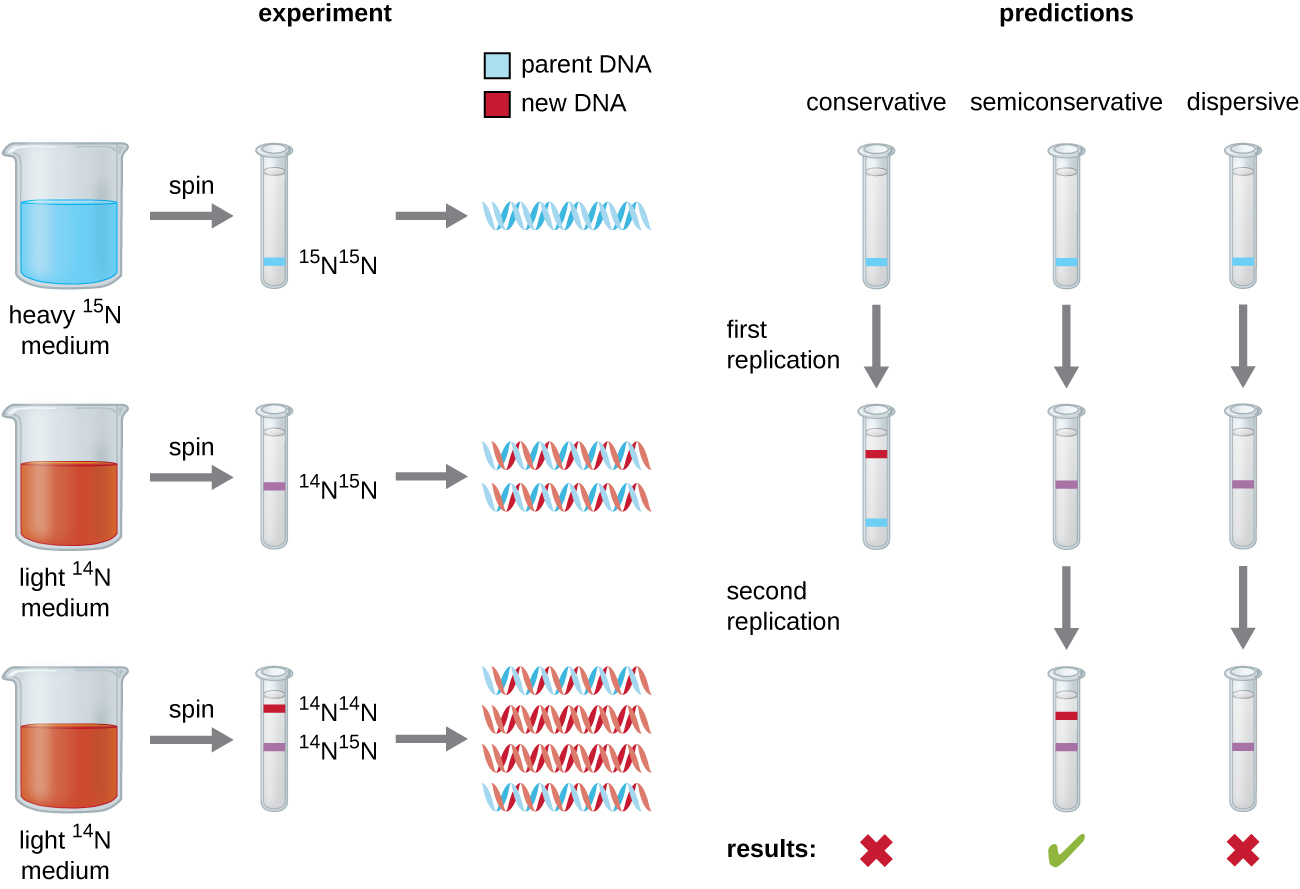
Ejercicio\(\PageIndex{1}\)
¿Cuál habría sido la conclusión del experimento de Meselson y Stahl si, después de la primera generación, hubieran encontrado dos bandas de ADN?
Replicación de ADN en bacterias
La replicación del ADN ha sido bien estudiada en bacterias principalmente por el pequeño tamaño del genoma y los mutantes disponibles. E. coli tiene 4.6 millones de pares de bases (Mbp) en un solo cromosoma circular y todo se replica en aproximadamente 42 minutos, partiendo de un único origen de replicación y procediendo alrededor del círculo bidireccionalmente (es decir, en ambas direcciones). Esto significa que se agregan aproximadamente 1000 nucleótidos por segundo. El proceso es bastante rápido y ocurre con pocos errores.
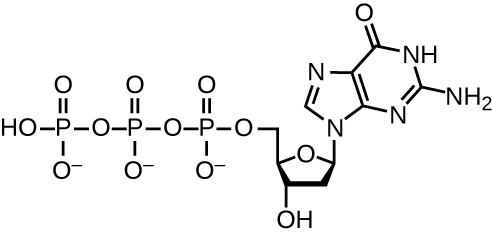
La replicación del ADN utiliza una gran cantidad de proteínas y enzimas (Tabla\(\PageIndex{1}\)). Uno de los actores clave es la enzima ADN polimerasa, también conocida como ADN pol. En bacterias, se conocen tres tipos principales de ADN polimerasas: ADN pol I, ADN pol II y ADN pol III. Ahora se sabe que el ADN pol III es la enzima requerida para la síntesis de ADN; el ADN pol I y el ADN pol II se requieren principalmente para su reparación. DNA pol III agrega desoxirribonucleótidos cada uno complementario a un nucleótido en la cadena molde, uno por uno al grupo 3'-OH de la cadena de ADN en crecimiento. La adición de estos nucleótidos requiere energía. Esta energía está presente en los enlaces de tres grupos fosfato unidos a cada nucleótido (un nucleótido trifosfato), similar a cómo se almacena la energía en los enlaces fosfato de adenosina trifosfato (ATP) (Figura\(\PageIndex{3}\)). Cuando se rompe el enlace entre los fosfatos y se libera el difosfato, la energía liberada permite la formación de un enlace fosfodiéster covalente por síntesis de deshidratación entre el nucleótido entrante y el grupo 3'-OH libre en la cadena de ADN en crecimiento.
Iniciación
El inicio de la replicación ocurre en una secuencia específica de nucleótidos llamada origen de replicación, donde diversas proteínas se unen para iniciar el proceso de replicación. E. coli tiene un único origen de replicación (al igual que la mayoría de los procariotas), llamado ORi, en su único cromosoma. El origen de replicación es de aproximadamente 245 pares de bases de longitud y es rico en secuencias de adenina-timina (AT).
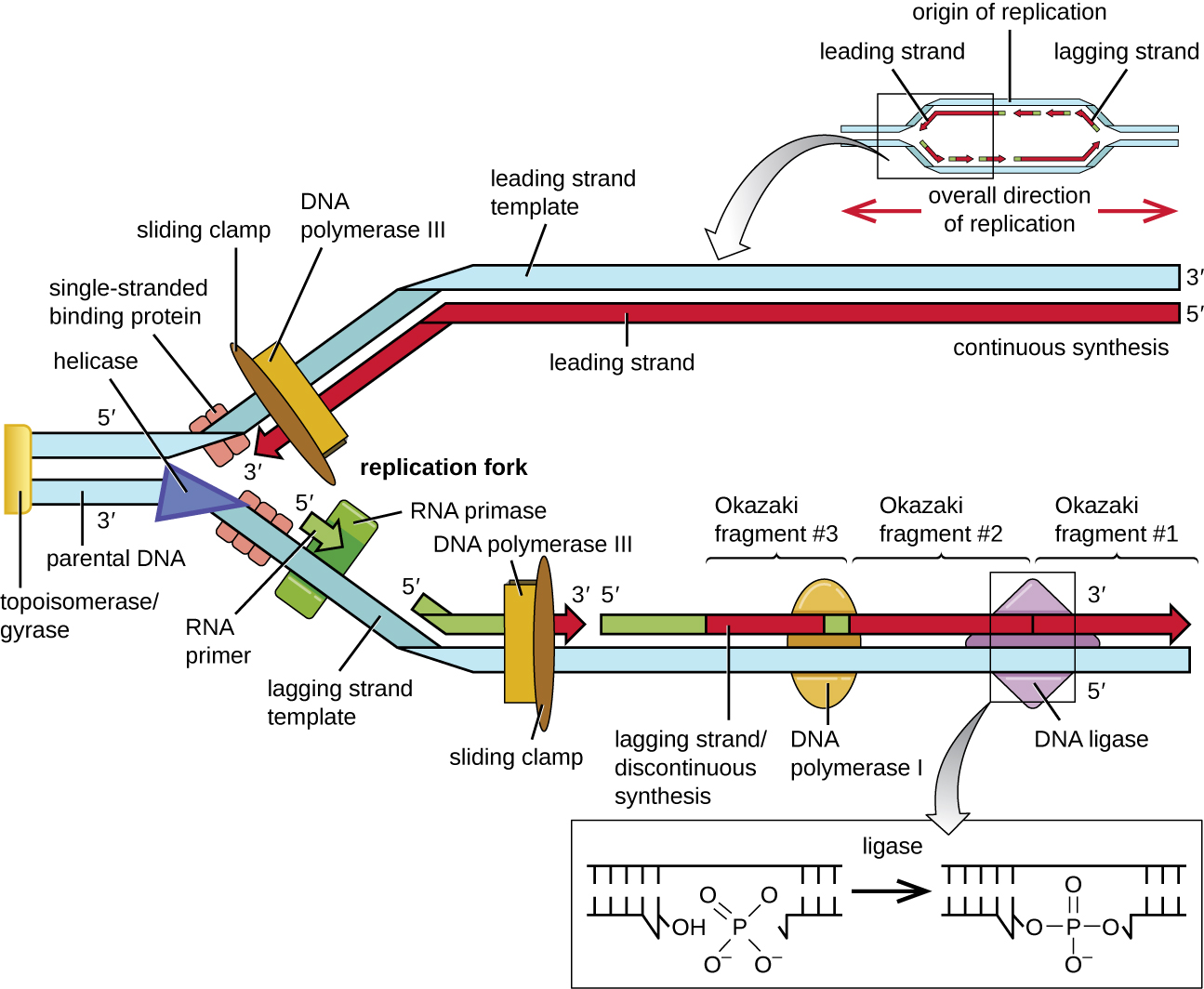
Algunas de las proteínas que se unen al origen de replicación son importantes para hacer que las regiones monocatenarias de ADN sean accesibles para la replicación. El ADN cromosómico suele estar envuelto alrededor de histonas (en eucariotas y arqueas) o proteínas similares a histonas (en bacterias), y está superenrollado, o envuelto extensamente y retorcido sobre sí mismo. Este empaque hace que la información en la molécula de ADN sea inaccesible. Sin embargo, las enzimas llamadas topoisomerasas cambian la forma y el superenrollamiento del cromosoma. Para que comience la replicación del ADN bacteriano, el cromosoma superenrollado se relaja con la topoisomerasa II, también llamada ADN girasa. Una enzima llamada helicasa luego separa las cadenas de ADN rompiendo los enlaces de hidrógeno entre los pares de bases nitrogenadas. Recordemos que las secuencias AT tienen menos enlaces de hidrógeno y, por lo tanto, tienen interacciones más débiles que las secuencias de guanina-citosina (GC). Estas enzimas requieren hidrólisis de ATP. A medida que el ADN se abre, se forman estructuras en forma de Y llamadas horquillas de replicación. Dos horquillas de replicación se forman en el origen de la replicación, permitiendo la replicación bidireccional y la formación de una estructura que parece una burbuja cuando se ve con un microscopio electrónico de transmisión; como resultado, esta estructura se denomina burbuja de replicación. El ADN cerca de cada horquilla de replicación está recubierto con proteínas de unión monocatenarias para evitar que el ADN monocatenario se reenrolle en una doble hélice.
Una vez que el ADN monocatenario es accesible en el origen de la replicación, puede comenzar la replicación del ADN. Sin embargo, ADN pol III es capaz de agregar nucleótidos solo en la dirección 5' a 3' (una nueva cadena de ADN solo puede extenderse en esta dirección). Esto se debe a que la ADN polimerasa requiere un grupo 3'-OH libre al que puede añadir nucleótidos formando un enlace fosfodiéster covalente entre el extremo 3'-OH y el fosfato 5' del siguiente nucleótido. Esto también significa que no puede agregar nucleótidos si no se dispone de un grupo 3'-OH libre, lo que es el caso de una sola cadena de ADN. El problema se resuelve con la ayuda de una secuencia de ARN que proporciona el extremo 3'-OH libre. Debido a que esta secuencia permite el inicio de la síntesis de ADN, se le llama apropiadamente el cebador. El cebador tiene una longitud de cinco a 10 nucleótidos y es complementario al ADN parental o molde. Se sintetiza por ARN primasa, que es una ARN polimerasa. A diferencia de las ADN polimerasas, las ARN polimerasas no necesitan un grupo 3'-OH libre para sintetizar una molécula de ARN. Ahora que el cebador proporciona el grupo 3'-OH libre, la ADN polimerasa III ahora puede extender este cebador de ARN, agregando nucleótidos de ADN uno por uno que son complementarios a la cadena molde (Figura\(\PageIndex{1}\)).
Alargamiento
Durante la elongación en la replicación del ADN, la adición de nucleótidos ocurre a su velocidad máxima de aproximadamente 1000 nucleótidos por segundo. La ADN polimerasa III solo puede extenderse en la dirección 5' a 3', lo que plantea un problema en la horquilla de replicación. La doble hélice del ADN es antiparalela; es decir, una hebra está orientada en la dirección 5' a 3' y la otra está orientada en la dirección 3' a 5' (ver Estructura y Función del ADN). Durante la replicación, una hebra, que es complementaria a la cadena de ADN parental de 3' a 5', se sintetiza continuamente hacia la horquilla de replicación porque la polimerasa puede agregar nucleótidos en esta dirección. Esta cadena sintetizada continuamente se conoce como la cadena principal. La otra cadena, complementaria al ADN parental 5' a 3', crece alejándose de la horquilla de replicación, por lo que la polimerasa debe retroceder hacia la horquilla de replicación para comenzar a agregar bases a un nuevo cebador, nuevamente en la dirección que se aleja de la horquilla de replicación. Lo hace hasta que se topa con la hebra previamente sintetizada y luego vuelve a retroceder (Figura\(\PageIndex{4}\)). Estas etapas producen pequeños fragmentos de secuencia de ADN conocidos como fragmentos de Okazaki, cada uno separado por cebador de ARN. Los fragmentos de Okazaki llevan el nombre del equipo de investigación japonés y de la pareja casada Reiji y Tsuneko Okazaki, quienes los descubrieron por primera vez en 1966. La hebra con los fragmentos de Okazaki se conoce como la hebra rezagada, y se dice que su síntesis es discontinua.
La cadena principal se puede extender a partir de un cebador solo, mientras que la hebra rezagada necesita un nuevo cebador para cada uno de los fragmentos cortos de Okazaki. La dirección general de la hebra retrasada será de 3' a 5', y la de la cadena delantera 5' a 3'. Una proteína llamada pinza deslizante mantiene la ADN polimerasa en su lugar a medida que continúa agregando nucleótidos. La pinza deslizante es una proteína en forma de anillo que se une al ADN y mantiene la polimerasa en su lugar. Más allá de su papel en la iniciación, la topoisomerasa también evita el sobrebobinado de la doble hélice del ADN antes de la horquilla de replicación a medida que el ADN se está abriendo; lo hace al provocar cortes temporales en la hélice del ADN y luego volver a sellarla. A medida que avanza la síntesis, los cebadores de ARN son reemplazados por ADN. Los cebadores se eliminan por la actividad exonucleasa de la ADN polimerasa I, y se rellenan los huecos. Las muescas que permanecen entre el ADN recién sintetizado (que reemplazó al cebador de ARN) y el ADN previamente sintetizado son selladas por la enzima ADN ligasa que cataliza la formación de enlace fosfodiéster covalente entre el extremo 3'-OH de un fragmento de ADN y el extremo 5' fosfato del otro fragmento, estabilizar la cadena principal de azúcar-fosfato de la molécula de ADN.
Terminación
Una vez que el cromosoma completo ha sido replicado, debe ocurrir la terminación de la replicación del ADN. Aunque se sabe mucho sobre el inicio de la replicación, se sabe menos sobre el proceso de terminación. Después de la replicación, los genomas circulares completos resultantes de procariotas se concatenan, lo que significa que los cromosomas circulares de ADN están entrelazados y deben separarse entre sí. Esto se logra a través de la actividad de la topoisomerasa bacteriana IV, que introduce roturas bicatenarias en las moléculas de ADN, permitiendo que se separen entre sí; la enzima luego vuelve a sellar los cromosomas circulares. La resolución de concatémeros es un problema exclusivo de la replicación del ADN procariota debido a sus cromosomas circulares. Debido a que tanto la ADN girasa bacteriana como la topoisomerasa IV son distintas de sus contrapartes eucariotas, estas enzimas sirven como dianas para una clase de medicamentos antimicrobianos llamados quinolonas.
| Enzima o Factor | Función |
|---|---|
| ADN pol I | La actividad exonucleasa elimina el cebador de ARN y lo reemplaza con ADN recién sintetizado |
| ADN pol III | Enzima principal que añade nucleótidos en la dirección 5' a 3' |
| Helicasa | Abre la hélice de ADN rompiendo los enlaces de hidrógeno entre las bases nitrogenadas |
| Ligasa | Sella los huecos entre los fragmentos de Okazaki en la hebra rezagada para crear una cadena de ADN continua |
| Primase | Sintetica los cebadores de ARN necesarios para iniciar la replicación |
| Proteínas de unión de cadena | Se une al ADN monocatenario para evitar el enlace de hidrógeno entre las cadenas de ADN, reformando el ADN bicatenario |
| Pinza deslizante | Ayuda a mantener el ADN pol III en su lugar cuando se agregan nucleótidos |
| Topoisomerasa II (ADN girasa) | Relaja el cromosoma superenrollado para hacer que el ADN sea más accesible para el inicio de la replicación; ayuda a aliviar el estrés sobre el ADN al desenrollar, al causar roturas y luego volver a sellar el ADN |
| Topoisomerasa IV | Introduce la ruptura monocatenaria en cromosomas concatenados para liberarlos unos de otros y luego vuelve a cerrar el ADN |
Ejercicio\(\PageIndex{2}\)
- ¿Qué enzima rompe los enlaces de hidrógeno que mantienen unidas las dos cadenas de ADN para que pueda ocurrir la replicación?
- ¿Es la hebra rezagada o la hebra delantera la que se sintetiza en la dirección hacia la apertura de la horquilla de replicación?
- ¿Qué enzima se encarga de eliminar los cebadores de ARN en el ADN bacteriano recién replicado?
Replicación de ADN en eucariotas
Los genomas eucariotas son mucho más complejos y más grandes que los genomas procariotas y suelen estar compuestos por múltiples cromosomas lineales (Tabla\(\PageIndex{2}\)). El genoma humano, por ejemplo, tiene 3 mil millones de pares de bases por conjunto haploide de cromosomas, y 6 mil millones de pares de bases se insertan durante la replicación. Existen múltiples orígenes de replicación en cada cromosoma eucariota (Figura\(\PageIndex{5}\)); el genoma humano tiene 30,000 a 50,000 orígenes de replicación. La tasa de replicación es de aproximadamente 100 nucleótidos por segundo, 10 veces más lenta que la replicación procariota.
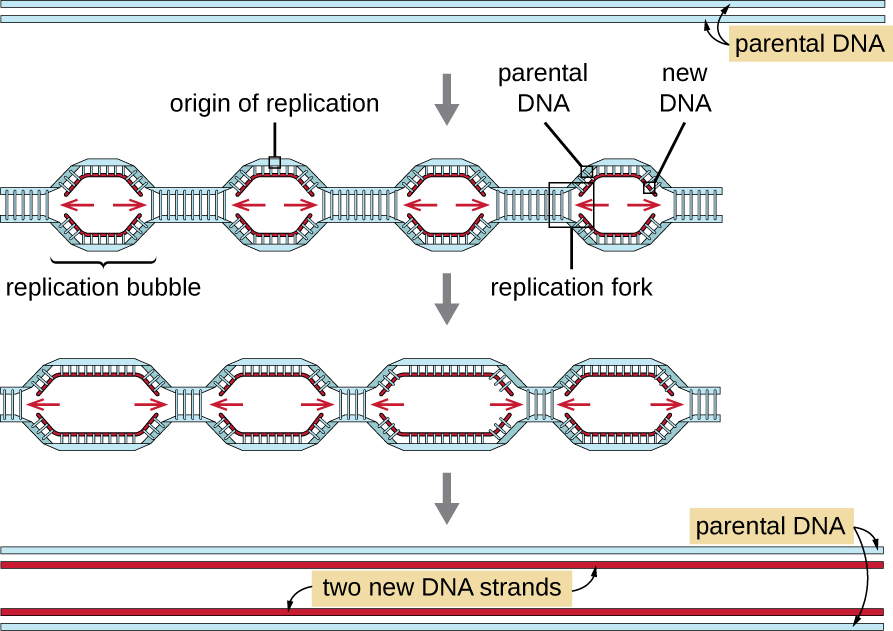
Los pasos esenciales de replicación en eucariotas son los mismos que en los procariotas. Antes de que pueda comenzar la replicación, el ADN tiene que estar disponible como molde. El ADN eucariota está altamente superenrollado y empaquetado, lo que es facilitado por muchas proteínas, incluidas las histonas (ver Estructura y función de los genomas celulares). En el origen de la replicación, un complejo de prereplicación compuesto por varias proteínas, incluyendo helicasa, forma y recluta otras enzimas involucradas en el inicio de la replicación, incluyendo topoisomerasa para relajar el superenrollamiento, proteína de unión monocatenaria, ARN primasa y ADN polimerasa. Tras el inicio de la replicación, en un proceso similar al encontrado en procariotas, el alargamiento es facilitado por las ADN polimerasas eucariotas. La cadena principal es sintetizada continuamente por la enzima polimerasa eucariota pol δ, mientras que la hebra rezagada es sintetizada por pol ε. Una proteína de pinza deslizante mantiene la ADN polimerasa en su lugar para que no se caiga del ADN. La enzima ribonucleasa H (RNasa H), en lugar de una ADN polimerasa como en bacterias, elimina el cebador de ARN, que luego se reemplaza con nucleótidos de ADN. Los huecos que quedan son sellados por ADN ligasa.
Debido a que los cromosomas eucariotas son lineales, uno podría esperar que su replicación sea más sencilla. Al igual que en los procariotas, la ADN polimerasa eucariota puede agregar nucleótidos solo en la dirección 5' a 3'. En la cadena principal, la síntesis continúa hasta que alcanza el final del cromosoma u otra horquilla de replicación progresando en la dirección opuesta. En la hebra rezagada, el ADN se sintetiza en tramos cortos, cada uno de los cuales es iniciado por un cebador separado. Cuando la horquilla de replicación alcanza el final del cromosoma lineal, no hay lugar para hacer un cebador para que el fragmento de ADN se copie al final del cromosoma. Estos extremos permanecen así desapareados y, con el tiempo, pueden acortarse progresivamente a medida que las células continúan dividiéndose.
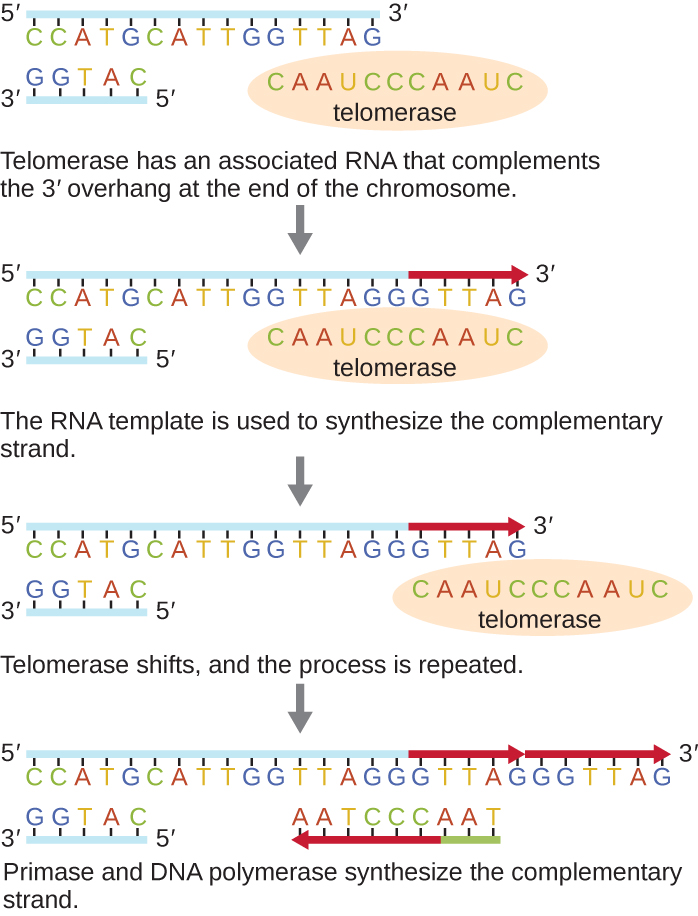
Los extremos de los cromosomas lineales se conocen como telómeros y consisten en secuencias repetitivas no codificantes. Los telómeros protegen las secuencias codificantes de que se pierdan a medida que las células continúan dividiéndose. En humanos, una secuencia de seis pares de bases, TTAGGG, se repite de 100 a 1000 veces para formar el telómero. El descubrimiento de la enzima telomerasa (Figura\(\PageIndex{6}\)) aclaró nuestra comprensión de cómo se mantienen los extremos cromosómicos. La telomerasa contiene una parte catalítica y un molde de ARN incorporado. Se une al extremo del cromosoma, y se agregan bases complementarias al molde de ARN en el extremo 3' de la cadena de ADN. Una vez que el extremo 3' de la plantilla de hebra retrasada es suficientemente alargado, la ADN polimerasa puede agregar los nucleótidos complementarios a los extremos de los cromosomas. De esta manera, se replican los extremos de los cromosomas. En humanos, la telomerasa es típicamente activa en células germinales y células madre adultas; no es activa en células somáticas adultas y puede estar asociada con el envejecimiento de estas células. Los microbios eucariotas, incluyendo hongos y protozoos, también producen telomerasa para mantener la integridad cromosómica. Por su descubrimiento de la telomerasa y su acción, Elizabeth Blackburn (1948—) recibió el Premio Nobel de Medicina o Fisiología en 2009.
| Inmueble | Bacterias | Eucariotes |
|---|---|---|
| Estructura del genoma | Cromosoma circular único | Cromosomas lineales múltiples |
| Número de orígenes por cromosoma | Sencillo | Múltiple |
| Tasa de replicación | 1000 nucleótidos por segundo | 100 nucleótidos por segundo |
| Telomerasa | No presente | Presente |
| Eliminación de cebador de ARN | ADN pol I | RNasa H |
| Alargamiento del hilo | ADN pol III | pol δ, pol ε |
Ejercicio\(\PageIndex{3}\)
- ¿En qué se diferencia el origen de replicación entre eucariotas y procariotas?
- ¿Qué enzimas polimerasa son responsables de la síntesis de ADN durante la replicación eucariota?
- ¿Qué se encuentra en los extremos de los cromosomas en eucariotas y por qué?
Replicación de ADN de Elementos Extracromosómicos: Plásmidos y Virus
Para copiar sus ácidos nucleicos, los plásmidos y virus utilizan frecuentemente variaciones en el patrón de replicación del ADN descrito para los genomas procariotas. Para obtener más información sobre la amplia gama de estrategias de replicación viral, consulte El ciclo de vida viral.
Replicación Círculo Rodante
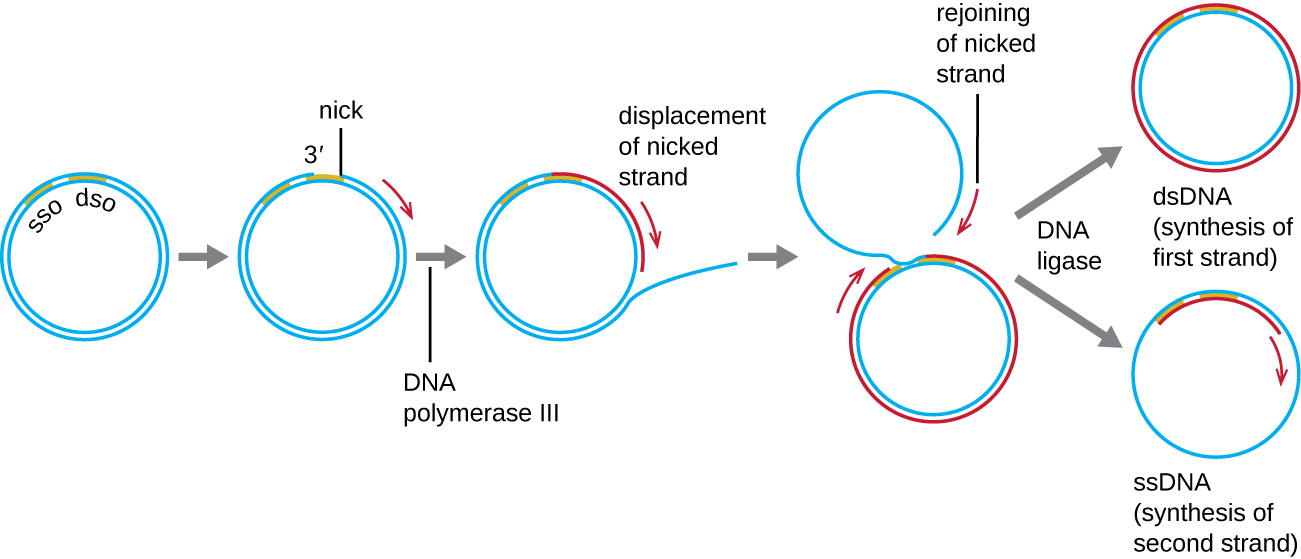
Mientras que muchos plásmidos bacterianos (ver Características únicas de las células procariotas) se replican por un proceso similar al utilizado para copiar el cromosoma bacteriano, otros plásmidos, varios bacteriófagos y algunos virus de eucariotas utilizan replicación en círculo rodante (Figura\(\PageIndex{7}\)). La naturaleza circular de los plásmidos y la circularización de algunos genomas virales en la infección lo hacen posible. La replicación del círculo rodante comienza con el corte enzimático de una cadena de la molécula circular bicatenaria en el sitio de origen bicatenario (dso). En bacterias, la ADN polimerasa III se une al grupo 3'-OH de la cadena mellada y comienza a replicar unidireccionalmente el ADN usando la hebra no mellada como molde, desplazando la hebra mellada a medida que lo hace. La finalización de la replicación del ADN en el sitio de la mella original da como resultado el desplazamiento completo de la cadena mellada, que luego puede recircularizarse en una molécula de ADN monocatenario. La ARN primasa luego sintetiza un cebador para iniciar la replicación del ADN en el sitio de origen monocatenario (sso) de la molécula de ADN monocatenario (ssADN), dando como resultado una molécula de ADN bicatenario (ADNds) idéntica a la otra molécula de ADN circular.
Ejercicio\(\PageIndex{4}\)
¿Hay una hebra rezagada en la replicación de círculo rodante? ¿Por qué o por qué no?
Conceptos clave y resumen
- El proceso de replicación del ADN es semiconservativo, lo que da como resultado dos moléculas de ADN, cada una con una hebra parental de ADN y una hebra recién sintetizada.
- En bacterias, el inicio de la replicación ocurre en el origen de la replicación, donde el ADN superenrollado es desenrollado por la ADN girasa, hecho monocatenario por helicasa y unido por proteína de unión monocatenaria para mantener su estado varado. La primasa sintetiza un cebador de ARN corto, proporcionando un grupo 3'-OH libre al que la ADN polimerasa III puede agregar nucleótidos de ADN.
- Durante la elongación, la cadena principal del ADN se sintetiza continuamente a partir de un único cebador. La hebra rezagada se sintetiza discontinuamente en fragmentos cortos de Okazaki, cada uno requiriendo su propio cebador. Los cebadores de ARN se eliminan y reemplazan con nucleótidos de ADN por ADN polimerasa I bacteriana, y la ADN ligasa sella los huecos entre estos fragmentos.
- La terminación de la replicación en bacterias implica la resolución de concatémeros circulares de ADN por topoisomerasa IV para liberar las dos copias del cromosoma circular.
- Los eucariotas suelen tener múltiples cromosomas lineales, cada uno con múltiples orígenes de replicación. En general, la replicación en eucariotas es similar a la de los procariotas.
- La naturaleza lineal de los cromosomas eucariotas requiere telómeros para proteger los genes cerca del final de los cromosomas. La telomerasa extiende los telómeros, impidiendo su degradación, en algunos tipos celulares.
- La replicación en círculo rodante es un tipo de síntesis rápida de ADN unidireccional de una molécula de ADN circular utilizada para la replicación de algunos plásmidos.


