14.5: Probando la Efectividad de los Antimicrobianos
- Page ID
- 54924
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Objetivos de aprendizaje
- Explicar el concepto de farmacorresistencia
- Describir cómo los microorganismos desarrollan o adquieren resistencia a los medicamentos
- Describir los diferentes mecanismos de resistencia a los medicamentos antimicrobianos
La resistencia a los antimicrobianos no es un fenómeno nuevo. En la naturaleza, los microbios evolucionan constantemente para superar los compuestos antimicrobianos producidos por otros microorganismos. El desarrollo humano de los antimicrobianos y su uso clínico generalizado simplemente ha proporcionado otra presión selectiva que promueve una mayor evolución. Varios factores importantes pueden acelerar la evolución de la farmacorresistencia. Estos incluyen el uso excesivo y el mal uso de antimicrobianos, el uso inadecuado de antimicrobianos, la dosificación subterapéutica y el incumplimiento del tratamiento recomendado por el paciente.
La exposición de un patógeno a un compuesto antimicrobiano puede seleccionar mutaciones cromosómicas que confieren resistencia, que pueden transferirse verticalmente a generaciones microbianas posteriores y eventualmente convertirse en predominantes en una población microbiana que se expone repetidamente al antimicrobiano. Alternativamente, muchos genes responsables de la resistencia a los fármacos se encuentran en plásmidos o en transposones que pueden transferirse fácilmente entre microbios a través de la transferencia génica horizontal (ver Cómo los procariotas asexuales logran la diversidad genética). Los transposones también tienen la capacidad de mover genes de resistencia entre plásmidos y cromosomas para promover aún más la propagación de la resistencia.
Mecanismos para la farmacorresistencia
Existen varios mecanismos comunes para la farmacorresistencia, los cuales se resumen en la Figura\(\PageIndex{1}\). Estos mecanismos incluyen la modificación enzimática del fármaco, la modificación de la diana antimicrobiana y la prevención de la penetración o acumulación del fármaco.
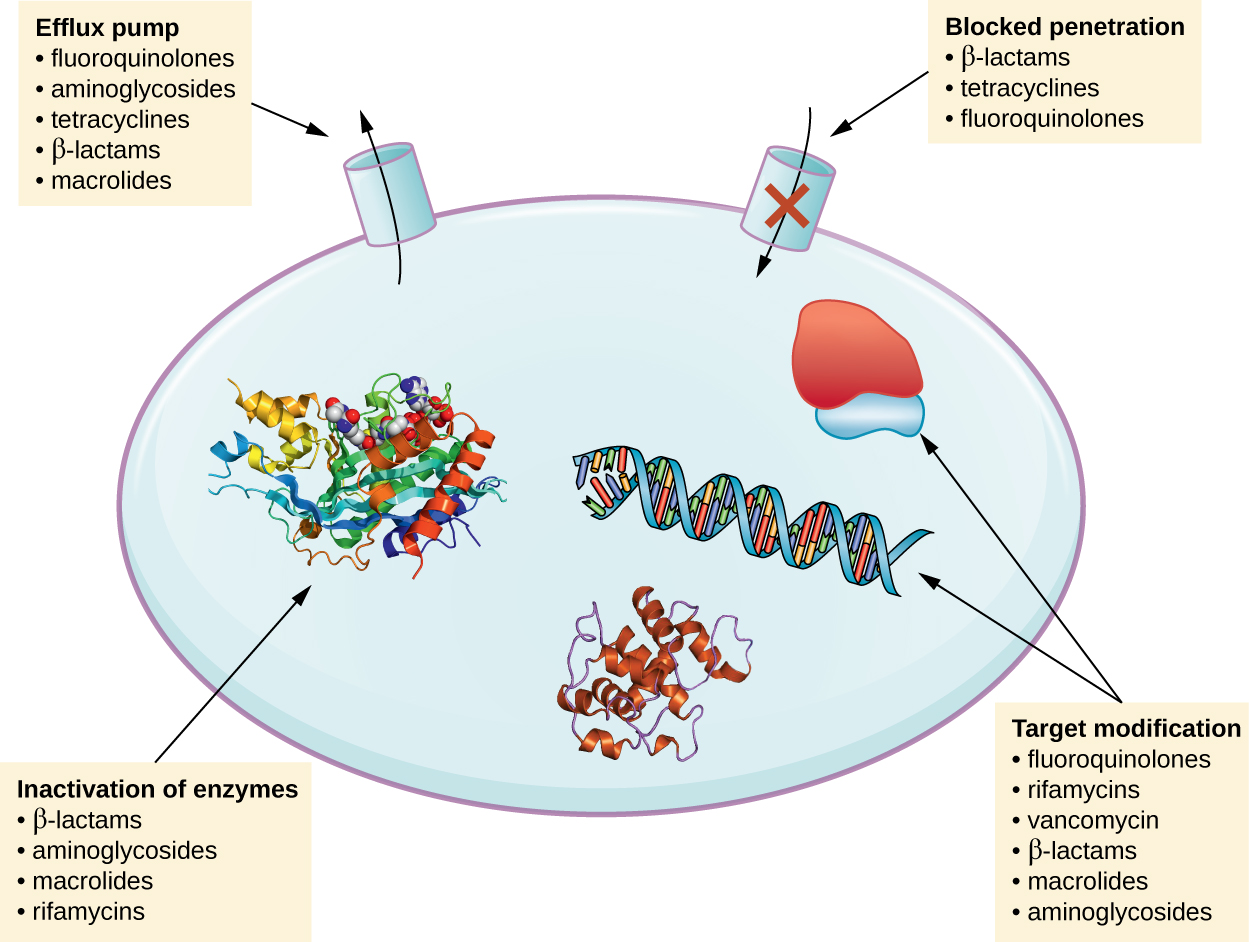
Modificación o inactivación de medicamentos
Los genes de resistencia pueden codificar enzimas que modifican químicamente un antimicrobiano, inactivándolo o destruyendo un antimicrobiano a través de la hidrólisis. La resistencia a muchos tipos de antimicrobianos ocurre a través de este mecanismo. Por ejemplo, la resistencia a los aminoglucósidos puede ocurrir a través de la transferencia enzimática de grupos químicos a la molécula del fármaco, afectando la unión del fármaco a su diana bacteriana. Para las β-lactamas, la resistencia bacteriana puede implicar la hidrólisis enzimática del enlace β-lactama dentro del anillo β-lactama de la molécula del fármaco. Una vez que se rompe el enlace β-lactama, el fármaco pierde su actividad antibacteriana. Este mecanismo de resistencia está mediado por β-lactamasas, que son el mecanismo más común de resistencia a β-lactama. La inactivación de la rifampina ocurre comúnmente a través de glicosilación, fosforilación o ribosilación de adenosina difosfato (ADP), y la resistencia a macrólidos y lincosamidas también puede ocurrir debido a la inactivación enzimática del fármaco o modificación.
Prevención de la Absorción o Efluencia Celular
Los microbios pueden desarrollar mecanismos de resistencia que implican inhibir la acumulación de un fármaco antimicrobiano, lo que luego impide que el fármaco alcance su diana celular. Esta estrategia es común entre los patógenos gramnegativos y puede implicar cambios en la composición lipídica de la membrana externa, la selectividad del canal porino y/o las concentraciones de los canales porinos. Por ejemplo, un mecanismo común de resistencia a carbapenem entre Pseudomonas aeruginosa es disminuir la cantidad de su porina OPRD, que es el portal primario de entrada para los carbapenémicos a través de la membrana externa de este patógeno. Adicionalmente, muchas bacterias patógenas grampositivas y gramnegativas producen bombas de eflujo que transportan activamente un fármaco antimicrobiano fuera de la célula y evitan la acumulación de fármaco a un nivel que sería antibacteriano. Por ejemplo, la resistencia a β-lactamas, tetraciclinas y fluoroquinolonas ocurre comúnmente a través del eflujo activo fuera de la célula, y es bastante común que una sola bomba de eflujo tenga la capacidad de translocar múltiples tipos de antimicrobianos.
Modificación de objetivos
Debido a que los medicamentos antimicrobianos tienen objetivos muy específicos, los cambios estructurales en esas dianas pueden prevenir la unión del fármaco, haciendo que el fármaco sea inefic A través de mutaciones espontáneas en los genes que codifican dianas de fármacos antibacterianos, las bacterias tienen una ventaja evolutiva que les permite desarrollar resistencia a los fármacos. Este mecanismo de desarrollo de resistencia es bastante común. Los cambios genéticos que impactan el sitio activo de las proteínas de unión a penicilina (PBP) pueden inhibir la unión de fármacos β-lactámicos y proporcionar resistencia a múltiples fármacos dentro de esta clase. Este mecanismo es muy común entre cepas de Streptococcus pneumoniae, que alteran sus propias PBP a través de mecanismos genéticos. En contraste, las cepas de Staphylococcus aureus desarrollan resistencia a la meticilina (SARM) a través de la adquisición de una nueva PBP de baja afinidad, en lugar de alterar estructuralmente sus PBP existentes. Esta nueva PBP de baja afinidad no solo proporciona resistencia a la meticilina sino que proporciona resistencia a prácticamente todos los fármacos β-lactámicos, con la excepción de las nuevas cefalosporinas de quinta generación diseñadas específicamente para matar el SARM. Otros ejemplos de esta estrategia de resistencia incluyen alteraciones en
- subunidades de ribosomas, que proporcionan resistencia a macrólidos, tetraciclinas y aminoglucósidos;
- estructura de lipopolisacárido (LPS), que proporciona resistencia a las polimixinas;
- ARN polimerasa, proporcionando resistencia a la rifampina;
- ADN girasa, proporcionando resistencia a las fluoroquinolonas;
- enzimas metabólicas, que proporcionan resistencia a los fármacos sulfa, sulfonas y trimetoprima; y
- cadenas peptídicas de la subunidad peptidoglicano, proporcionando resistencia a glicopéptidos
Sobreproducción objetivo o Bypass Enzimático
Cuando un fármaco antimicrobiano funciona como antimetabolito, dirigido a una enzima específica para inhibir su actividad, hay formas adicionales de que puede ocurrir resistencia microbiana. En primer lugar, el microbio puede sobreproducir la enzima diana de tal manera que existe una cantidad suficiente de enzima libre de antimicrobianos para llevar a cabo la reacción enzimática adecuada. En segundo lugar, la célula bacteriana puede desarrollar un bypass que elude la necesidad de la enzima diana funcional. Ambas estrategias se han encontrado como mecanismos de resistencia a las sulfonamidas. Se ha demostrado que la resistencia a la vancomicina entre S. aureus implica la disminución de la reticulación de las cadenas peptídicas en la pared celular bacteriana, lo que proporciona un aumento en las dianas para que la vancomicina se una en la pared celular externa. El aumento de la unión de vancomicina en la pared celular externa proporciona un bloqueo que evita que las moléculas de fármaco libres penetren hasta donde pueden bloquear la nueva síntesis de la pared celular.
Mímica de Target
Un mecanismo de resistencia recientemente descubierto llamado mimetismo diana implica la producción de proteínas que se unen y secuestran fármacos, evitando que los fármacos se unan a su diana. Por ejemplo, Mycobacterium tuberculosis produce una proteína con repeticiones regulares de pentapéptidos que parece imitar la estructura del ADN. Esta proteína se une a las fluoroquinolonas, secuestrándolas y evitando que se unan al ADN, proporcionando resistencia a M. tuberculosis a las fluoroquinolonas. Se ha encontrado que las proteínas que imitan el sitio A del ribosoma bacteriano también contribuyen a la resistencia a los aminoglucósidos. 1
Ejercicio\(\PageIndex{1}\)
Enumerar varios mecanismos para la farmacorresistencia.
Microbios Multiresistentes y Resistencia Cruzada
Desde una perspectiva clínica, nuestras mayores preocupaciones son los microbios multirresistentes (MDRs) y la resistencia cruzada. Los MDR se conocen coloquialmente como “superbacterias” y llevan uno o más mecanismos de resistencia, haciéndolos resistentes a múltiples antimicrobianos. En resistencia cruzada, un único mecanismo de resistencia confiere resistencia a múltiples fármacos antimicrobianos. Por ejemplo, tener una bomba de eflujo que pueda exportar múltiples medicamentos antimicrobianos es una forma común para que los microbios sean resistentes a múltiples fármacos mediante el uso de un solo mecanismo de resistencia. En los últimos años, han surgido varias superbacterias clínicamente importantes, y los CDC informan que las superbacterias son responsables de más de 2 millones de infecciones en Estados Unidos anualmente, resultando en al menos 23,000 muertes. 2 Varias de las superbacterias discutidas en las siguientes secciones han sido denominadas los patógenos ESKAPE. Este acrónimo hace referencia a los nombres de los patógenos (Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa y Enterobacter spp.) pero también es apropiado en que estos los patógenos son capaces de “escapar” de muchas formas convencionales de terapia antimicrobiana. Como tal, las infecciones por patógenos ESKAPE pueden ser difíciles de tratar y provocan un gran número de infecciones nosocomiales.
Staphylococcus aureus resistente a meticilina (MRSA)
La meticilina, una penicilina semisintética, fue diseñada para resistir la inactivación por β-lactamasas. Desafortunadamente, poco después de la introducción de la meticilina a la práctica clínica, aparecieron cepas de S. aureus resistentes a meticilina y comenzaron a propagarse. El mecanismo de resistencia, adquisición de una nueva PBP de baja afinidad, proporcionó a S. aureus resistencia a todas las β-lactamas disponibles. Las cepas de S. aureus resistente a meticilina (SARM) son patógenos oportunistas generalizados y una preocupación particular por infecciones de la piel y otras heridas, pero también pueden causar neumonía y septicemia. Aunque originalmente era un problema en entornos de salud (SARM adquirido en hospitales [HA-MRSA]), las infecciones por MRSA ahora también se adquieren a través del contacto con miembros contaminados del público en general, llamados SARM asociados a la comunidad (CA-MRSA). Aproximadamente un tercio de la población porta S. aureus como miembro de su microbiota nasal normal sin enfermedad, y alrededor del 6% de estas cepas son resistentes a la meticilina. 3 4
Ácido clavulánico: el pequeño ayudante de la penicilina
Con la introducción de la penicilina a principios de la década de 1940, y su posterior producción en masa, la sociedad comenzó a pensar en los antibióticos como curas milagrosas para una amplia gama de enfermedades infecciosas. Desafortunadamente, ya en 1945, la resistencia a la penicilina se documentó por primera vez y comenzó a propagarse. Más del 90% de los aislados clínicos actuales de S. aureus son resistentes a la penicilina. 5
Si bien desarrollar nuevos fármacos antimicrobianos es una solución a este problema, los científicos han explorado nuevos enfoques, entre ellos el desarrollo de compuestos que inactivan los mecanismos de resistencia. El desarrollo del ácido clavulánico representa un ejemplo temprano de esta estrategia. El ácido clavulánico es una molécula producida por la bacteria Streptococcus clavuligerus. Contiene un anillo β-lactama, lo que lo hace estructuralmente similar a la penicilina y otras β-lactamas, pero no muestra efectividad clínica cuando se administra por sí solo. En cambio, el ácido clavulánico se une irreversiblemente dentro del sitio activo de las β-lactamasas y evita que inactiven una penicilina coadministrada.
El ácido clavulánico se desarrolló por primera vez en la década de 1970 y se comercializó en masa en combinación con amoxicilina a partir de la década de 1980 bajo la marca Augmentin. Como suele ser el caso, pronto apareció resistencia a la combinación amoxicilina-ácido clavulánico. La resistencia suele ser el resultado de que las bacterias incrementen la producción de su β-lactamasa y abruman los efectos inhibitorios del ácido clavulánico, mutando su β-lactamasa para que ya no sea inhibida por el ácido clavulánico, o de adquirir una nueva β-lactamasa que no es inhibida por el ácido clavulánico. A pesar de las crecientes preocupaciones de resistencia, el ácido clavulánico y los inhibidores de β-lactamasa relacionados (sulbactam y tazobactam) representan una nueva estrategia importante: el desarrollo de compuestos que inhiben directamente las enzimas que confieren resistencia antimicrobiana.
Enterococos y Staphylococcus aureus resistentes a la vancomicina
La vancomicina solo es efectiva contra organismos grampositivos, y se usa para tratar infecciones de heridas, infecciones sépticas, endocarditis y meningitis causadas por patógenos resistentes a otros antibióticos. Se considera una de las últimas líneas de defensa contra este tipo de infecciones resistentes, incluyendo MRSA. Con el aumento de la resistencia a los antibióticos en las décadas de 1970 y 1980, el uso de vancomicina aumentó, y no es sorprendente que vimos la aparición y propagación de enterococos resistentes a vancomicina (VRE), S. aureus resistente a vancomicina (VRSA) y S. aureus intermedio a vancomicina (VISA). El mecanismo de resistencia a la vancomicina entre los enterococos es la modificación de la diana que implica un cambio estructural en el componente peptídico de las subunidades de peptidoglicano, evitando que la vancomicina se una. Estas cepas se propagan típicamente entre pacientes en entornos clínicos por contacto con trabajadores de la salud y superficies contaminadas y equipos médicos.
Las cepas VISA y VRSA difieren entre sí en el mecanismo de resistencia y el grado de resistencia que confiere cada mecanismo. Las cepas VISA presentan resistencia intermedia, con una concentración inhibitoria mínima (MIC) de 4—8 μg/mL, y el mecanismo implica un aumento en las dianas de vancomicina. Las cepas VISA disminuyen la reticulación de las cadenas peptídicas en la pared celular, proporcionando un aumento en las dianas de vancomicina que atrapan la vancomicina en la pared celular externa. En contraste, las cepas de VRSA adquieren resistencia a la vancomicina a través de la transferencia horizontal de genes de resistencia del VRE, una oportunidad brindada en individuos coinfectados tanto con VRE como con MRSA. Los VRSA presentan un mayor nivel de resistencia, con MICs de 16 μg/mL o superiores. 6 En el caso de los tres tipos de bacterias resistentes a la vancomicina, es necesaria una rápida identificación clínica por lo que se pueden implementar procedimientos adecuados para limitar la propagación. Las oxazolidinonas como linezolid son útiles para el tratamiento de estos patógenos oportunistas resistentes a la vancomicina, así como MRSA.
Patógenos Gram-negativos productores de β-lactamasa de espectro extendido
Los patógenos gramnegativos que producen β-lactamasas de espectro extendido (ESBL) muestran resistencia mucho más allá de las penicilinas. El espectro de β-lactamas inactivadas por ESBLs proporciona resistencia a todas las penicilinas, cefalosporinas, monobactamas y las combinaciones β-lactamasa-inhibidor, pero no a los carbapenémicos. Una preocupación aún mayor es que los genes que codifican las ESBLs generalmente se encuentran en plásmidos móviles que también contienen genes para resistencia a otras clases de fármacos (por ejemplo, fluoroquinolonas, aminoglucósidos, tetraciclinas), y pueden propagarse fácilmente a otras bacterias por transferencia génica horizontal. Estas bacterias multirresistentes son miembros de la microbiota intestinal de algunos individuos, pero también son causas importantes de infecciones oportunistas en pacientes hospitalizados, de quienes pueden propagarse a otras personas.
Bacterias Gram-negativas Resistentes a Carbapeem
La aparición de Enterobacteriaceae resistentes a carbapenémicos (CRE) y resistencia a carbapenem entre otras bacterias gramnegativas (p. ej., P. aeruginosa, Acinetobacter baumannii, Stenotrophomonas maltophila) es una preocupación creciente en el cuidado de la salud. Estos patógenos desarrollan resistencia a los carbapenémicos a través de una variedad de mecanismos, incluyendo la producción de carbapenemasas (β-lactamasas de amplio espectro que inactivan todas las β-lactamas, incluyendo carbapenémicos), salida activa de carbapenémicos fuera de la célula y/o prevención de la entrada de carbapenémicos a través de canales porínicos. Similar a las preocupaciones con los ESBLs, los patógenos gramnegativos resistentes a carbapenémicos suelen ser resistentes a múltiples clases de antibacterianos, y algunos incluso han desarrollado panresistencia (resistencia a todos los antibacterianos disponibles). Las infecciones con patógenos gramnegativos resistentes a carbapenémicos ocurren comúnmente en entornos de salud a través de la interacción con individuos contaminados o dispositivos médicos, o como resultado de cirugía.
Mycobacterium tuberculosis multirresistente
La aparición de Mycobacterium tuberculosis multirresistente (MDR-TB) y Mycobacterium tuberculosis extensamente resistente a fármacos (XDR-TB) también es de gran preocupación mundial. Las cepas MDR-TB son resistentes tanto a la rifampina como a la isoniazida, la combinación de fármacos prescrita típicamente para el tratamiento de la tuberculosis. Las cepas XDR-TB son adicionalmente resistentes a cualquier fluoroquinolona y al menos a uno de otros tres fármacos (amikacina, kanamicina o capreomicina) utilizados como segunda línea de tratamiento, dejando a estos pacientes muy pocas opciones de tratamiento. Ambos tipos de patógenos son particularmente problemáticos en personas inmunodeprimidas, incluidas las que padecen infección por VIH. El desarrollo de resistencia en estas cepas a menudo resulta del uso incorrecto de antimicrobianos para el tratamiento tuberculoso, seleccionando resistencia.
Ejercicio\(\PageIndex{2}\)
¿Cómo conduce la resistencia a los medicamentos a las superbacterias?
Para obtener más información sobre las 18 principales amenazas resistentes a los medicamentos para Estados Unidos, visite el sitio web de los CDC.
Agricultura en fábrica y resistencia a los medicamentos
Aunque la cría de animales ha sido durante mucho tiempo una parte importante de la agricultura en Estados Unidos, el aumento de las operaciones concentradas de alimentación animal (CAFO) desde la década de 1950 ha provocado algunos nuevos problemas ambientales, incluida la contaminación del agua y el aire con desechos biológicos, y cuestiones éticas con respecto a los derechos de los animales. también están asociados con el cultivo de animales de esta manera. Adicionalmente, el incremento de CAFO implica el uso extensivo de medicamentos antimicrobianos en la cría de ganado. Los antimicrobianos se utilizan para prevenir el desarrollo de enfermedades infecciosas en las zonas cercanas de las CAFO; sin embargo, la mayoría de los antimicrobianos utilizados en la agricultura industrial son para promover el crecimiento, es decir, para cultivar animales más grandes.
El mecanismo subyacente a este crecimiento mejorado sigue sin estar claro. Estos antibióticos pueden no ser necesariamente los mismos que los que se usan clínicamente para los humanos, pero están estructuralmente relacionados con los medicamentos que se usan para los humanos. Como resultado, el uso de medicamentos antimicrobianos en animales puede seleccionar la resistencia antimicrobiana, y estas bacterias resistentes se vuelven resistentes a los medicamentos que se usan típicamente en humanos. Por ejemplo, el uso de tilosina en animales parece seleccionar bacterias también resistentes cruzadamente a otros macrólidos, incluida la eritromicina, comúnmente utilizada en humanos.
Las concentraciones de las cepas bacterianas farmacorresistentes generadas por las CAFO aumentan en el agua y el suelo que rodean estas granjas. Si no son directamente patógenas en humanos, estas bacterias resistentes pueden servir como reservorio de elementos genéticos móviles que luego pueden pasar genes de resistencia a patógenos humanos. Afortunadamente, el proceso de cocción normalmente inactiva cualquier antimicrobiano que quede en la carne, por lo que los humanos normalmente no están ingiriendo directamente estos medicamentos. Sin embargo, muchas personas están pidiendo un uso más juicioso de estos medicamentos, tal vez cobrando tarifas a los usuarios de los agricultores para reducir el uso indiscriminado. De hecho, en 2012, la FDA publicó pautas para los agricultores que voluntariamente eliminaron gradualmente el uso de medicamentos antimicrobianos excepto bajo supervisión veterinaria y cuando sea necesario para garantizar la salud animal. Aunque seguir las pautas es voluntario en este momento, la FDA recomienda lo que llama el uso “juicioso” de medicamentos antimicrobianos en animales productores de alimentos en un esfuerzo por disminuir la resistencia a los antimicrobianos.
Enfoque Clínico: Parte 3
Desafortunadamente, la infección del tracto urinario de Marisa no se resolvió con el tratamiento con ciprofloxacino. Las pruebas de laboratorio mostraron que su infección fue causada por una cepa de Klebsiella pneumoniae con resistencia antimicrobiana significativa. El perfil de resistencia de este K. pneumoniae incluyó resistencia a la clase carbapenem de antibacterianos, un grupo de β-lactamas que normalmente se reserva para el tratamiento de bacterias altamente resistentes. K. pneumoniae es una varilla oportunista, encapsulada, gramnegativa que puede ser miembro de la microbiota normal del tracto intestinal, pero también puede causar una serie de enfermedades, incluyendo neumonía y UTI.
Se realizaron pruebas específicas de laboratorio en busca de producción de carbapenemasa en muestras de Marisa y dieron positivo. Con base en este resultado, en combinación con su historial de salud, se sospechó la producción de una carbapenemasa conocida como metalo-β-lactamasa de Nueva Delhi (NDM). Aunque no se conoce completamente el origen de la carbapenemasa NDM, muchos pacientes infectados con cepas que contienen NDM tienen antecedentes de viaje que involucran hospitalizaciones en India o países circundantes.
Ejercicio\(\PageIndex{1}\)
¿Cómo determinarían los médicos qué tipos de medicamentos antimicrobianos deben administrarse?
Conceptos clave y resumen
- La resistencia a los antimicrobianos va en aumento y es el resultado de la selección de cepas farmacorresistentes en entornos clínicos, el uso excesivo y uso indebido de antibacterianos, el uso de dosis subterapéuticas de fármacos antibacterianos y el mal cumplimiento por parte del paciente de las terapias con medicamentos antibacterianos.
- Los genes farmacorresistentes a menudo se transportan en plásmidos o en transposones que pueden sufrir transferencia vertical fácilmente y entre microbios a través de la transferencia génica horizontal.
- Los modos comunes de resistencia a los medicamentos antimicrobianos incluyen la modificación o inactivación de fármacos, la prevención de la captación o salida celular, la modificación de la diana, la sobreproducción de la diana o el bypass enzimático, y la mimetización
- Están surgiendo cepas microbianas problemáticas que muestran una amplia resistencia antimicrobiana; muchas de estas cepas pueden residir como miembros de la microbiota normal en individuos pero también pueden causar infección oportunista. La transmisión de muchas de estas cepas microbianas altamente resistentes a menudo ocurre en entornos clínicos, pero también puede ser adquirida en la comunidad.
Notas al pie
- 1 D.H. Fong, A.M. Berghuis. “Promiscuidad de sustrato de una enzima de resistencia a antibióticos aminoglucósidos vía mimetismo diana”. EMBO Diario 21 núm. 10 (2002) :2323—2331.
- 2 Centros de Control y Prevención de Enfermedades. “Resistencia Antibiótico/Antimicrobiana”. http://www.cdc.gov/drugresistance/index.html. Consultado el 2 de junio de 2016.
- 3 A.S. Kalokhe et al. “Susceptibilidad a los medicamentos de tuberculosis multirresistente y pruebas de diagnóstico molecular: una revisión de la literatura. Revista Americana de las Ciencias Médicas 345 núm. 2 (2013) :143—148.
- 4 Centros de Control y Prevención de Enfermedades. “Staphylococcus aureus resistente a meticilina (MRSA): Información General sobre SARM en la Comunidad”. http://www.cdc.gov/mrsa/community/index.html. Accedido junio 2, 2016
- 5 F.D. Lowy. “Resistencia Antimicrobiana: El Ejemplo de Staphylococcus aureus”. Revista de Investigación Clínica 111 núm. 9 (2003) :1265—1273.
- 6 Centros de Control y Prevención de Enfermedades. “Infecciones Asociadas a la Atención de la Salud (HIA): Información General sobre VISA/VRSA.” www.CDC.gov/hai/organisms/vis... visa_vrsa.html. Consultado el 2 de junio de 2016.


