3.3: Diferentes ensayos genético-toxicológicos
- Page ID
- 75376
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)- 1: Conocer diferentes tipos de ensayos genético-toxicológicos
- 2: Conocer cómo se utilizan diferentes ensayos genético-toxicológicos en toxicología cuando las células están expuestas a mutágenos.
El objetivo del ensayo de toxicología genética es determinar si algún químico o mutágeno producirá algún efecto adverso sobre el material genético o puede causar diferentes enfermedades, incluido el cáncer. Los ensayos se pueden realizar usando células bacterianas, de levadura o de mamífero. Se puede controlar temprano y salvar organismos vulnerables de químicos genotóxicos mediante la preformación de un ensayo de toxicología genética.
Los siguientes diferentes tipos de ensayos de toxicología genética se utilizan hoy en día:
- Ensayo de mutación inversa bacteriana (ensayo Ames)
- Ensayo de mutación genética
- PCR específica de alelo
- Secuenciación Dideoxi de Sanger
- Estudio de aberración cromosómica
- Ensayo de micronúcleos
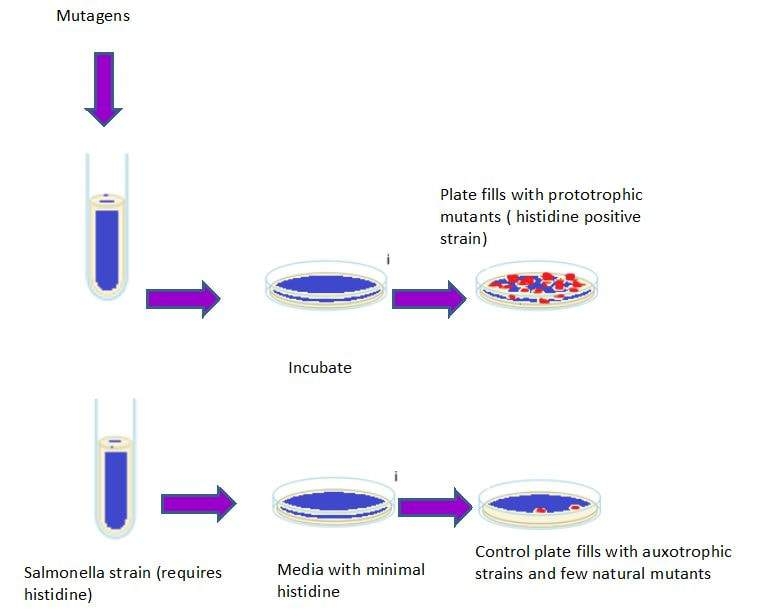
3.1: Ensayo de mutación inversa bacteriana (ensayo Ames)
Este ensayo fue descubierto por Bruce Ames en 1970. Este ensayo es ampliamente utilizado para probar la mutación génica. La técnica utiliza varias cepas de la bacteria Salmonella typhimurium que portan mutaciones en genes involucrados en la síntesis de histidina. Estas cepas son mutantes auxotróficas y requieren histidina para su crecimiento y no pueden producirla. Este ensayo examina la capacidad del químico o mutágeno para crear mutaciones o un estado “prototrófico” de cepas, cuando las cepas pueden crecer en un medio libre de histidina.
3.2: Ensayo de Mutación Genética
A continuación se presentan los diferentes ensayos moleculares para estudiar variantes de nucleótidos o alternancia de material genético causado por mutágenos:
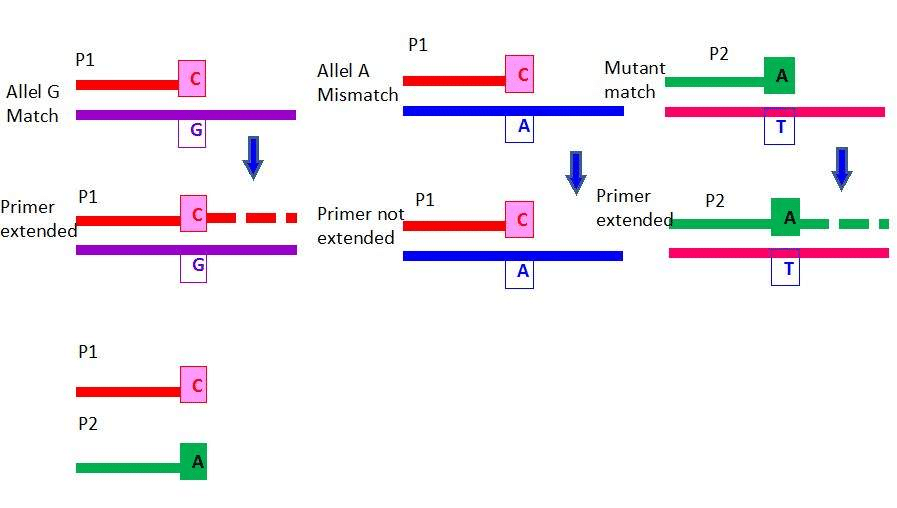
PCR específica de alelo
El polimorfismo de nucleótido único (SNP) resultante de la mutación por sustitución de bases se puede analizar mediante este método. En esta PCR en tiempo real, se agregan sondas informadoras fluorescentes a la mezcla de reacción y se selecciona una sonda informadora fluorescente para tipo silvestre y se usa otra sonda fluorescente para mutante. Los cebadores de PCR con sonda fluorescente coincidirán o desaparearán uno de los alelos en el extremo 3' del cebador. La ADN polimerasa extiende las sondas de manera complementaria y liberando las moléculas fluorescentes informadoras para su detección. Los ciclos de PCR con las sondas informadoras muestran las señales amplificadas y permiten la medición precisa de uno o ambos alelos de interés. De manera similar, el extremo 3' del cebador específico de mutante se extiende solo en presencia de ADN con esa mutación.
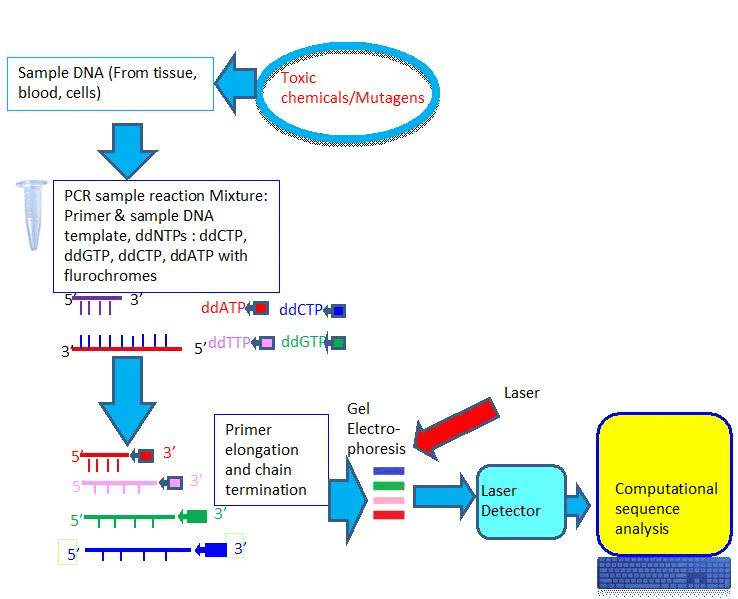
Secuenciación Dideoxi de Sanger
El objetivo de este método es detectar mutaciones desconocidas incluyendo variantes de un solo nucleótido (SNV) y pequeñas duplicaciones, inserciones, deleciones e indeles de interés causados por mutágenos. En este método, los cebadores de secuenciación se hibridaron con el producto de PCR y se extienden utilizando los cuatro desoxinucleótidos (dNTP), una mezcla de didesoxinucleótidos marcados fluorescentemente (ddNTPs) y ADN polimerasa. Cuatro ddNTPs están marcados con un colorante fluorescente diferente. La incorporación aleatoria de los ddNTPs marcados se muestra en la terminación de cadenas en cada ubicación a lo largo de la secuencia. La electroforesis en gel separa las hebras por tamaño. La espectroscopia de fluorescencia midió los nucleótidos de terminación.
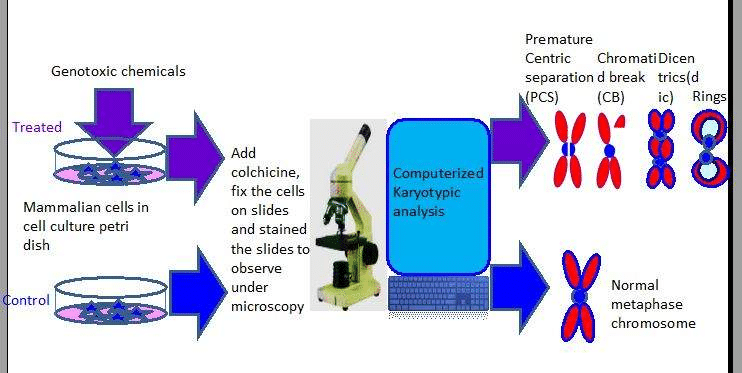
3.3: Estudio de aberración cromosómica
Se realizan ensayos citogenéticos de células de mamíferos para detectar diferentes tipos de aberraciones cromosómicas estructurales y numéricas causadas por químicos genotóxicos. Los efectos clastogénicos o aneugénicos de los químicos genotóxicos resultarán en un incremento en la frecuencia de reordenamientos complejos estructurales (separación centrada prematura, roturas cromosómicas, cromosomas dicéntricos, anillos) (Figura 4) o aberraciones numéricas del material genético en células de mamíferos.
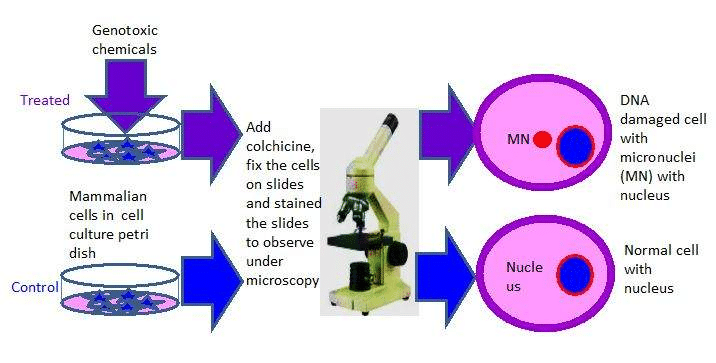
3.4: Ensayo de micronúcleos
El ensayo de micronúcleos se utiliza como herramienta para evaluar el daño genético causado por sustancias químicas genotóxicas. El número de micronúcleos (Figura 5) generados se relaciona directamente con la cantidad de daño del ADN en las células.
Tema 3: Puntos Clave
En esta sección, exploramos los siguientes puntos principales:
- 1: Definición de ensayo de toxicología genética
- 2: Diferentes tipos de ensayos genético-toxicológicos.
- 3: Cómo se realizan los ensayos de mutación genética mediante PCR específica de alelo y técnicas de secuenciación dideoxi de Sanger.
- 4: ¿Cuáles son los diferentes tipos de aberraciones cromosómicas observadas bajo el microscopio mediante el estudio de aberración cromosómica?
- 5: Cambios de micronúcleos observados al microscopio mediante el ensayo de Micronúcleos.
1. Se realizan ensayos citogenéticos de células de mamíferos para detectar diferentes tipos de aberraciones cromosómicas estructurales y numéricas causadas por una sustancia química genotóxica. Las aberraciones cromosómicas estructurales son:
separación centrada prematura
anillo
roturas cromosómicas
cromosomas dicéntricos
Todo lo anterior
- Contestar
-
Todo lo anterior
2. En la PCR específica de alelo, se agregan sondas informadoras fluorescentes a la mezcla de reacción y se selecciona una sonda informadora fluorescente para tipo silvestre y se usa otra sonda fluorescente para mutante.
Cierto
Falso
- Contestar
-
Cierto
3. ¿Qué instrumento se utiliza para medir los nucleótidos terminadores en la Secuenciación Dideoxi de Sanger?
Espectroscopia de fluorescencia
Espectrofotómetro
Microscopía de fluorescencia
Ninguna de las anteriores
- Contestar
-
Espectroscopia de fluorescencia
4. La técnica de Ames utiliza varias cepas de la bacteria Salmonella typhimurium que portan mutaciones en genes involucrados en:
Síntesis de Arginina.
Síntesis de histidina.
Síntesis de Lisina.
Ninguna de las anteriores.
- Contestar
-
Síntesis de histidina.


