2.3: Metabolismo de ADN y ARN
- Page ID
- 75509
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)Después de completar esta lección, usted será capaz de:
- Comprender el mecanismo de replicación del ADN.
- Comprender el proceso de expresión génica.
- Enumerar los principales tipos de mutación del ADN y mecanismo de reparación del ADN.
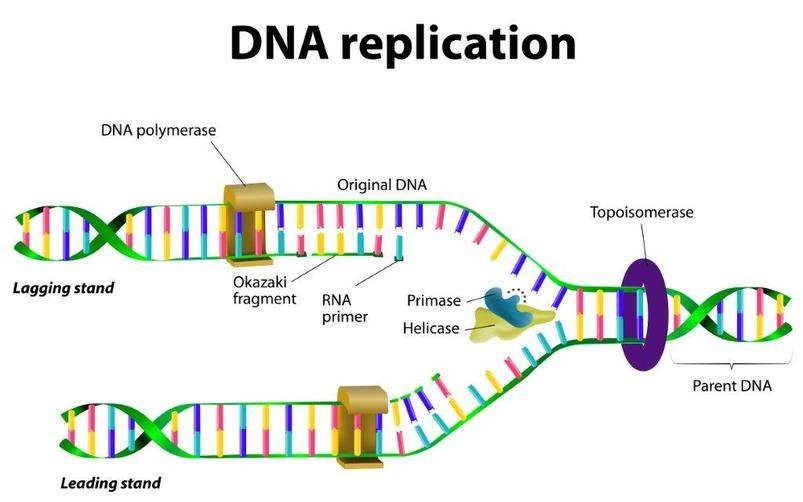
Replicación de ADN
El proceso de replicación del ADN consiste en desenrollar el ADN bicatenario, copiar cada cadena de ADN y luego separar las dos, nuevas, copias bicatenarias. El proceso inicia en un origen de replicación (ori), una secuencia de ácidos nucleicos donde puede comenzar la replicación. Hay alrededor de 100 mil orígenes de replicación en cada célula humana. Esto significa que la replicación del ADN puede comenzar simultáneamente en diferentes posiciones al mismo tiempo. La horquilla de replicación es el punto donde se separan dos cadenas de ADN, una denominada cadena principal y la otra hebra retrasada, y se produce la copia del ADN. La estructura de ADN de doble hélice enrollada es inicialmente desenrollada por la enzima ADN helicasa rompiendo los enlaces de hidrógeno entre los ácidos nucleicos complementarios. Las proteínas de unión monocatenarias se unen a las nuevas cadenas de ADN para mantenerlas separadas.
Una enzima denominada primasa produce entonces una cadena corta de ARN que sirve como cebador para el resto del proceso. La enzima ADN polimerasa replica cada cadena de ADN en la dirección 5' a 3' agregando el nucleótido trifosfato correcto y coincidente al extremo 3′-hidroxilo de la cadena del cebador. A medida que se agrega cada nuevo ácido nucleico, se forma un nuevo enlace fosfodiéster, utilizando la energía contenida en el grupo difosfato restante.
Transcripción de ARN
La transcripción de ARN es el proceso mediante el cual un segmento particular de ADN se transcribe en una secuencia de ARN equivalente.
- mRNA: Para genes que codifican para una proteína.
- ARNt: Para un ARN de transferencia.
- ARNr: Para el ensamblaje de un ribosoma.
- MiARN (micro ARN): Que se une al ARNm e inhibe su traducción.
- ARNip (ARN interferente pequeño): Que se une al ARNm y ayuda en su degradación.
- ARNnp (ARN nuclear pequeño): Que participa en el procesamiento del ARN como parte de los empalmeosomas.
- SNORNa (ARN nucleolar pequeño): Que participan en el procesamiento de ARN nucleolar.
La transcripción comienza con la unión de la enzima ARN polimerasa a una secuencia promotora en el ADN, región reguladora que dicta dónde debe comenzar la transcripción.
El ADN se transcribe de 3' a 5' y se produce solo en una de las cadenas de ADN, la cadena molde.
Al igual que en la replicación del ADN, la energía para la formación del enlace fosfodiéster se deriva de la hidrólisis de los dos enlaces fosfato terminales del nucleósido trifosfato.
Múltiples ARN polimerasas pueden transcribirse en una única secuencia génica de ADN, permitiendo una rápida producción del producto de ARN.
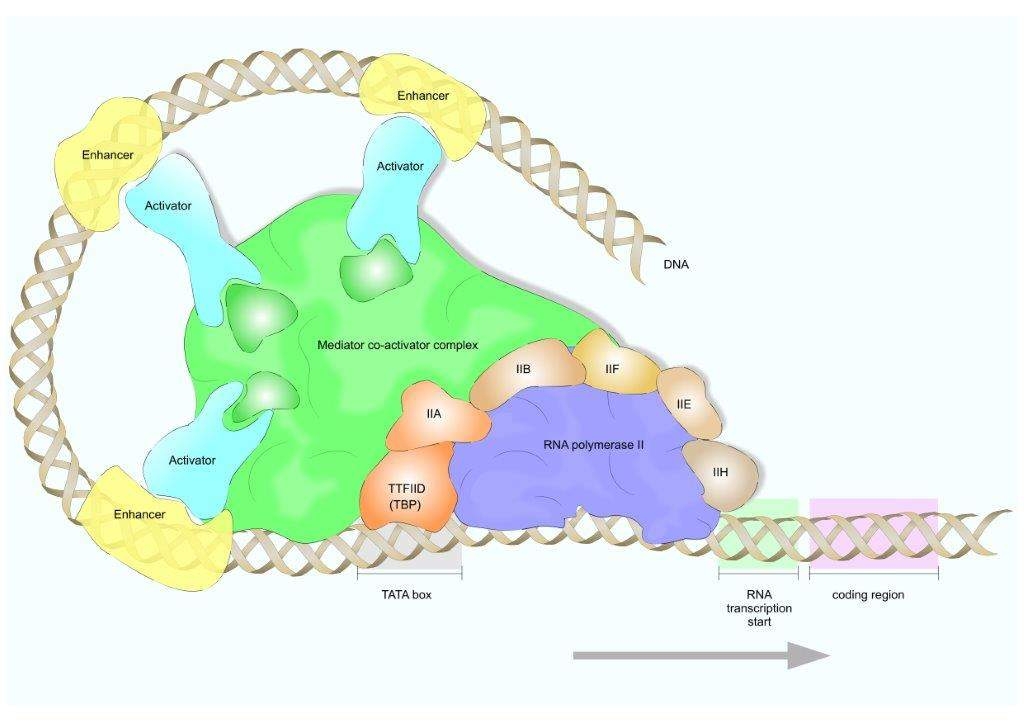
Potenciador: Esta es una región corta de ADN que puede unirse por factores de transcripción para aumentar o facilitar la transcripción de un gen en particular. Pueden localizarse lejos del gen, aguas arriba o aguas abajo del sitio de inicio.
Factores de Transcripción (TFs): Estos incluyen un amplio número de proteínas, excluyendo la ARN polimerasa, que promueve (como activador), o bloquea (como represor) el reclutamiento de la ARN polimerasa al ADN. Los TFs se unen a regiones promotoras del ADN.
Región Promotora: Estas son secuencias de ADN específicas, generalmente localizadas aguas arriba y cerca de los sitios de inicio de la transcripción de los genes y sirven como sitio de unión para proteínas llamadas factores de transcripción que reclutan ARN polemerasa. Ejemplo: TATA bos, isla CpG.
Figura\(\PageIndex{1}\): Maquinaria de transcripción de ARN
Procesamiento de ARN
Los transcritos de ARN recién sintetizados se procesan antes de su uso en la célula como ARN maduro.
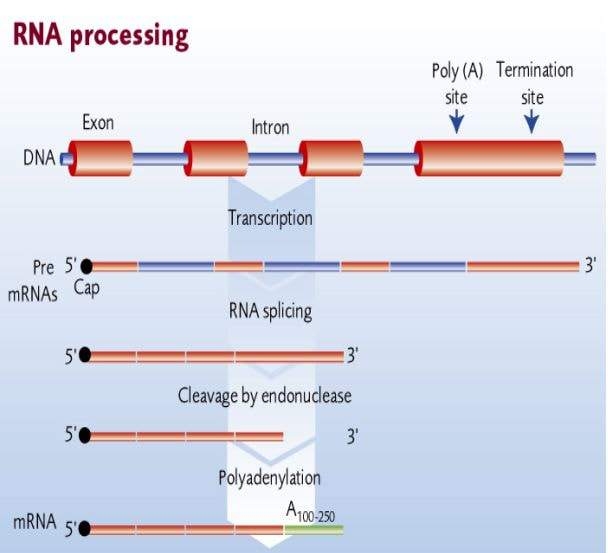
Se agrega un ácido nucleico de 7-metil guanosina al extremo 5' (conocido como capuchón 5') del pre-ARNm a medida que emerge de la ARN polimerasa II (Pol II). La caperuza protege al ARN de ser degradado por las enzimas y sirve como punto de ensamblaje para que las proteínas comiencen la traducción a proteína.
Eliminación de intrones presentes en el pre-ARNm y empalme de los exones restantes, en un proceso llamado empalme de ARN. Las series continuas de bases de ADN que codifican una proteína se ven interrumpidas por secuencias de bases que no se traducen. Las secuencias traducidas se denominan exones (secuencias expresadas) y las secuencias no traducidas como intrones (secuencias intervinientes). Esto completa la molécula de ARNm, que ahora está lista para su exportación al citosol. (El resto de la transcripción se degrada y la ARN polimerasa abandona el ADN).
3.4 Traducción de Proteínas
La síntesis de proteínas requiere la interacción de ARNm, ARNt, varias proteínas accesorias, llamadas factor de iniciación (IF) y factor de elongación (EF), y ribosomas.
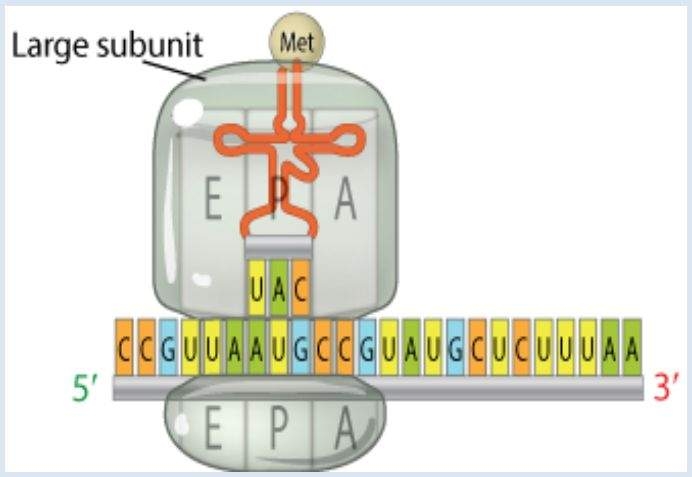 La subunidad ribosómica grande se une a la subunidad ribosómica pequeña para completar el complejo de iniciación. La molécula de ARNt se une a un aminoácido en la parte superior de la estructura. En la base si el ARNt existe la secuencia de tres bases conocida como el anticodón. Este anticodón se une, a través del apareamiento de bases, a un codón de tres bases sobre ARNm Es esta interacción entre el ARNm y el aminoácido-ARNt la que proporciona el alto grado de fidelidad observado en la transferencia de información genética del ADN a las proteínas.
La subunidad ribosómica grande se une a la subunidad ribosómica pequeña para completar el complejo de iniciación. La molécula de ARNt se une a un aminoácido en la parte superior de la estructura. En la base si el ARNt existe la secuencia de tres bases conocida como el anticodón. Este anticodón se une, a través del apareamiento de bases, a un codón de tres bases sobre ARNm Es esta interacción entre el ARNm y el aminoácido-ARNt la que proporciona el alto grado de fidelidad observado en la transferencia de información genética del ADN a las proteínas.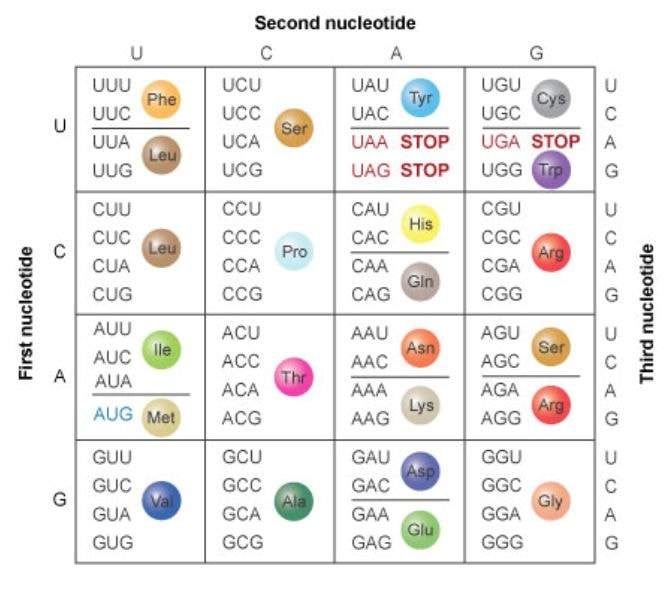 El Código Triplete (Código Genético)
El Código Triplete (Código Genético)
Una secuencia de tres bases en el ADN identifica cada uno de los 20 aminoácidos que se van a incorporar a la proteína recién sintetizada. Esta información se incorpora al ARNm que se sintetiza usando ADN como molde. Considerando que se requieren tres bases como mínimo, y tenemos 4 nucleótidos (43 = 64 palabras clave son posibles). Esto es más de los 20 aminoácidos. De hecho, se utilizan muchos tripletes para definir un mismo aminoácido. Además, algunas secuencias tripletes “extra” se utilizan como codones de parada para terminar la síntesis de proteínas. AUG se utiliza como codón de inicio para el aminoácido N-terminal en eucariotas.
Usando las mismas reglas de unión que las cadenas dobles de ADN (por ejemplo, un ARNt que se une al codón de ARNm de inicio AUG tiene una secuencia anticodón de UAC), asegurando el orden específico de AA requerido para la producción adecuada de la proteína.
Figura\(\PageIndex{4}\): Código genético - © 2014 Nature Education
El ARN juega tres papeles distintos e importantes:
- mRNA: El intermediario entre gen y proteína — proporciona el mensaje.
- ARNt: La clave o adaptador: lee el código genético, aporta aminoácidos a la cadena polipeptídica en crecimiento
- ARNr: En ribosoma, proporciona un andamio para la síntesis de proteínas, cataliza la formación de enlaces peptídicos
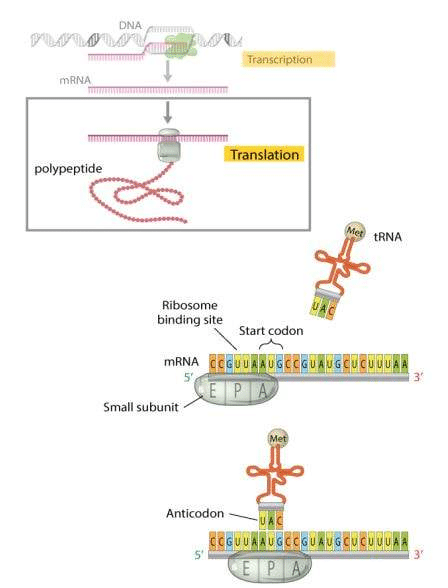
Las proteínas accesorias IF y EF cumplen una serie de funciones, incluyendo permitir la unión de la molécula de ARNm al ribosoma, movimiento del ARNm a lo largo del ribosoma hasta el punto de inicio de la síntesis, acoplamiento del ARNt-aminoácido y movimiento del ARNm y cadena peptídica en crecimiento, así como garantía de precisión.
La biosíntesis de proteínas, se puede dividir en tres fases: iniciación, elongación y terminación.
Iniciación
El inicio de la síntesis de proteínas comienza cuando el factor iniciador de proteínas IF-3 se une a la subunidad pequeña del ribosoma y provoca su disociación. La subunidad ribosómica pequeña se une entonces al lado 5' del ARNm que lleva información en un código triplete del ADN. Luego, la subunidad pequeña se traslada donde se encuentra con la subunidad ribosómica grande, otros factores iniciadores de proteínas y ARNt iniciador. El ARNt se une a la metionina en un sitio en el ribosoma conocido como el sitio P.
Alargamiento
En la fase de elongación de la síntesis de proteínas, un aminoacil-ARNt específico, dirigido por interacciones de enlaces de hidrógeno entre la región anticodón del aminoacil-ARNt y la región de codón del ARNm, se agrega a un sitio distinto del sitio P, el sitio A. Los sitios A y P están muy próximos, permitiendo la formación de enlaces peptídicos entre los aminoácidos. El dipéptido unido al ARNt recién sintetizado luego se mueve del sitio A al sitio P. Después de una translocación del ribosoma en la dirección 5' - 3' a lo largo del ARNm se produce para exponer un nuevo codón. Luego, otra identidad de aminoácido-ARNt y se une al ARNm en el sitio A y se inicia nuevamente la reacción de peptidil-transferasa. A medida que la cadena polipeptídica crece a través de ciclos posteriores de incorporación de residuos de aminoácidos, emerge del ribosoma y experimenta plegamiento en sus conformaciones nativas secundarias y terciarias.
Terminación
En la etapa de terminación, la síntesis del enlace peptídico cesa cuando se alcanza un codón de terminación en el ARNm. Este sitio de terminación no se unirá a aminoacil-ARNt y se detiene la síntesis de péptidos. Los factores de liberación permiten que la proteína recién sintetizada se disocie del ribosoma.
3.5 Expresión génica
La expresión génica es un proceso de dos etapas en el que el ADN se convierte en una proteína.
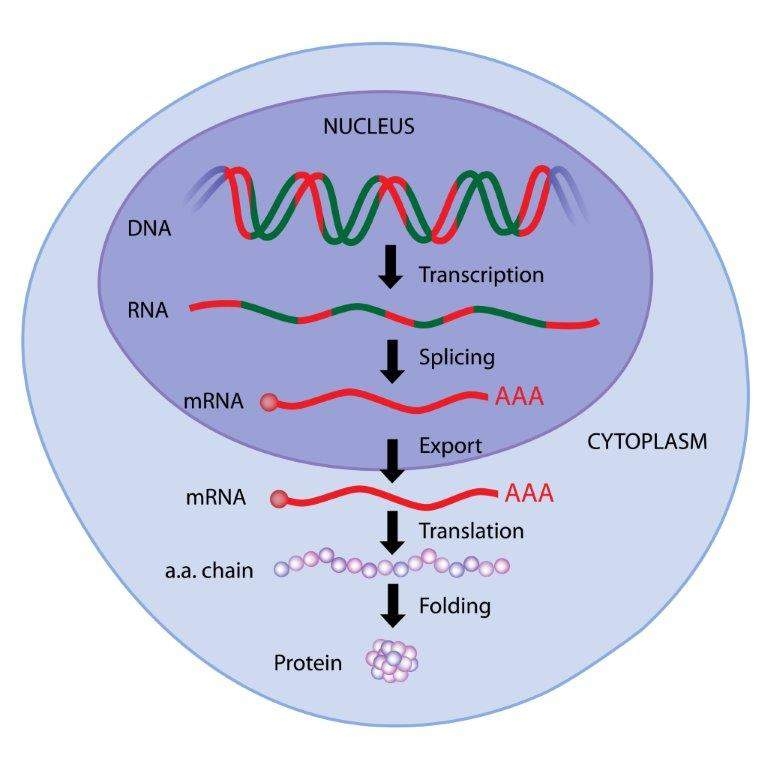 Paso 1: El primer paso es la transcripción de ADN a ARN. En este paso, la información de la copia de archivo del ADN se imprime en el ARNm. La estructura del ARN es un poco diferente, contiene ribosa en lugar de desoxirribosa, y las cuatro bases que se unen a él son citosina (C), guanina (G), adenina (A) y uracilo (U). Durante la transcripción, el ADN se despliega y el ARNm se crea emparejando bases de ARNm con las bases del ADN. En este proceso, C en el ADN se traduce en G, G a C, A a U y T a A. Después de transcribir el ARNm se transporta al ribosoma.
Paso 1: El primer paso es la transcripción de ADN a ARN. En este paso, la información de la copia de archivo del ADN se imprime en el ARNm. La estructura del ARN es un poco diferente, contiene ribosa en lugar de desoxirribosa, y las cuatro bases que se unen a él son citosina (C), guanina (G), adenina (A) y uracilo (U). Durante la transcripción, el ADN se despliega y el ARNm se crea emparejando bases de ARNm con las bases del ADN. En este proceso, C en el ADN se traduce en G, G a C, A a U y T a A. Después de transcribir el ARNm se transporta al ribosoma.
Paso 2: El segundo paso, la traducción de proteínas ocurre en el ribosoma. Durante la traducción, la secuencia de codones (triplete de bases) del ARNm se traduce, con la ayuda del ARNt, en una secuencia de aminoácidos
Figura\(\PageIndex{5}\): La expresión génica parece ser un proceso sencillo, el mecanismo que controla la expresión génica que causa la mayoría de las diferencias fenotípicas en los organismos.
3.6 Mutación
 Las mutaciones son cambios en la secuencia genética (secuencia de ADN o ARN), y son una causa principal de diversidad entre los organismos. Aunque algunas de las mutaciones son beneficiosas, ofreciendo resistencia a la enfermedad o mejora de la estructura y/o función, algunas otras mutaciones específicas pueden llevar a la enfermedad y/o la muerte de la célula u organismo.
Las mutaciones son cambios en la secuencia genética (secuencia de ADN o ARN), y son una causa principal de diversidad entre los organismos. Aunque algunas de las mutaciones son beneficiosas, ofreciendo resistencia a la enfermedad o mejora de la estructura y/o función, algunas otras mutaciones específicas pueden llevar a la enfermedad y/o la muerte de la célula u organismo.
Las mutaciones pueden ocurrir debido a asaltos del ambiente o pueden ocurrir mutaciones espontáneas durante la replicación del ADN. Se estima que las mutaciones ocurren a una tasa aproximada de 1000 a 1,000,000 por célula por día en el genoma humano, y se cree que cada nueva célula contiene aproximadamente 120 nuevas mutaciones.
TIPOS DE MUTACIÓN
Mutaciones puntuales cuando solo un par de bases se cambia a otro par de bases. Se pueden clasificar de la siguiente manera:
- Transición: Cuando un nucleótido de purina se cambia a una purina diferente (A ↔ G) o un nucleótido de pirimidina se cambia a un nucleótido de pirimidina diferente [C ↔ T (U)].
- Transversión: Cuando se invierte la orientación de un solo nucleótido de purina y pirimidina [A/G ↔ C/T (U)].
- Silencioso: Cuando se codifica el mismo AA.
- Missense: Cuando se codifica un AA diferente.
- Neutro: Cuando ocurre un cambio de AA pero no afecta la estructura o función de la proteína.
- Tonterías: Cuando resulta un codón de parada, se termina la traducción y se acorta la proteína resultante.
Mutaciones de inserción y deleción, que juntas se conocen como indels. Los indels pueden tener una amplia variedad de longitudes. En el extremo corto del espectro, los indeles de uno o dos pares de bases dentro de las secuencias codificantes tienen el mayor efecto, ya que inevitablemente causarán un desplazamiento de marco, es decir, cambiarán la lectura completa de la secuencia de ARNm. A nivel intermedio, los indels pueden afectar partes de un gen o grupos enteros de genes. En el nivel más grande, los cromosomas enteros o incluso copias completas del genoma pueden verse afectados por inserciones o deleciones. En este alto nivel, también es posible invertir o translocar secciones enteras de un cromosoma, y los cromosomas pueden incluso fusionarse o romperse.
Si se pierde una gran cantidad de genes como resultado de uno de estos procesos, entonces las consecuencias suelen ser muy dañinas.
3.7 Reparación de ADN
El cuerpo humano cuenta con mecanismos para detectar y reparar los diversos tipos de daños que pueden ocurrir al ADN, sin importar si este daño es causado por el medio ambiente o por errores en la replicación.
Debido a que el ADN es una molécula que juega un papel activo y crítico en la división celular, durante el ciclo celular, los mecanismos de puntos de control aseguran que el ADN esté intacto antes de permitir que se produzca la replicación del ADN y la división celular. Las fallas en estos puntos de control pueden llevar a una acumulación de daños, lo que a su vez conduce a mutaciones.
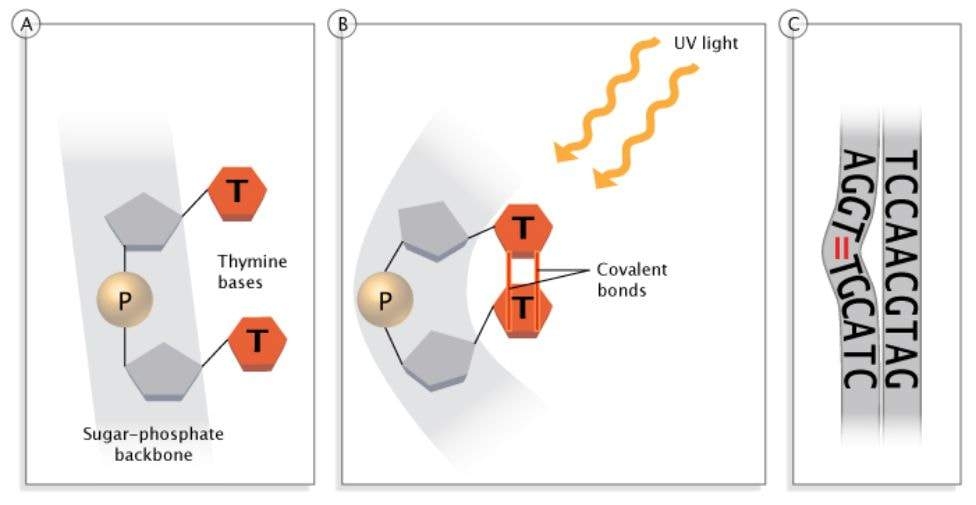
La radiación UV provoca lesiones en el ADN que pueden distorsionar la estructura del ADN, introduciendo curvas o torceduras e impidiendo así la transcripción y replicación. Estas lesiones pueden repararse a través de un proceso conocido como reparación de escisión de nucleótidos (NER), un mecanismo donde una enzima cataliza la eliminación de nucleótidos dañados, y el reemplazo de la secuencia correcta, guiada por la cadena de ADN complementaria intacta. Los defectos en este mecanismo están relacionados con enfermedades humanas como el cáncer de piel.
Otro mecanismo de reparación que maneja el daño espontáneo del ADN causado por la oxidación o hidroxilación generado por el metabolismo es la reparación por escisión de bases (BER). En este mecanismo, las enzimas conocidas como ADN glicosilasas eliminan las bases dañadas al cortarlas literalmente de la cadena de ADN a través de la escisión de los enlaces covalentes entre las bases y la cadena principal de azúcar-fosfato. El hueco resultante se llena luego con una polimerasa reparadora especializada y se sella con ligasa.
El daño al ADN también puede ocurrir en forma de roturas bicatenarias, que son causadas por la radiación ionizante, incluidos los rayos gamma y los rayos X. Las roturas bicatenarias pueden repararse a través de uno de dos mecanismos: unión de extremos no homólogos (NHEJ), donde una enzima llamada ADN ligasa IV utiliza piezas sobresalientes de ADN adyacentes a la rotura para unirse y rellenar los extremos; o reparación de recombinación homóloga (HRR) donde el cromosoma homólogo en sí se utiliza como plantilla para la reparación.
Tema 3: Puntos Clave
En esta sección, exploramos los siguientes puntos principales:
- El proceso de replicación del ADN está controlado por varias proteínas que actúan juntas para asegurar el correcto emparejamiento de bases para la creación de la nueva cadena de ADN.
- La transcripción es el primer paso de la expresión génica, en el que un segmento particular de ADN es copiado en ARN (ARNm) por la enzima ARN polimerasa.
- En la traducción, el ARN mensajero (ARNm), producido por transcripción a partir de ADN, es decodificado por un ribosoma y ARNt para producir una cadena de aminoácidos o proteína específica.
- Las mutaciones son cambios en la secuencia genética (secuencia de ADN o ARN), que pueden ser beneficiosos o pueden resultar en daños, si no se reparan.
1. El corte y empalme de ARN es el proceso que implica la eliminación de intrones presentes en el pre-ARNm y el corte y empalme de los exones restantes. ¿Verdadero o Falso?
Cierto
Falso
- Contestar
-
true
2. ¿Cuál de las siguientes no pertenece al proceso de replicación del ADN?
Primase
ADN Polimerasa
ARN Polimerasa
- Contestar
-
ARN Polimerasa
3. Durante la traducción de proteínas, la secuencia de codones (tripletes de bases) del ARNm es importante para:
Mantener la estructura del ARNm
Tradujo la secuencia correcta de aminoácidos.
Replicación de ADN
- Contestar
-
Tradujo la secuencia correcta de aminoácidos.


