2.4: Mecanismos Epigenéticos
- Page ID
- 75471
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\( \newcommand{\dsum}{\displaystyle\sum\limits} \)
\( \newcommand{\dint}{\displaystyle\int\limits} \)
\( \newcommand{\dlim}{\displaystyle\lim\limits} \)
\( \newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\)
( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\id}{\mathrm{id}}\)
\( \newcommand{\Span}{\mathrm{span}}\)
\( \newcommand{\kernel}{\mathrm{null}\,}\)
\( \newcommand{\range}{\mathrm{range}\,}\)
\( \newcommand{\RealPart}{\mathrm{Re}}\)
\( \newcommand{\ImaginaryPart}{\mathrm{Im}}\)
\( \newcommand{\Argument}{\mathrm{Arg}}\)
\( \newcommand{\norm}[1]{\| #1 \|}\)
\( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\)
\( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\AA}{\unicode[.8,0]{x212B}}\)
\( \newcommand{\vectorA}[1]{\vec{#1}} % arrow\)
\( \newcommand{\vectorAt}[1]{\vec{\text{#1}}} % arrow\)
\( \newcommand{\vectorB}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\( \newcommand{\vectorC}[1]{\textbf{#1}} \)
\( \newcommand{\vectorD}[1]{\overrightarrow{#1}} \)
\( \newcommand{\vectorDt}[1]{\overrightarrow{\text{#1}}} \)
\( \newcommand{\vectE}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash{\mathbf {#1}}}} \)
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \)
\(\newcommand{\longvect}{\overrightarrow}\)
\( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)
\(\newcommand{\avec}{\mathbf a}\) \(\newcommand{\bvec}{\mathbf b}\) \(\newcommand{\cvec}{\mathbf c}\) \(\newcommand{\dvec}{\mathbf d}\) \(\newcommand{\dtil}{\widetilde{\mathbf d}}\) \(\newcommand{\evec}{\mathbf e}\) \(\newcommand{\fvec}{\mathbf f}\) \(\newcommand{\nvec}{\mathbf n}\) \(\newcommand{\pvec}{\mathbf p}\) \(\newcommand{\qvec}{\mathbf q}\) \(\newcommand{\svec}{\mathbf s}\) \(\newcommand{\tvec}{\mathbf t}\) \(\newcommand{\uvec}{\mathbf u}\) \(\newcommand{\vvec}{\mathbf v}\) \(\newcommand{\wvec}{\mathbf w}\) \(\newcommand{\xvec}{\mathbf x}\) \(\newcommand{\yvec}{\mathbf y}\) \(\newcommand{\zvec}{\mathbf z}\) \(\newcommand{\rvec}{\mathbf r}\) \(\newcommand{\mvec}{\mathbf m}\) \(\newcommand{\zerovec}{\mathbf 0}\) \(\newcommand{\onevec}{\mathbf 1}\) \(\newcommand{\real}{\mathbb R}\) \(\newcommand{\twovec}[2]{\left[\begin{array}{r}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\ctwovec}[2]{\left[\begin{array}{c}#1 \\ #2 \end{array}\right]}\) \(\newcommand{\threevec}[3]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\cthreevec}[3]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \end{array}\right]}\) \(\newcommand{\fourvec}[4]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\cfourvec}[4]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \end{array}\right]}\) \(\newcommand{\fivevec}[5]{\left[\begin{array}{r}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\cfivevec}[5]{\left[\begin{array}{c}#1 \\ #2 \\ #3 \\ #4 \\ #5 \\ \end{array}\right]}\) \(\newcommand{\mattwo}[4]{\left[\begin{array}{rr}#1 \amp #2 \\ #3 \amp #4 \\ \end{array}\right]}\) \(\newcommand{\laspan}[1]{\text{Span}\{#1\}}\) \(\newcommand{\bcal}{\cal B}\) \(\newcommand{\ccal}{\cal C}\) \(\newcommand{\scal}{\cal S}\) \(\newcommand{\wcal}{\cal W}\) \(\newcommand{\ecal}{\cal E}\) \(\newcommand{\coords}[2]{\left\{#1\right\}_{#2}}\) \(\newcommand{\gray}[1]{\color{gray}{#1}}\) \(\newcommand{\lgray}[1]{\color{lightgray}{#1}}\) \(\newcommand{\rank}{\operatorname{rank}}\) \(\newcommand{\row}{\text{Row}}\) \(\newcommand{\col}{\text{Col}}\) \(\renewcommand{\row}{\text{Row}}\) \(\newcommand{\nul}{\text{Nul}}\) \(\newcommand{\var}{\text{Var}}\) \(\newcommand{\corr}{\text{corr}}\) \(\newcommand{\len}[1]{\left|#1\right|}\) \(\newcommand{\bbar}{\overline{\bvec}}\) \(\newcommand{\bhat}{\widehat{\bvec}}\) \(\newcommand{\bperp}{\bvec^\perp}\) \(\newcommand{\xhat}{\widehat{\xvec}}\) \(\newcommand{\vhat}{\widehat{\vvec}}\) \(\newcommand{\uhat}{\widehat{\uvec}}\) \(\newcommand{\what}{\widehat{\wvec}}\) \(\newcommand{\Sighat}{\widehat{\Sigma}}\) \(\newcommand{\lt}{<}\) \(\newcommand{\gt}{>}\) \(\newcommand{\amp}{&}\) \(\definecolor{fillinmathshade}{gray}{0.9}\)- 1: Identificar los principales mecanismos epigenéticos relacionados con el control de la expresión génica.
4.1
Introducción
 La epigenética se define como cambios potencialmente heredables y reversibles en la expresión génica mediados por la metilación del ADN, modificaciones de proteínas histonas o por ARN no codificantes que no se deben a ninguna alteración en la secuencia del ADN. Estos procesos afectan individual o conjuntamente la estabilidad del transcrito, el plegado del ADN, el posicionamiento de nucleosomas, la compactación de la cromatina y, finalmente, la organización nuclear. Determinan si un gen está silenciado o activado y cuándo y dónde ocurre esto.
La epigenética se define como cambios potencialmente heredables y reversibles en la expresión génica mediados por la metilación del ADN, modificaciones de proteínas histonas o por ARN no codificantes que no se deben a ninguna alteración en la secuencia del ADN. Estos procesos afectan individual o conjuntamente la estabilidad del transcrito, el plegado del ADN, el posicionamiento de nucleosomas, la compactación de la cromatina y, finalmente, la organización nuclear. Determinan si un gen está silenciado o activado y cuándo y dónde ocurre esto.
El cambio epigenético es una ocurrencia regular y natural, esencial para el desarrollo celular normal, pero también puede estar influenciado por varios factores como la edad, el medio ambiental/estilo de vida y el estado de enfermedad.
4.2
Metilación del ADN
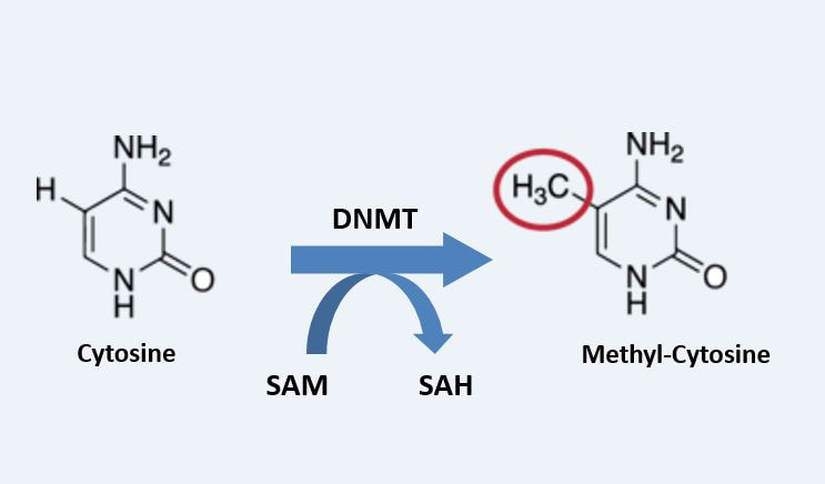 La metilación del ADN es una modificación covalente del ADN, en la que se transfiere un grupo metilo de la S-adenosilmetionina (SAM), que se convierte en S-adenosililhomocisteína (SAH), a la posición 5 de la citosina por una familia de enzimas conocidas como ADN metiltransferasas (DNMT).
La metilación del ADN es una modificación covalente del ADN, en la que se transfiere un grupo metilo de la S-adenosilmetionina (SAM), que se convierte en S-adenosililhomocisteína (SAH), a la posición 5 de la citosina por una familia de enzimas conocidas como ADN metiltransferasas (DNMT).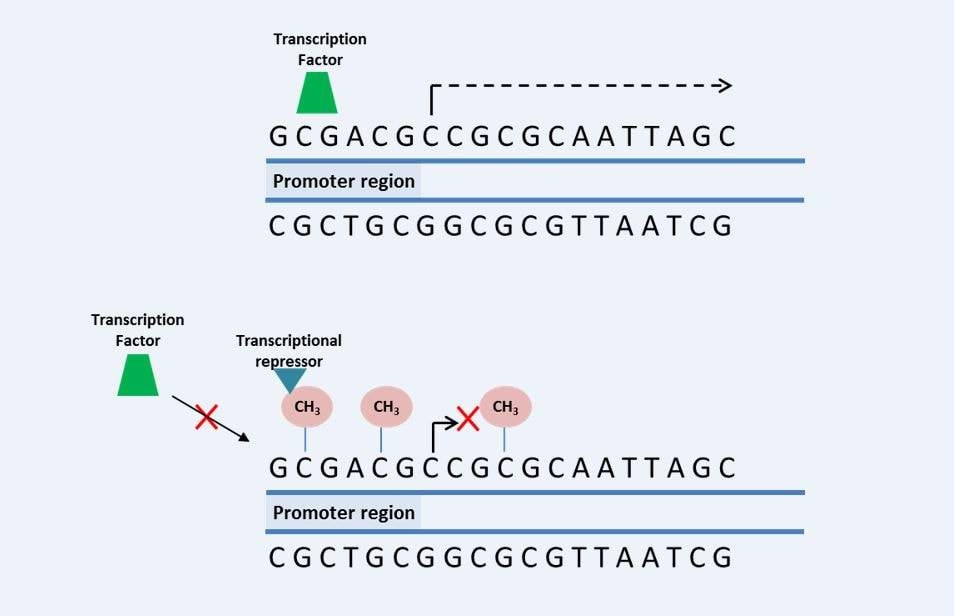 La metilación del ADN ocurre predominantemente en citosinas localizadas cerca de guaninas, conocidas como dinucleótidos CpG (CpG) o isla CpG. Estas islas CpG se encuentran en la región promotora de genes. El grupo metilo inhibe la unión de los factores de transcripción a su sitio de reconocimiento, dando como resultado la inhibición de la transcripción génica. Además, el grupo metilo puede atraer otras proteínas conocidas como represores transcripcionales que contribuyen a la inhibición de la transcripción génica.
La metilación del ADN ocurre predominantemente en citosinas localizadas cerca de guaninas, conocidas como dinucleótidos CpG (CpG) o isla CpG. Estas islas CpG se encuentran en la región promotora de genes. El grupo metilo inhibe la unión de los factores de transcripción a su sitio de reconocimiento, dando como resultado la inhibición de la transcripción génica. Además, el grupo metilo puede atraer otras proteínas conocidas como represores transcripcionales que contribuyen a la inhibición de la transcripción génica.4.3
Modificaciones postraduccionales de las histonas
El nucleosoma está compuesto por cinco proteínas histonas (H1, H2A, B, H3 y H4). El extremo N de estas proteínas histonas está sujeto a modificaciones covalentes tales como metilación, fosforilación, acetilación, ubiquitinación o sumoilación por un grupo de enzimas modificadoras de histonas. Las alteraciones en estas proteínas contribuyen a la accesibilidad y compacidad de la cromatina, y resultan en la activación o supresión de genes particulares.
Ejemplos de tipos y roles de modificaciones de histonas
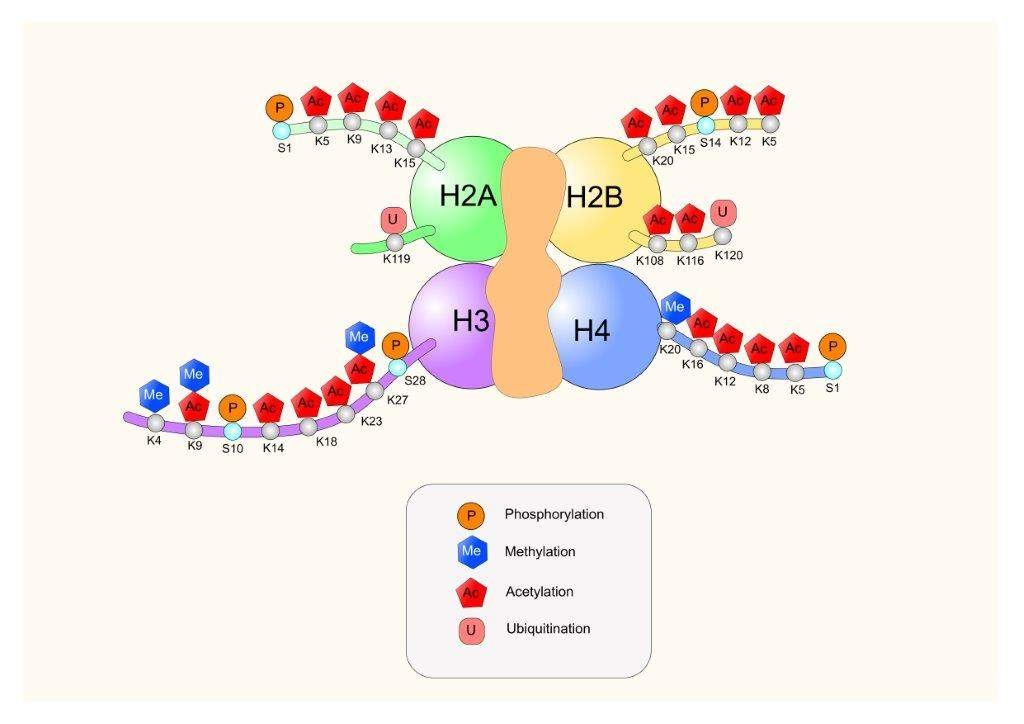
Activación de la transcripción génica:
Acelilación en la lisina 9 (K9) de la histona 3 (H3) - H3K9ac
Acetilación en la lisina 27 (K27) de la histona 3 (H3) - H3K27ac
Trimetilación en la lisina 4 (K4) de la histona 3 (H3) - H3K4me3
Represión de la transcripción génica:
Trimetilación en la lisina 4 (K3) de la histona 3 (H3) - H3K9me3
Trimetilación en la lisina 27 (K27) de la histona 3 (H3) - H3K27me3
Trimetilación en la lisina 20 (K20) de la histona 3 (H3) - H3K20me3
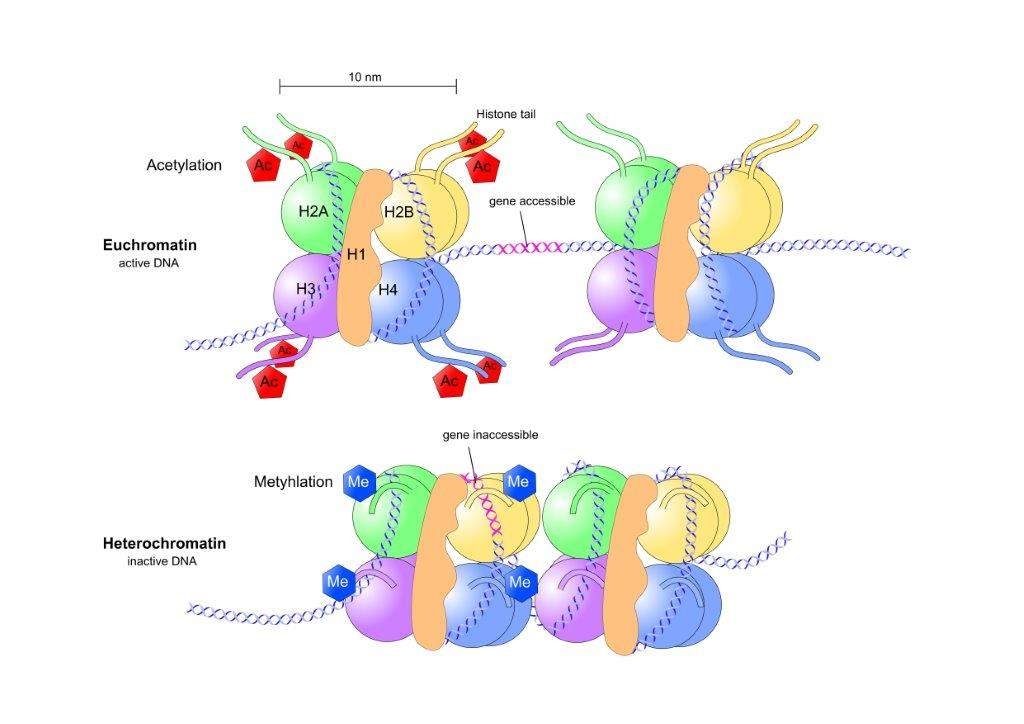 La metilación del ADN y las modificaciones postraduccionales de histonas juegan un papel importante en el establecimiento de la estructura de la cromatina y en consecuencia en la modulación de la expresión génica. Estos mecanismos epigenéticos pueden ser independientes o pueden ocurrir juntos para controlar la expresión génica. También pueden influir entre sí, por ejemplo, la metilación de histonas puede ayudar a dirigir los patrones de metilación del ADN, y la metilación del ADN podría servir como molde para algunas modificaciones de histonas después de la replicación del ADN.
La metilación del ADN y las modificaciones postraduccionales de histonas juegan un papel importante en el establecimiento de la estructura de la cromatina y en consecuencia en la modulación de la expresión génica. Estos mecanismos epigenéticos pueden ser independientes o pueden ocurrir juntos para controlar la expresión génica. También pueden influir entre sí, por ejemplo, la metilación de histonas puede ayudar a dirigir los patrones de metilación del ADN, y la metilación del ADN podría servir como molde para algunas modificaciones de histonas después de la replicación del ADN.4.4
MicroARN
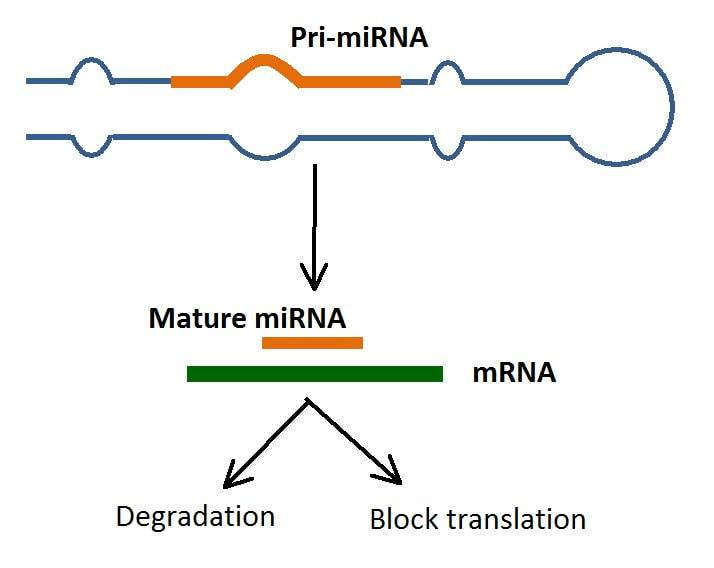
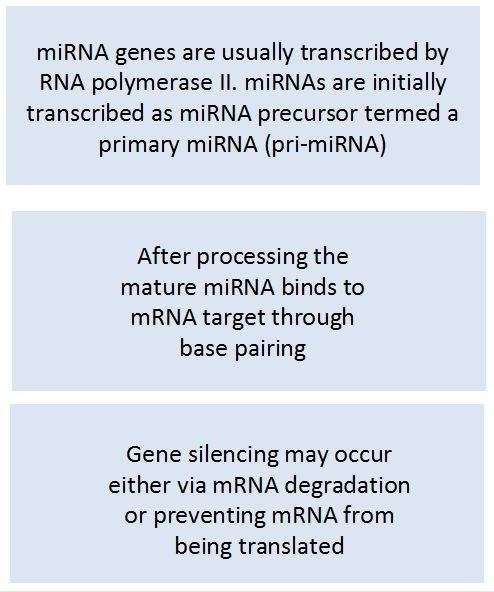
Los miARN tienen un papel importante en la regulación génica y pueden influir en las funciones biológicas, incluyendo la diferenciación y proliferación celular durante el desarrollo normal y las respuestas patológicas. Son pequeñas moléculas de ARN no codificantes (que contienen aproximadamente 22 nucleótidos), derivadas de regiones de transcritos de ARN que se pliegan sobre sí mismas para formar horquillas cortas. Regulan la expresión génica a nivel postranscripcional. Varios miARN pueden unirse a regiones específicas del ARN mensajero (ARNm) y bloquear su traducción a proteínas. Se cree que la alteración de la expresión de los miARN contribuye a la progresión de la tumorigénesis y otras enfermedades.
Tema 4: Puntos clave
En esta sección, exploramos los siguientes puntos principales:
- 1: Los mecanismos epigenéticos pueden influir en la expresión génica sin alteración en la secuencia de ADN.
- 2: Los principales mecanismos epigenéticos son la metilación del ADN, la modificación postraduccional de las histonas y la alteración en la expresión de microARN.
- 3: El cambio epigenético es una ocurrencia regular y natural, pero también puede estar influenciado por varios factores como la edad, el medio ambiental/estilo de vida y el estado de enfermedad.
- 4: A diferencia de las alteraciones genéticas, las alteraciones epigenéticas se consideran reversibles.
1. Se consideran mecanismos epigenéticos:
Reversible
Irreversible
- Contestar
-
Reversible
2. La metilación del ADN y las modificaciones postraduccionales de histonas juegan un papel importante en el establecimiento de la estructura de la cromatina y en consecuencia en la modulación de la expresión génica. ¿Verdadero o Falso?
Falso
Cierto
- Contestar
-
Cierto
3. La metilación del ADN puede inhibir la unión de factores de transcripción a su sitio de reconocimiento, en regiones promotoras dando como resultado:
Inhibición de la transcripción génica
Activación de la transcripción génica
- Contestar
-
Inhibición de la transcripción génica


