7.4: Replicación eucariota
- Page ID
- 53014
Dada la naturaleza crucial de la replicación cromosómica para que exista la vida, no es sorprendente encontrar que la replicación del ADN eucariota es muy similar al proceso procariota. En esta sección se destacarán algunas de las diferencias, que generalmente son elaboraciones sobre la versión procariota.
A diferencia de los procariotas, los cromosomas eucariotas a menudo tienen múltiples orígenes de replicación. Considerando el tamaño de los cromosomas eucariotas, esto es necesario para terminar la replicación completa de manera oportuna. Cada uno de estos orígenes define un replicón, o el tramo del ADN que se replica desde un origen particular. Los replicones no se replican exactamente al mismo tiempo (aunque todos dentro de la misma fase del ciclo celular, ver capítulo 15), por lo que es importante asegurarse de que los replicones se usen solo una vez durante un ciclo celular.
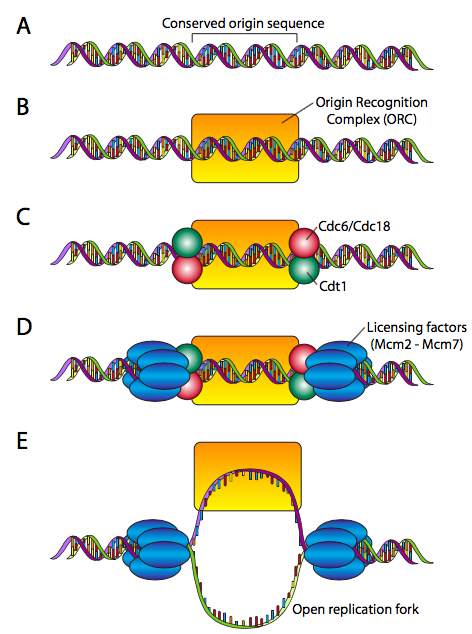
Esto requiere un mecanismo de “licenciamiento”. Durante la fase del ciclo celular antes de iniciar la replicación del ADN, se ensambla un complejo prerreplicativo en cada origen (Figura\(\PageIndex{13}\)). Los orígenes son muy variables en composición y longitud, que van desde ~100 hasta más de 10000 pares de bases. Las proteínas pre-RC, por otro lado, están muy conservadas. El pre-RC comienza haciendo el ORC (complejo de reconocimiento de origen, no una criatura que lucha contra Frodo y Aragorn), que se compone de seis subunidades (Orc1-Orc6). Aunque no existe una homología de secuencia significativa, el ORC se aproxima a la función de la proteína DNAA en E. coli. Para completar el pre-RC, el ORC recluta un par de proteínas, Cdc6 y Cdt1 a cada lado, y se unen al complejo MCM (un hexámero de Mcm2-Mcm6 que tiene una actividad helicasa inactiva), lo que lleva a la pre-RC con licencia completa. El origen ya está listo para su activación.
La activación del pre-RC en un origen de replicación requiere primero Mcm10, lo que facilita las proteínas quinasas que fosforilan el complejo MCM, activando la actividad helicasa y haciendo que la horquilla de replicación esté lista para aceptar la máquina de replicación.
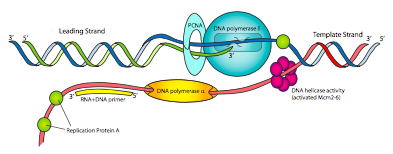
Los componentes de la máquina de replicación eucariota pueden tener diferentes nombres de la procariota, pero las funciones deben ser muy familiares (Figura\(\PageIndex{14}\)). Hay una primasa para hacer cebadores de ARN, y estrechamente asociada con la primasa está la ADN polimerasa α. Pol α no tiene ni una exonucleasa 5'-3' ni una exonucleasa correctora 3'-5', pero puede sintetizar ADN. Sin embargo, esta no es la ADN polimerasa primaria. El complejo primase/pol a es esencialmente una primasa elegante que comienza con un cebador corto de ARN <10nt y luego agrega otro fragmento corto de ADN de ~15 nt, haciendo un cebador híbrido.
El factor de replicación C (RFC), que actúa como el complejo de pinza cargadora procariota, luego reemplaza pol α con PCNA, la versión eucariota de la pinza β. Esto luego recluta ADN polimerasa δ, que es la ADN polimerasa replicativa primaria, equivalente a Pol III procariota en función, y necesaria para la síntesis de cadenas tanto líderes como rezagadas. Finalmente, en lugar de SSB, las células eucariotas utilizan la proteína de replicación A (RPA) para organizar y controlar el ADN monocatenario a medida que se genera durante el proceso replicativo.
Es posible que hayas notado que ninguna de las ADN polimerasas eucariotas discutidas hasta ahora tiene una actividad exonucleasa de 5' a 3' como la utilizada por la ADN polimerasa I procariota para eliminar los cebadores. En lugar de eso, RNasaH1 y FEN-1 eliminan los cebadores de ARN (todos menos un ribonucleótido, y el último ribonucleótido, respectivamente). Curiosamente, FEN-1 también extirpa trozos de ADN de hebra rezagada dentro de aproximadamente 15 pares de bases del ARN si contienen errores. Esto parece ayudar a aliviar el problema de la menor fidelidad de replicación por parte de pol α, que no tiene capacidad de corrección. Después de que RNasaH1 y FEN-1 hayan eliminado los cebadores y los errores cercanos al cebador, pol δ rellena los nucleótidos faltantes y una enzima ligasa se une a los fragmentos. Pol ε tiene una actividad exonucleasa 3'-5' que corta el ADN monocatenario en pequeños fragmentos de oligonucleótidos y también se asocia con la máquina de replicación. No se entiende claramente la función de Pol ε.


