2.8: Apéndice
- Page ID
- 54680
Homología
Uno de los objetivos clave del alineamiento de secuencias es identificar secuencias homólogas (por ejemplo, genes) en un genoma. Dos secuencias homólogas están relacionadas evolutivamente, específicamente por descendencia de un ancestro común. Los dos tipos primarios de homólogos son ortólogos y parálogos (consultar la Figura 2.14 11). Existen otras formas de homología (por ejemplo, xenólogos), pero están fuera del alcance de estas notas.
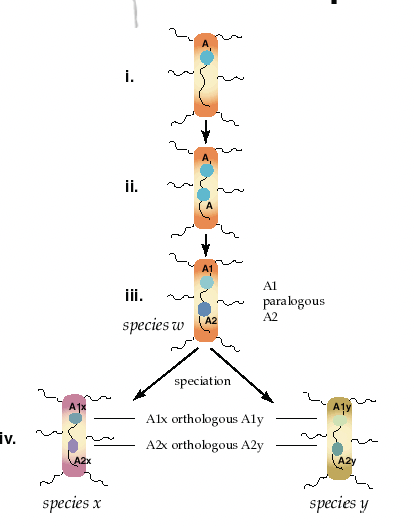
Los ortólogos surgen de eventos de especiación, dando lugar a dos organismos con una copia del mismo gen. Por ejemplo, cuando una sola especie A se especia en dos especies B y C, hay genes en las especies B y C que descienden de un gen común en la especie A, y estos genes en B y C son ortólogos (los genes continúan evolucionando independientemente entre sí, pero aún realizan la misma función relativa).
Los parálogos surgen de eventos de duplicación dentro de una especie. Por ejemplo, cuando se produce una duplicación génica en algunas especies A, la especie tiene un gen B original y una copia génica B′, y los genes B y B′ son paralogus. Generalmente, las secuencias ortólogas entre dos especies estarán más estrechamente relacionadas entre sí que las secuencias parálogas. Esto ocurre porque los ortólogos suelen conservar (aunque no siempre) la función a lo largo del tiempo, mientras que los parálogos suelen cambiar con el tiempo, por ejemplo especializando la (sub) función de un gen o evolucionando una nueva función. Como resultado, determinar secuencias ortólogas es generalmente más importante que identificar secuencias parálogas cuando se mide la relación evolutiva.
Selección Natural
El tema de la selección natural es un tema demasiado grande para resumirlo efectivamente en pocos párrafos cortos; en cambio, este apéndice introduce tres tipos amplios de selección natural: selección positiva, selección negativa y selección neutra.
- La selección positiva ocurre cuando un rasgo es evolutivamente ventajoso y aumenta la aptitud de un individuo, de modo que un individuo con el rasgo es más probable que tenga descendencia (robusta). A menudo se asocia con el desarrollo de nuevos rasgos.
- La selección negativa ocurre cuando un rasgo es evolutivamente desventajoso y disminuye la condición física de un individuo. La selección negativa actúa para reducir la prevalencia de alelos genéticos que reducen la aptitud de una especie. La selección negativa también se conoce como selección purificadora debido a su tendencia a 'purificar' los alelos genéticos hasta que solo los alelos más exitosos existen en la población.
- La selección neutra describe la evolución que ocurre aleatoriamente, como resultado de que los alelos no afectan a la aptitud de un individuo. En ausencia de presiones selectivas, no se produce ninguna selección positiva o negativa, y el resultado es la selección neutra.
Programación dinámica v. Algoritmos codiciosos
La programación dinámica y los algoritmos codiciosos son algo similares, y le corresponde a uno conocer las distinciones entre los dos. Los problemas que pueden resolverse mediante programación dinámica suelen ser problemas de optimización que presentan dos rasgos: 1. subestructura óptima y 2. subproblemas superpuestos.
Los problemas solucionables por algoritmos codiciosos requieren tanto estos rasgos como (3) la propiedad de elección codiciosa. Cuando se trata de un problema “en la naturaleza”, a menudo es fácil determinar si satisface (1) y (2) pero difícil determinar si debe tener la propiedad de elección codiciosa. No siempre está claro si las elecciones óptimas a nivel local producirán una solución óptima a nivel mundial.
Para los biólogos computacionales, hay dos puntos útiles a tener en cuenta con respecto a si emplear programación dinámica o programación codiciosa. Primero, si un problema puede resolverse usando un algoritmo codicioso, entonces puede resolverse usando programación dinámica, mientras que lo contrario no es cierto. En segundo lugar, las estructuras problemáticas que permiten algoritmos codiciosos normalmente no aparecen en la biología computacional.
Para dilucidar este segundo punto, podría ser útil considerar las estructuras que permiten que la programación codiciosa funcione, pero tal discusión nos llevaría demasiado lejos. El estudiante interesado (preferiblemente uno con antecedentes matemáticos) debe mirar a los matroides y avaridos, que son estructuras que tienen la propiedad de elección codiciosa. Para nuestros propósitos, simplemente vamos a afirmar que los problemas biológicos suelen involucrar a entidades que son altamente sistémicas y que hay pocas razones para sospechar suficiente estructura en la mayoría de los problemas para emplear algoritmos codiciosos.
Pseudocódigo para el algoritmo Needleman-Wunsch
El primer problema en el primer conjunto de problemas le pide que termine una implementación del algoritmo Needleman-Wunsch (NW), y el código Python de trabajo para el algoritmo se omite intencionalmente. En cambio, este apéndice resume los pasos generales del algoritmo NW (Sección 2.5) en un solo lugar.
Problema: Dadas dos secuencias S y T de longitud m y n, una matriz de sustitución vU de puntuaciones coincidentes, y una penalización por hueco G, determinan la alineación óptima de S y T y la puntuación del alineamiento.
Algoritmo:
- Crear dos m + 1 por n + 1 matrices A y B. A será la matriz de puntuación y B será la matriz de seguimiento. La entrada (i, j) de la matriz A contendrá la puntuación de la alineación óptima de las secuencias S [1,.., i] y T [1,.., j], y la entrada (i, j) de la matriz B contendrá un puntero a la entrada a partir de la cual se construyó el alineamiento óptimo.
- Inicializar la primera fila y columna de la matriz de puntuación A de tal manera que las puntuaciones tengan en cuenta las penalizaciones por brecha, e inicializar la primera fila y columna de la matriz de trazabilidad B de la manera obvia.
- Pasar por las entradas (i, j) de la matriz A en algún orden razonable, determinando el alineamiento óptimo de las secuencias S [1,... , i] y T [1,... , j] usando las entradas (i − 1, j − 1), (i − 1, j) y (i, j − 1). Establezca el puntero en la matriz B a la entrada correspondiente a partir de la cual se construyó la alineación óptima en (i, j).
- Una vez completadas todas las entradas de las matrices A y B, la puntuación de la alineación óptima se puede encontrar en la entrada (m, n) de la matriz A.
- Construir la alineación óptima siguiendo la trayectoria de los punteros comenzando en la entrada (m, n) de la matriz B y terminando en la entrada (0, 0) de la matriz B.
11 R.B. - BIOS 60579


