12.7: Tecnologías- en el laboratorio húmedo, ¿cómo podemos encontrarlas?
- Page ID
- 54508
¿Cómo encontraríamos los ARNNC? Tenemos alrededor de 20-30 ejemplos de ARNcn con evidencia de importancia, pero hay más por ahí. Los mapas de estado de la cromatina (de ENCODE, chip-seq) se pueden utilizar para encontrar unidades transcripcionales que no se superpongan a las proteínas. Podemos caminar a lo largo del mapa y buscar genes (mirar a ojo el mapa de la cromatina para encontrar los ARNcns). Casi el 90% de las veces que se encuentre una firma de este tipo, se transcribirá ARN a partir de ella. Podemos validar esto a través de Northern Blot
Al mirar un mapa de cromatina para encontrar NCRNAs, esencialmente estamos mirando a través del mapa con una ventana de un tamaño dado y viendo cuánta señal vs. ruido estamos recibiendo, en comparación con lo que podríamos esperar de una hipótesis de azar aleatorio. Como tanto las ventanas grandes como las pequeñas tienen beneficios, ambas deben usarse en cada sección del mapa. Las ventanas más grandes encapulan más información; las ventanas más pequeñas son más sensibles.
Después de encontrar regiones integénicas, encontramos regiones conservadas.
Comprobamos si las nuevas regiones están bajo presión selectiva; menos mutaciones en regiones conservadas. Si un nucleótido nunca tiene una mutación entre especies, está altamente conservado.
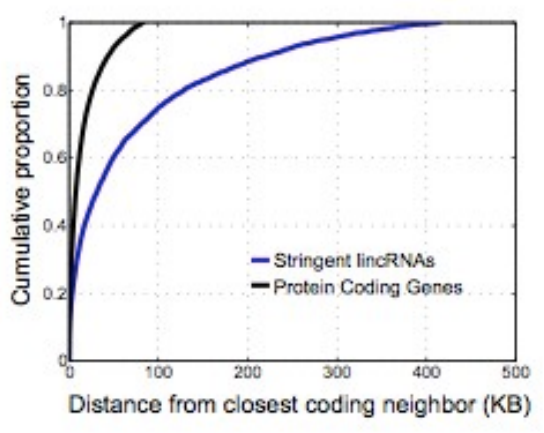
Los ARNs de LINC están más conservados que los intrones, pero menos conservados que los intrones que codifican proteínas, posiblemente debido a secuencias no conservadas en las regiones de bucle de los LINCRNA.
Encontrar cuáles son las funciones de los linCRNA: “Culpabilidad por asociación”: Podemos encontrar proteínas que se correlacionan con el linARN particular en términos de expresión; los LINCRNA probablemente se correlacionen con una vía particular. De esta manera, adquirimos un código de barras multidimensional para cada LincRNA (lo que es y con lo que no está relacionado). Podemos agrupar las firmas de lincRNA e identificar patrones comunes. Mucho tiene que ver con los genes del ciclo celular. (Este enfoque funciona 60-70% del tiempo)
Como la mayoría de los LINCRNA tienen más de 3000 bases, muchos contienen secuencias para marcos de lectura abiertos de 100 aminoácidos, simplemente por casualidad. Esto da como resultado muchos falsos negativos durante la detección.
Se ha encontrado que muchos LINCRNA tienden a vecinos de las regiones del desarrollo del genoma. También tienden a ser de baja expresión en comparación con los genes codificantes de proteínas.
© fuente desconocida. Todos los derechos reservados. Este contenido está excluido de nuestra licencia Creative Commons. Para obtener más información, consulte http://ocw.mit.edu/help/faq-fair-use/.
Ejemplo: p53
Validación independiente: se utilizan modelos animales, donde uno es un p53 de tipo silvestre, y uno es un knockout. Inducimos p53, luego preguntamos si los LINCRNA se encienden. 32 de los 39 LINCRNA encontrados asociados con p53 fueron inducidos temporalmente al encender p53.
Un ARN en particular se sentó junto a un gen codificante de proteínas en la vía p53. Intentamos averiguar si p53 se unía al promotor y lo encendimos. Para ello, clonamos el promotor de lincRNA, y preguntamos ¿p53 lo enciende? Se realizó una prueba de detección de la proteína p53, para ver si se asoció con el LincRNA del promotor. Resultó que el lincRNA está directamente relacionado con p53 - p53 lo enciende. P53 también desactiva los genes; ciertos LINCRNA actúan como represor.
A partir de este ejemplo (y otros), empezamos a ver que los ARN suelen tener un compañero proteico
El ARN puede unir innumerables proteínas diferentes, permitiendo que las células tengan mucha diversidad. De esta manera es similar a la fosforilación. Los ARN se unen a importantes complejos de cromatina y se requieren para reprogramar las células de la piel en células madre.


